Cet article décrit comment calculer et ajouter automatiquement des p-values sur des ggplot facets avec différentes échelles en utilisant les packages R ggpubr et rstatix. Pour les graphiques multi-panneaux dont les échelles de l’axe des y sont à peu près similaires sur chaque panneau, vous pouvez suivre les étapes décrites dans cet article : Comment Ajouter des P-values aux Graphiques GGPLOT avec Facet.
Ici, nous allons utiliser une donnée de démonstration pour créer des panneaux de graphiques avec des échelles y très différentes. Vous apprendrez comment calculer automatiquement les positions y des p-values lorsque l’échelle des facets est configurée sur l’option free. Des exemples sont représentés pour les box plots et les bar plots.
Sommaire:
Prérequis
Lisez l’article apparenté suivant : Comment Ajouter des P-values aux Graphiques GGPLOT avec Facet.
Assurez-vous d’avoir installé les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixcontient des fonctions R facilitant les analyses statistiques.
Commencez par charger les packages requis suivants:
library(ggpubr)
library(rstatix)Préparation des données
# Transformer `dose` en variable factorielle
df <- ToothGrowth
df$dose <- as.factor(df$dose)
# Ajouter une variable de regroupement aléatoire
df$group <- factor(rep(c("grp1", "grp2"), 30))
# Ajoutez des valeurs extrêmement élevées dans la colonne 1 aux lignes c(1, 3, 5).
df[c(1, 3, 5), 1] <- c(500, 495, 505)
head(df, 3)## len supp dose group
## 1 500.0 VC 0.5 grp1
## 2 11.5 VC 0.5 grp2
## 3 495.0 VC 0.5 grp1Tests statistiques
Faire un ggplot avec les variables supp et group, et comparer les niveaux de la variable dose sur l’axe des x. Le test post hoc de Tukey est utilisé pour les comparaisons par paires.
stat.test <- df %>%
group_by(group, supp) %>%
tukey_hsd(len ~ dose)
stat.test ## # A tibble: 12 x 11
## supp group term group1 group2 null.value estimate conf.low conf.high p.adj p.adj.signif
## * <fct> <fct> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 OJ grp1 dose 0.5 1 0 6.32 0.275 12.4 0.0403 *
## 2 OJ grp1 dose 0.5 2 0 11.3 5.26 17.3 0.000852 ***
## 3 OJ grp1 dose 1 2 0 4.98 -1.06 11.0 0.112 ns
## 4 VC grp1 dose 0.5 1 0 -286. -548. -23.5 0.0328 *
## 5 VC grp1 dose 0.5 2 0 -276. -539. -14.0 0.0389 *
## 6 VC grp1 dose 1 2 0 9.46 -253. 272. 0.995 ns
## # … with 6 more rowsFaire un ggplot avec des échelles fixes
# Créer des bar plots avec les niveaux de significativité
# Cacher les ns (non significatif)
# Ajoutez 15 % d'espace entre les étiquettes et la bordure supérieure du graphique
stat.test <- stat.test %>% add_xy_position(x = "dose", fun = "mean_se")
ggbarplot(
df, x = "dose", y = "len", fill = "#00AFBB",
add = "mean_se", facet = c("supp", "group")
) +
stat_pvalue_manual(stat.test, hide.ns = TRUE, tip.length = 0, step.increase = 0) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.15)))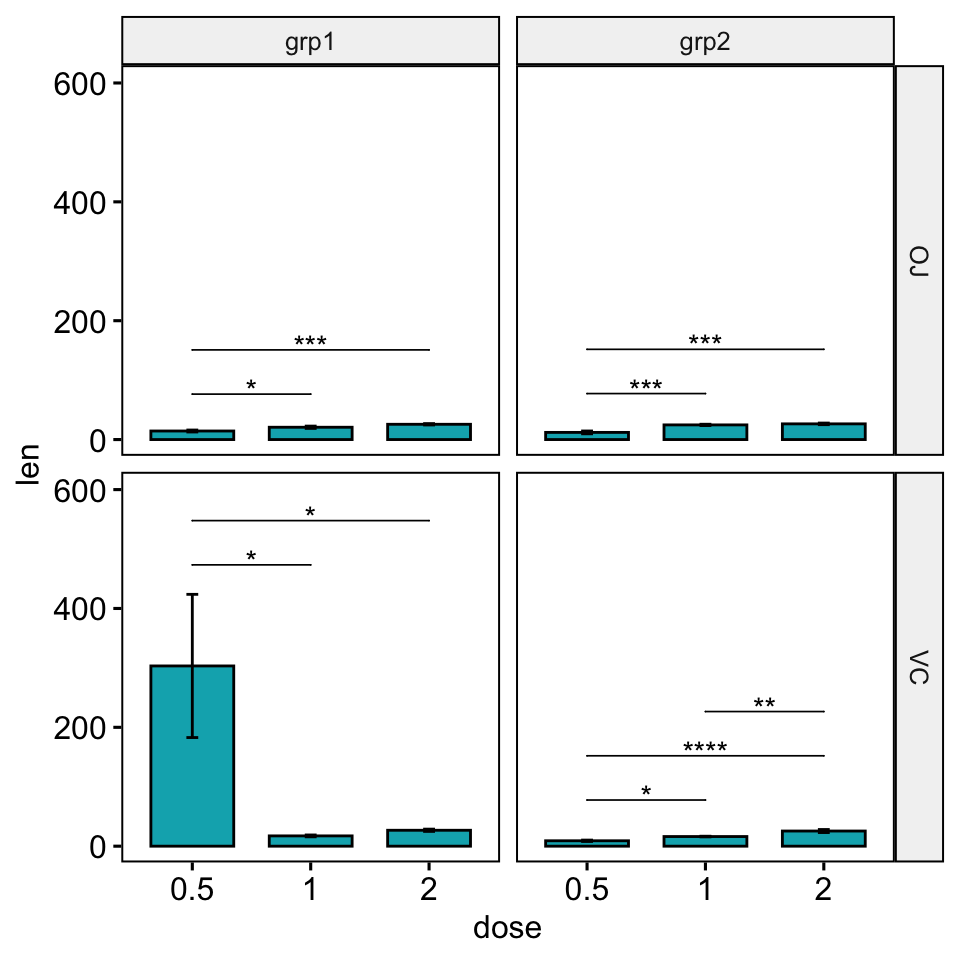
Faire un ggplot avec des échelles libres
Facet wrap
Vous devez spécifier l’option scales = "free" à la fois dans les fonctions add_xy_position() et ggbarplot().
stat.test <- stat.test %>%
add_xy_position(x = "dose", fun = "mean_se", scales = "free")
ggbarplot(
df, x = "dose", y = "len", fill = "#00AFBB",
add = "mean_se", facet.by = c("supp", "group")
) +
facet_wrap(vars(supp, group), scales = "free") +
stat_pvalue_manual(stat.test, hide.ns = TRUE, tip.length = 0) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.15)))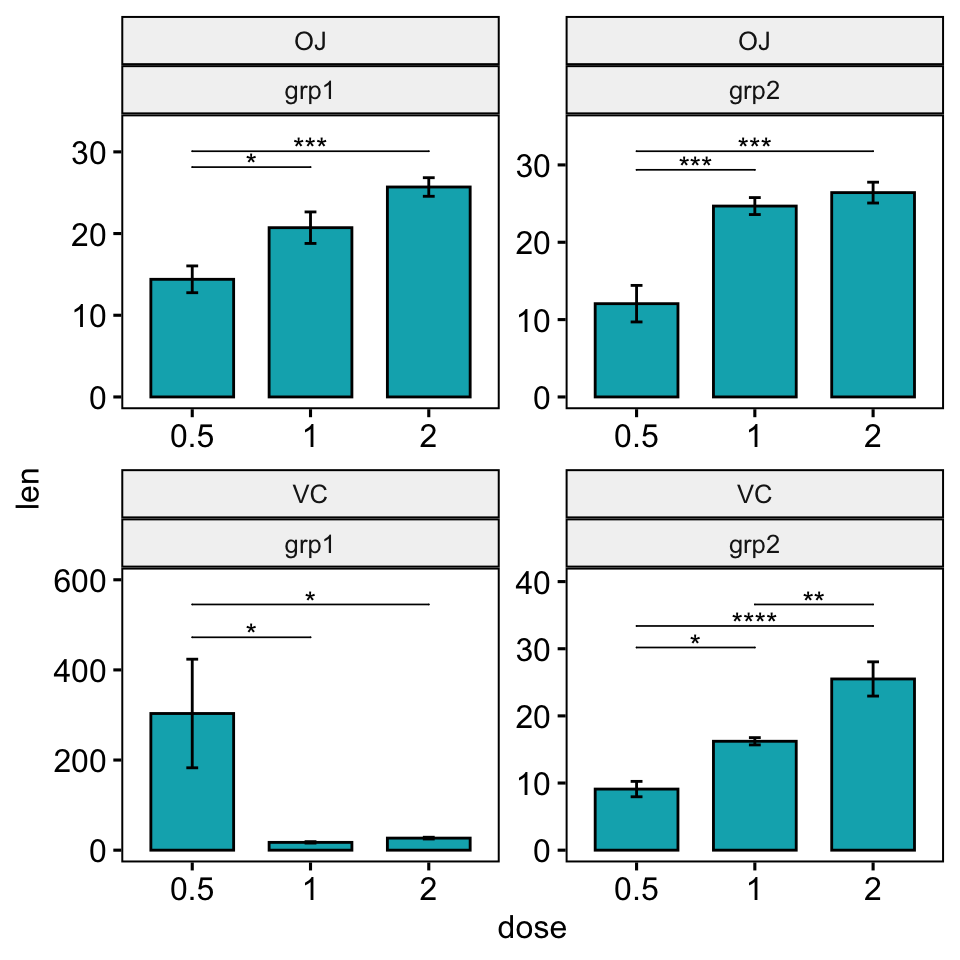
Facet grid
Lorsque l’argument scales = "free" est ajouté dans facet grid, les graphiques d’une même ligne ne peuvent pas avoir un axe des y différent. De même, il ne peut y avoir qu’un seul axe des abscisses pour chaque colonne. En faisant appel à la fonction facet_wrap(), chaque graphique est affiché indépendamment, ce qui permet de rendre “libre” ses axes x et y.
Facet grid est utile lorsque vous souhaitez comparer les graphiques d’une même catégorie, ce qui peut être fait en configurant les mêmes échelles d’axes. En revanche, facet wrap est plus utile pour les graphiques qui sont plus indépendantes les unes des autres.
Il existe deux solutions possibles pour personnaliser la position y des niveaux de significativité.
Solution 1 : Utiliser l’option step.increase
Le comportement par défaut de la fonction add_xy_position() est de calculer automatiquement une valeur globale d’augmentation de pas (step.increase) entre les crochets. Ce calcul suppose que les échelles y des panneaux du graphique sont fixes.
Dans la situation où vous voulez des échelles libres, vous pouvez:
- Paramétrez l’option “step.increase” à 0 lors de l’appel de la fonction
add_xy_position(). - Précisez uniquement l’option
step.increasein the functionstat_pvalue_manual(). Dans ce cas, l’option step.increase sera adapté à chaque panneau de graphique.
stat.test <- stat.test %>%
add_xy_position(x = "dose", fun = "mean_se", step.increase = 0)
bp <- ggbarplot(
df, x = "dose", y = "len", fill = "#00AFBB", add = "mean_se",
facet.by = c("supp", "group"), scales = "free"
)
bp +
stat_pvalue_manual(stat.test, hide.ns = TRUE, tip.length = 0, step.increase = 0.2) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.15)))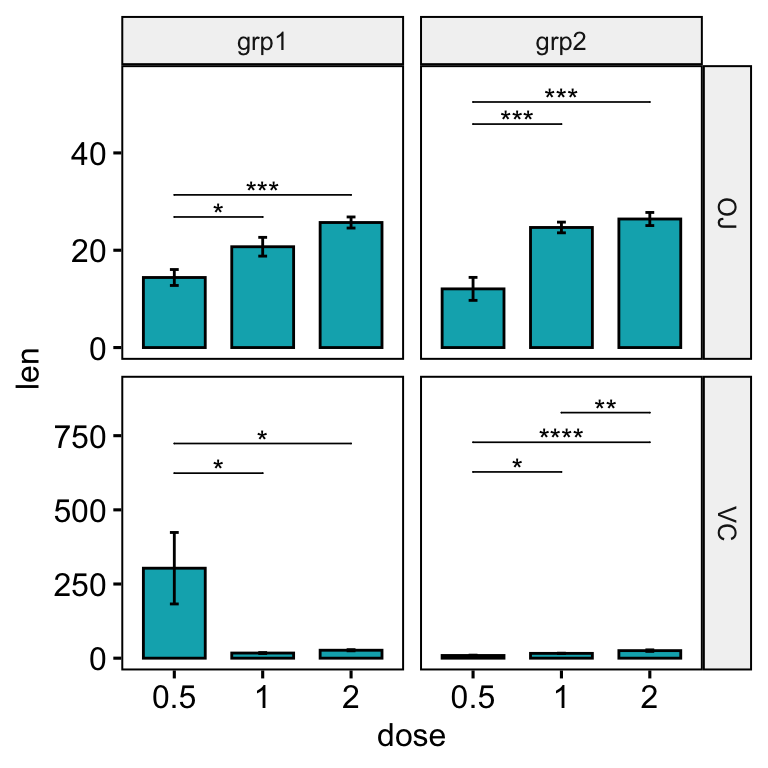
Solution 2 : Utiliser loption scales = “free”.
Dans facet grid, les échelles des panneaux de ggplot générés ne sont pas totalement libres. Par conséquent, vous aurez besoin de plus de personnalisation pour adapter l’aspect de la position y du niveau de significativité. Vous devrez jouer avec les options step.increase et brachet.nudge.y dans la fonction stat_pvalue_manual().
# Graphique par défaut
stat.test <- stat.test %>%
add_xy_position(x = "dose", fun = "mean_se", scales = "free")
bp <- ggbarplot(
df, x = "dose", y = "len", fill = "#00AFBB", add = "mean_se",
facet.by = c("supp", "group"), scales = "free"
)
bp +
stat_pvalue_manual(stat.test, hide.ns = TRUE, tip.length = 0) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.15)))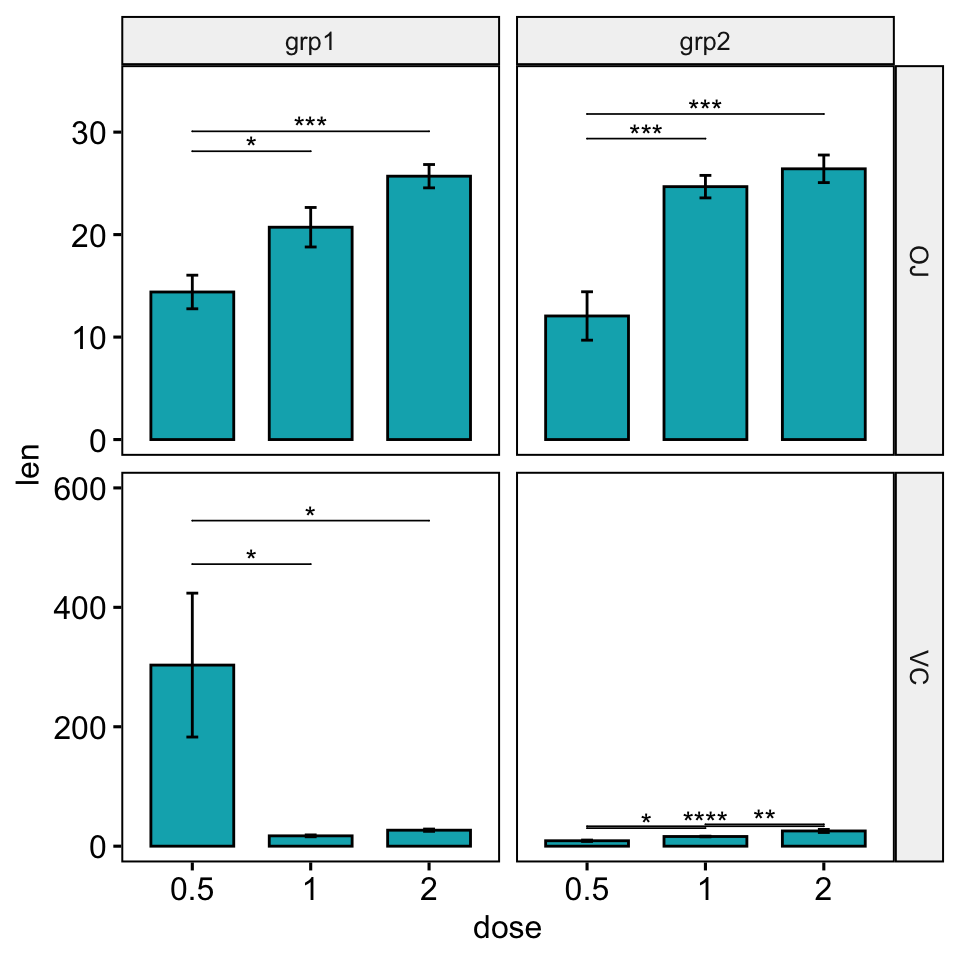
# Augmenter le pas entre les crochets
bp +
stat_pvalue_manual(
stat.test, hide.ns = TRUE, tip.length = 0,
step.increase = 0.1
) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.15)))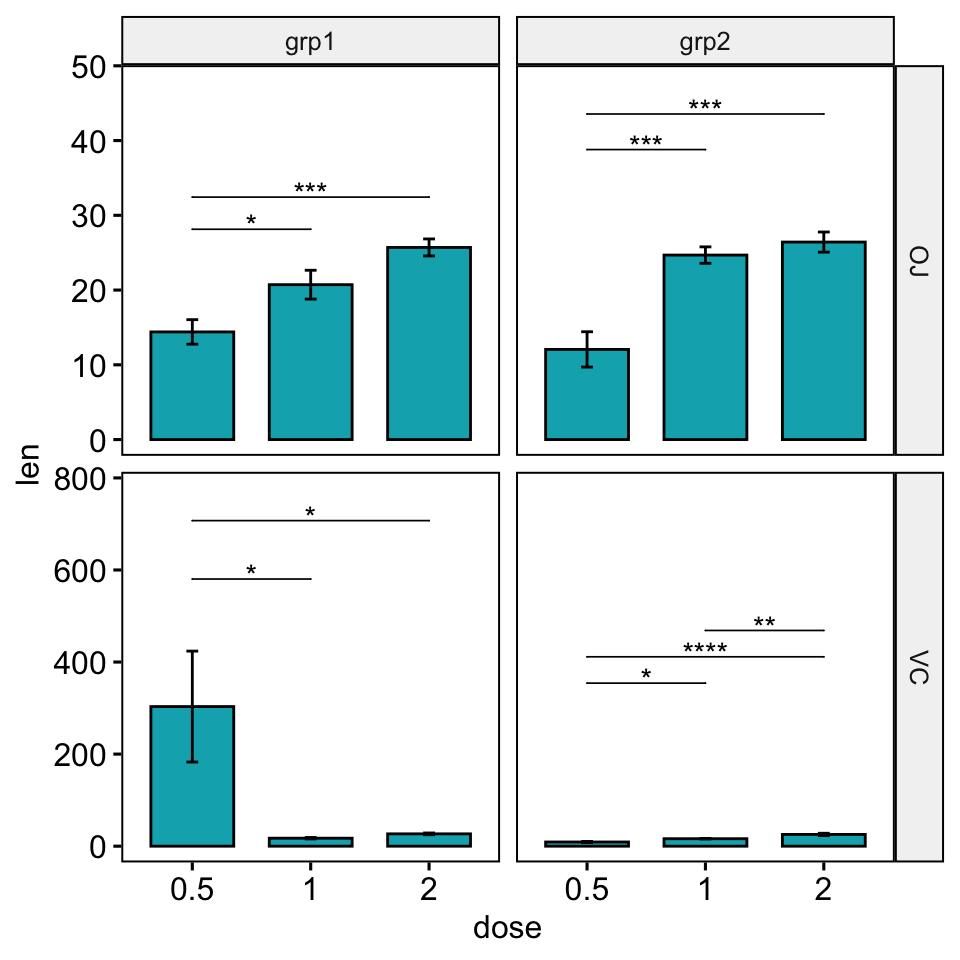
# Mettez les crochets en bas
# Précisez l'option bracket.nudge.y pour chacun des panels
# Doit être de la même longueur que le nombre de comparaisons
bracket.nudge.y <- c(
-2, -3, # Panel 1 : grp1/OJ
-100, -160, # Panel 2 : grp1/VC
-10, -11, # Panel 3 : grp2/OJ
-250, -250, -250 # Panel 4 : grp2/VC
)
bp +
stat_pvalue_manual(
stat.test, hide.ns = TRUE, tip.length = 0,
step.increase = 0.09, bracket.nudge.y = bracket.nudge.y
) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.15)))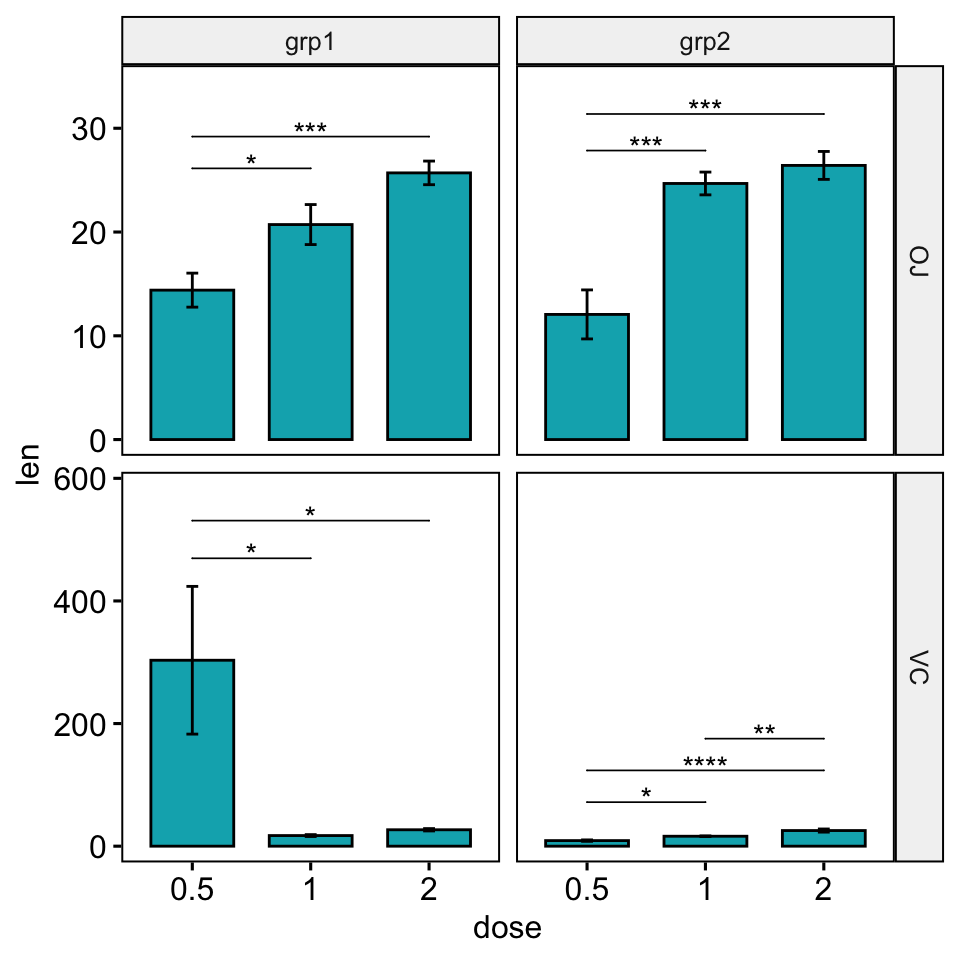
Conclusion
Cet article décrit comment ajouter des p-values sur des ggplots facets avec différentes échelles sur l’axe des y. Voir les autres questions fréquemment posées : ggpubr FAQ.
Version:
 English
English






No Comments