Cet article décrit comment calculer et ajouter automatiquement des p-values sur des graphiques ggplot multi-panneaux obtenus avec facet en utilisant les packages R ggpubr et rstatix.
Vous apprendrez à:
- Ajouter des p-values à un ggplot multi-panneaux (facet) contenant deux ou plusieurs groupes par panneau. Des exemples sont représentés pour les box plots et les bar plots.
- Ajouter des p-values à un graphique groupé multi-panneaux obtenu avec ggplot facet (box plots et bar plots). Des exemples, contenant trois groupes par position x, sont présentés.
- Afficher les p-values combinées aux niveaux de significativité sur les graphiques
Nous suivrons les étapes suivantes pour ajouter des niveaux de significativité sur un ggplot:
- Calculer facilement des tests statistiques (
t_test()ouwilcox_test()) en utilisant le packagerstatix - Auto-calculez les positions des étiquettes des p-values en utilisant la fonction
add_xy_position()[dans le package rstatix]. - Ajoutez les p-values au graphique en utilisant la fonction
stat_pvalue_manual()[dans le package ggpubr]. Les options clés suivantes sont illustrées dans certains des exemples:- L’option
bracket.nudge.yest utilisée pour monter ou descendre les crochets. - L’option
step.increaseest utilisée pour ajouter de l’espace entre les crochets. - L’option
vjustest utilisée pour ajuster verticalement la position des étiquettes des p-values
- L’option
Notez que, dans certains cas, les étiquettes de p-values sont partiellement cachées par la bordure supérieure du graphique. Dans ce cas, la fonction ggplot2 scale_y_continuous(expand = expansion(mult = c(0, 0.1))) peut être utilisée pour ajouter des espaces entre les étiquettes et la bordure supérieure du graphique. L’option mult = c(0, 0.1) indique que des espaces de 0% et 10% sont respectivement ajoutés en bas et en haut du graphique.
Sommaire:
Prérequis
Assurez-vous d’avoir installé les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixcontient des fonctions R facilitant les analyses statistiques.
Commencez par charger les packages requis suivants:
library(ggpubr)
library(rstatix)Préparation des données
# Transformer `dose` en variable factorielle
df <- ToothGrowth
df$dose <- as.factor(df$dose)
# Ajouter une variable de regroupement aléatoire
df$group <- factor(rep(c("grp1", "grp2"), 30))
head(df, 3)## len supp dose group
## 1 4.2 VC 0.5 grp1
## 2 11.5 VC 0.5 grp2
## 3 7.3 VC 0.5 grp1Graphiques multi-panneaux contenant deux groupes par panneau
Facet wrap avec une variable
Tests statistiques
Faire un ggplot de la variable dose et comparer les niveaux de la variable supp sur l’axe des x.
stat.test <- df %>%
group_by(dose) %>%
t_test(len ~ supp) %>%
adjust_pvalue(method = "bonferroni") %>%
add_significance()
stat.test ## # A tibble: 3 x 11
## dose .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 0.5 len OJ VC 10 10 3.17 15.0 0.00636 0.0191 *
## 2 1 len OJ VC 10 10 4.03 15.4 0.00104 0.00312 **
## 3 2 len OJ VC 10 10 -0.0461 14.0 0.964 1 nsBox plots
# Créer un box plot
bxp <- ggboxplot(
df, x = "supp", y = "len", fill = "#00AFBB",
facet.by = "dose"
)
# Faire un ggplot facet et ajouter des p-values
stat.test <- stat.test %>% add_xy_position(x = "supp")
bxp + stat_pvalue_manual(stat.test)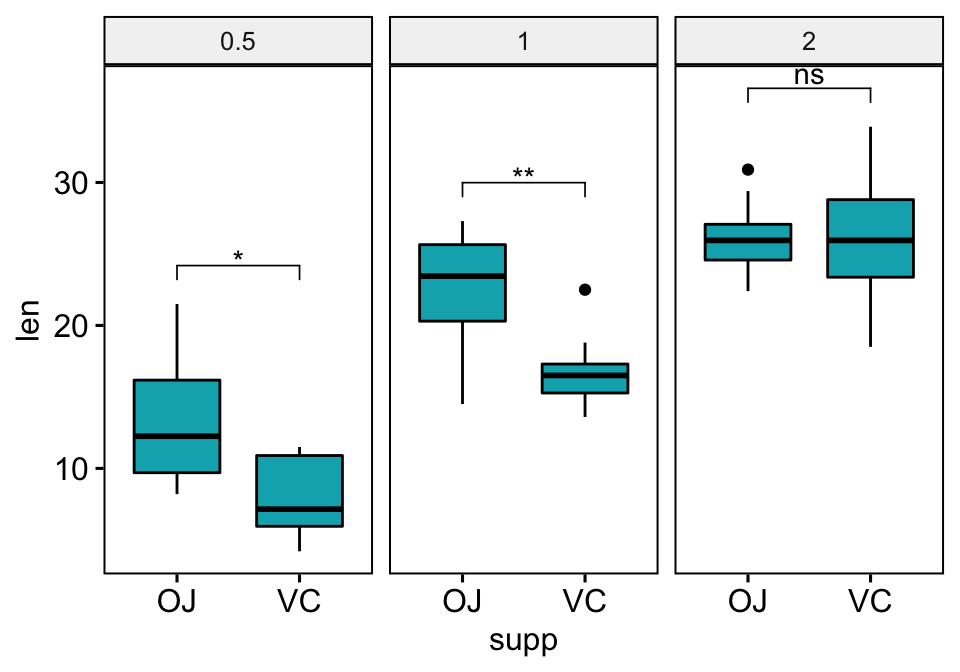
# Faire un ggplot facet à échelle libre et ajouter les points dispersés (jitter)
# Faites baisser le crochet en utilisant `bracket.nudge.y`
# Cacher les ns (non significatif)
# Afficher les p-values ajustées et les niveaux de significativité
# Ajoutez 10 % d'espaces entre les étiquettes des p-values et la bordure du graphique
bxp <- ggboxplot(
df, x = "supp", y = "len", fill = "#00AFBB",
facet.by = "dose", scales = "free", add = "jitter"
)
bxp +
stat_pvalue_manual(
stat.test, bracket.nudge.y = -2, hide.ns = TRUE,
label = "{p.adj}{p.adj.signif}"
) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.1)))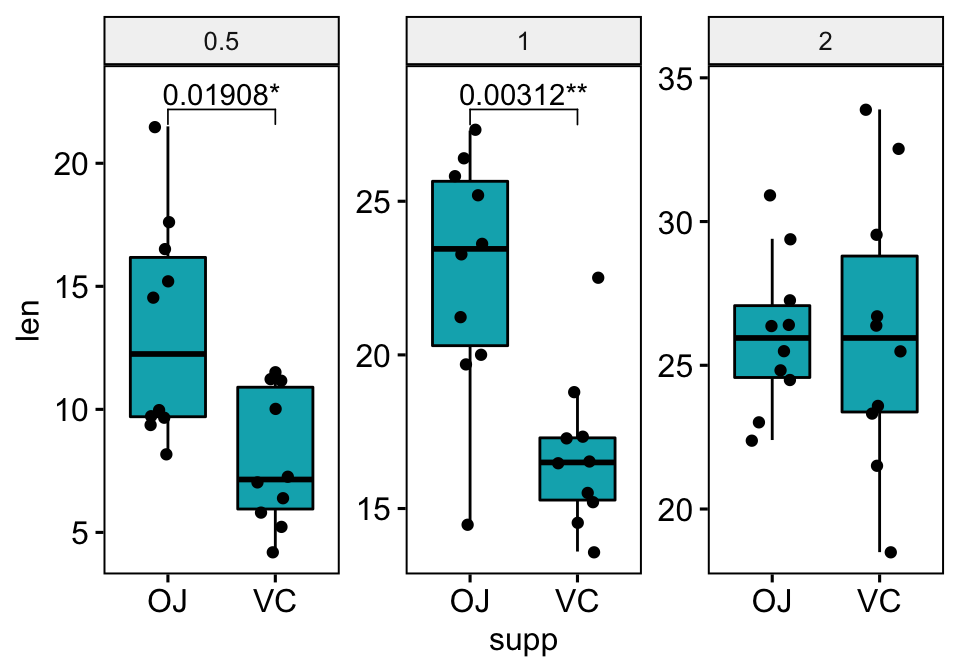
Bar plots
L’option add = "mean_sd" est spécifiée dans la fonction de graphique à barres pour créer un graphique à barres avec des barres d’erreur (mean +/- SD). Vous devez spécifier la même fonction de statistiques sommaires pour calculer automatiquement les positions des étiquettes de p-values dans add_xy_position() en utilisant l’option fun.
# Créer un graphique avec des barres d'erreur (moyenne +/- sd)
bp <- ggbarplot(
df, x = "supp", y = "len", add = "mean_sd",
fill = "#00AFBB", facet.by = "dose"
)
# Ajoutez des p-values sur les graphiques en barres
stat.test <- stat.test %>% add_xy_position(fun = "mean_sd", x = "supp")
bp + stat_pvalue_manual(stat.test)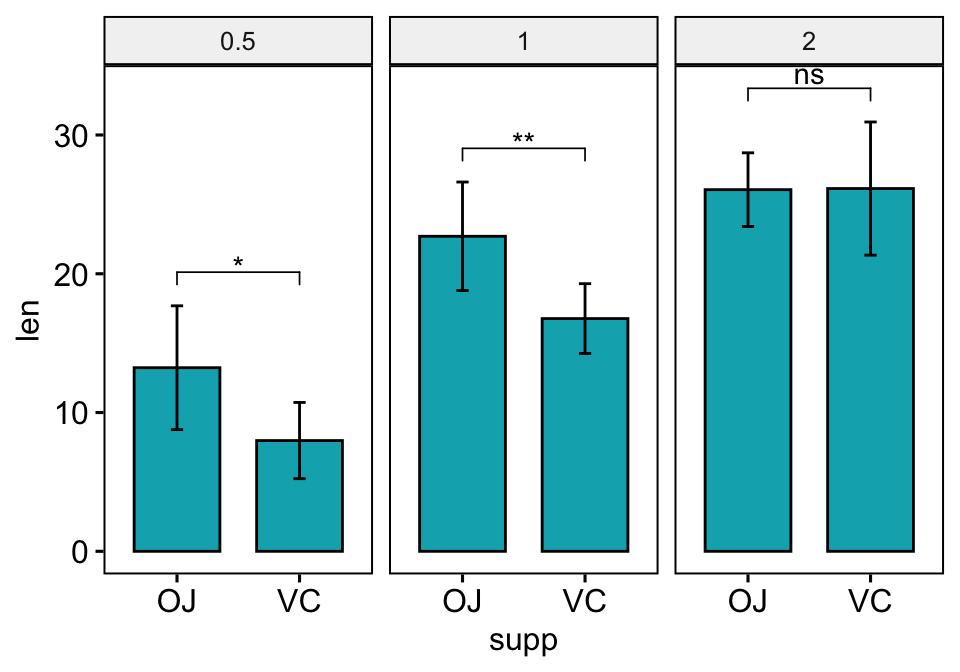
Bar plots avec des points dispersés (jitter)
Vous devez spécifier fun = "max" lors du calcul des positions des étiquettes des p-values, de sorte que le premier crochet commence au maximum des points de données. Cela permet d’éviter le chevauchement entre les points de données et les crochets.
# Créer un graphique avec des barres d'erreur (moyenne +/- sd)
bp <- ggbarplot(
df, x = "supp", y = "len", add = c("mean_sd", "jitter"),
fill = "#00AFBB", facet.by = "dose"
)
# Ajoutez des p-values sur les graphiques en barres
stat.test <- stat.test %>% add_xy_position(fun = "max", x = "supp")
bp + stat_pvalue_manual(stat.test)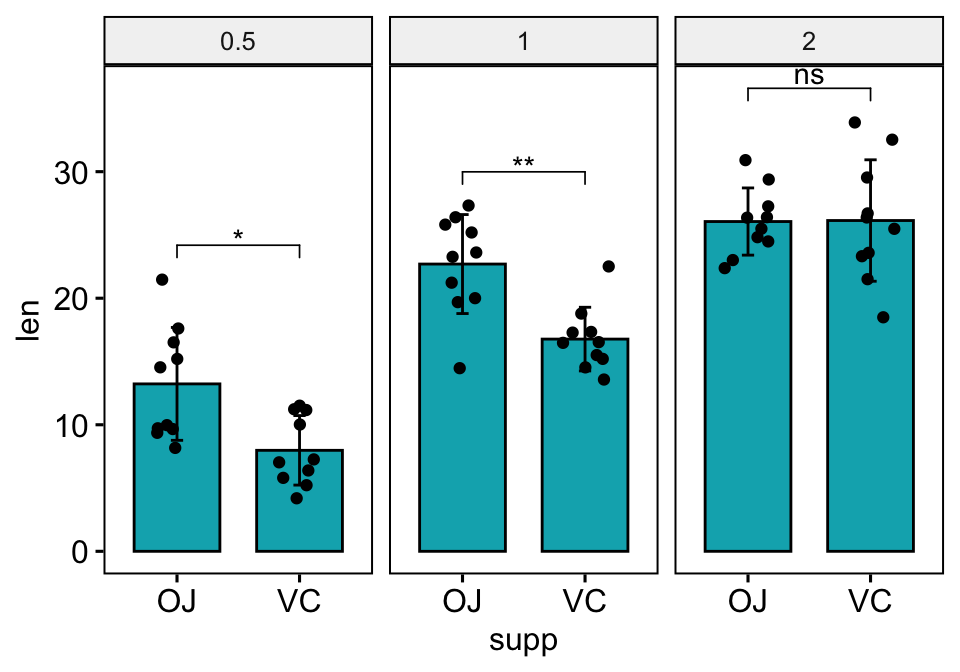
Facet grid avec deux variables
Test statistique
Faire un ggplot facet en fonction des variables dose et group, et comparaison des niveaux de la variable supp sur l’axe des abscisses.
stat.test <- df %>%
group_by(group, dose) %>%
t_test(len ~ supp) %>%
adjust_pvalue(method = "bonferroni") %>%
add_significance()
stat.test ## # A tibble: 6 x 12
## dose group .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## <fct> <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 0.5 grp1 len OJ VC 5 5 3.71 7.35 0.00697 0.0418 *
## 2 1 grp1 len OJ VC 5 5 1.37 7.65 0.208 1 ns
## 3 2 grp1 len OJ VC 5 5 -0.485 6.52 0.643 1 ns
## 4 0.5 grp2 len OJ VC 5 5 1.13 5.79 0.304 1 ns
## 5 1 grp2 len OJ VC 5 5 6.91 5.84 0.00051 0.00306 **
## 6 2 grp2 len OJ VC 5 5 0.319 6.07 0.76 1 nsBox plots
# Créer des box plots avec les niveaux de significativité
# Cacher les ns (non significatif)
stat.test <- stat.test %>% add_xy_position(x = "supp")
ggboxplot(
df, x = "supp", y = "len", fill = "#E7B800",
facet = c("group", "dose")
) +
stat_pvalue_manual(stat.test, hide.ns = TRUE)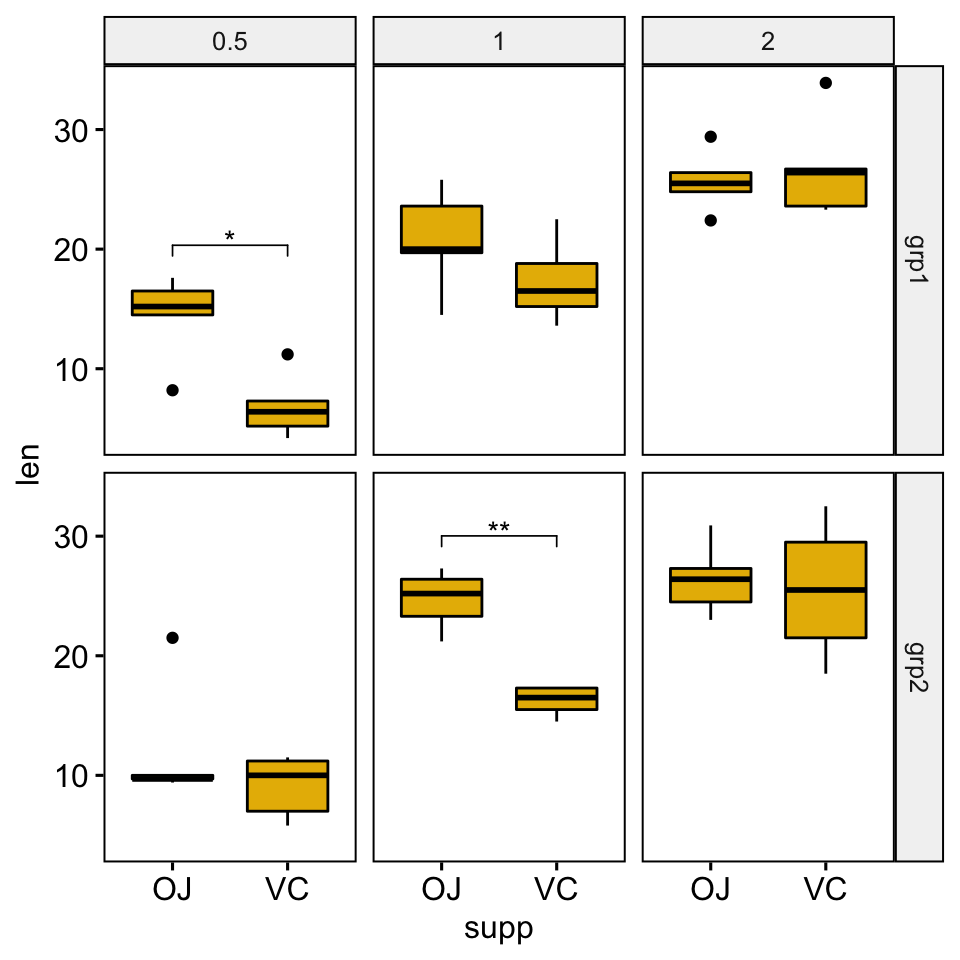
Bar plots
# Créer des bar plots avec les niveaux de significativité
# Cacher les ns (non significatif)
stat.test <- stat.test %>% add_xy_position(x = "supp", fun = "mean_sd")
ggbarplot(
df, x = "supp", y = "len", fill = "#E7B800",
add = c("mean_sd", "jitter"), facet = c("group", "dose")
) +
stat_pvalue_manual(stat.test, hide.ns = TRUE)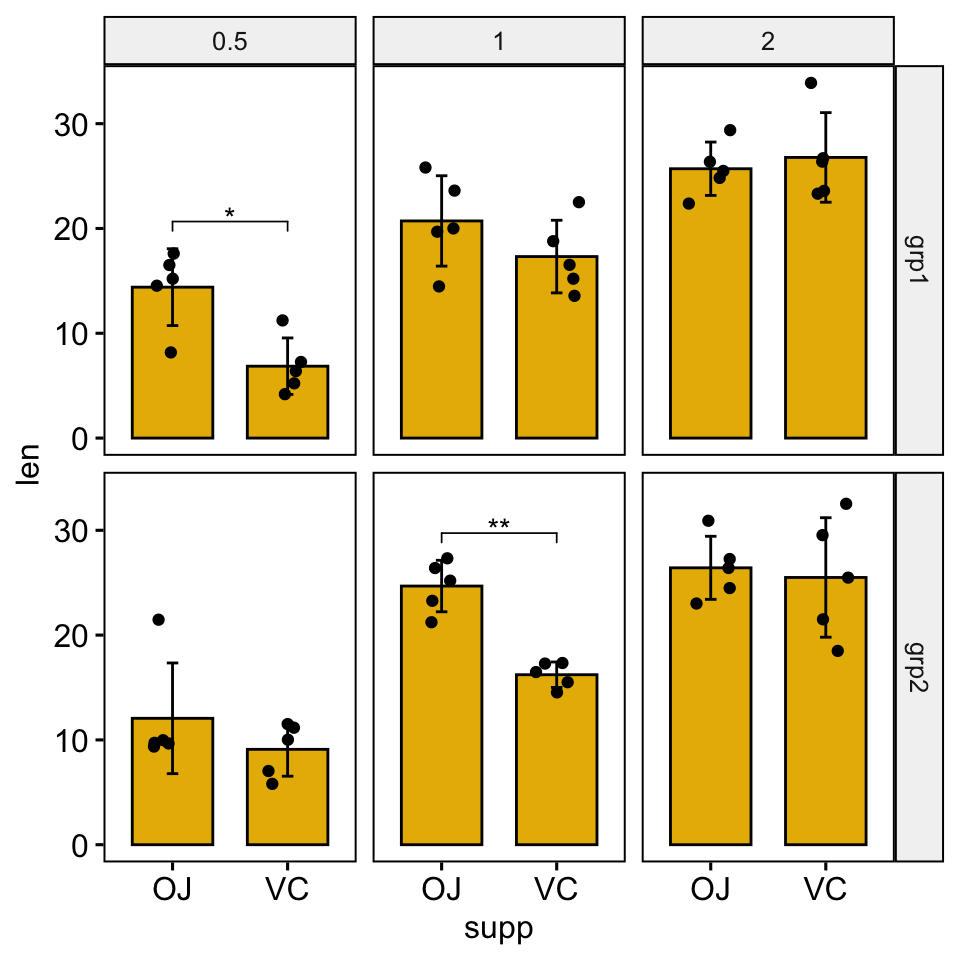
Graphiques multi-panneaux contenant trois groupes ou plus par panneau
Facet wrap avec une variable
Effectuer toutes les comparaisons par paires
Regrouper par la variable supp et ensuite effectuer des comparaisons par paires entre les niveaux de la variable dose.
Test statistique:
stat.test <- df %>%
group_by(supp) %>%
t_test(len ~ dose)
stat.test## # A tibble: 6 x 11
## supp .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 OJ len 0.5 1 10 10 -5.05 17.7 0.0000878 0.000176 ***
## 2 OJ len 0.5 2 10 10 -7.82 14.7 0.00000132 0.00000396 ****
## 3 OJ len 1 2 10 10 -2.25 15.8 0.039 0.039 *
## 4 VC len 0.5 1 10 10 -7.46 17.9 0.000000681 0.00000136 ****
## 5 VC len 0.5 2 10 10 -10.4 14.3 0.0000000468 0.00000014 ****
## 6 VC len 1 2 10 10 -5.47 13.6 0.0000916 0.0000916 ****Ajouter les p-values sur les graphiques. La fonction ggplot2 scale_y_continuous(expand = expansion(mult = c(0, 0.1))) est utilisée pour ajouter des espaces supplémentaires entre les étiquettes et la bordure supérieure du graphique
# Box plot avec p-values
stat.test <- stat.test %>% add_y_position()
ggboxplot(df, x = "dose", y = "len", fill = "#FC4E07", ggplot = "supp") +
stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.1)))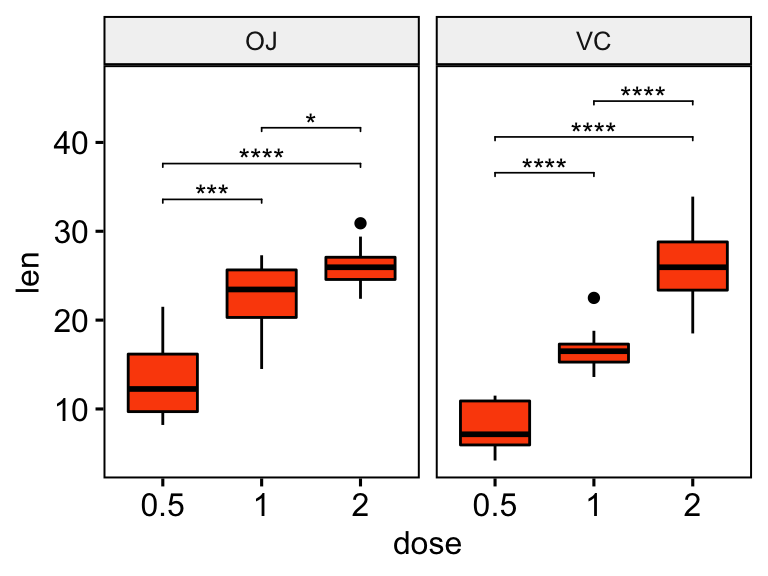
# Bar plot avec p-values
# Ajouter 10 % d'espace sur l'axe des y au-dessus des graphiques
stat.test <- stat.test %>% add_y_position(fun = "mean_sd")
ggbarplot(
df, x = "dose", y = "len", fill = "#FC4E07",
add = "mean_sd", facet.by = "supp"
) +
stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.1)))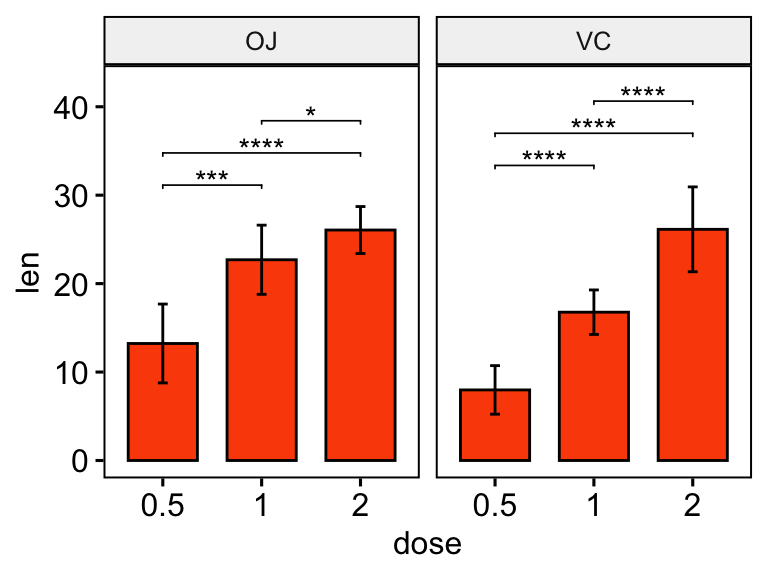
Comparaisons par paires par rapport à un groupe de référence
Test statistique:
stat.test <- df %>%
group_by(supp) %>%
t_test(len ~ dose, ref.group = "0.5")
stat.test## # A tibble: 4 x 11
## supp .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 OJ len 0.5 1 10 10 -5.05 17.7 0.0000878 0.0000878 ****
## 2 OJ len 0.5 2 10 10 -7.82 14.7 0.00000132 0.00000264 ****
## 3 VC len 0.5 1 10 10 -7.46 17.9 0.000000681 0.000000681 ****
## 4 VC len 0.5 2 10 10 -10.4 14.3 0.0000000468 0.0000000936 ****# Box plot avec p-values
stat.test <- stat.test %>% add_y_position()
ggboxplot(df, x = "dose", y = "len", fill = "#FC4E07", ggplot = "supp") +
stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.1)))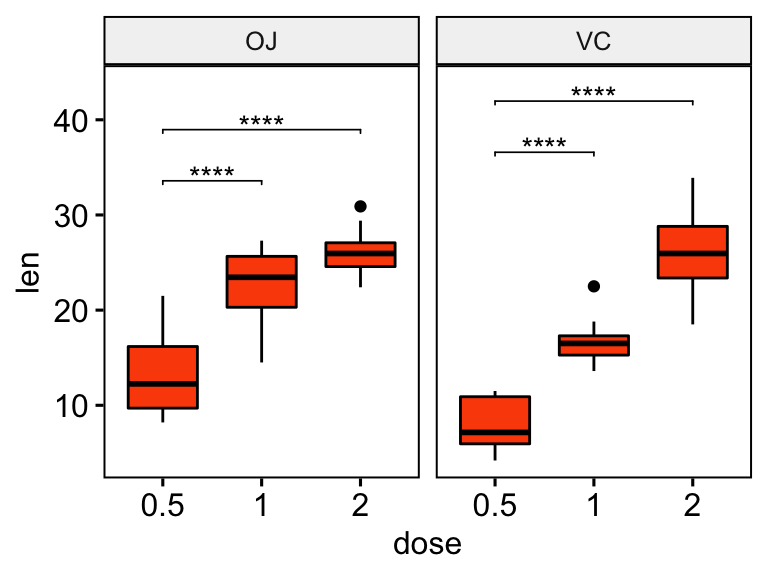
# Indiquer que les niveaux de significativité à x = groupe2
# Faites baisser les symboles de significativité en utilisant vjust
stat.test <- stat.test %>% add_y_position()
ggboxplot(df, x = "dose", y = "len", fill = "#FC4E07", ggplot = "supp") +
stat_pvalue_manual(stat.test, label = "p.adj.signif", x = "group2", vjust = 2) 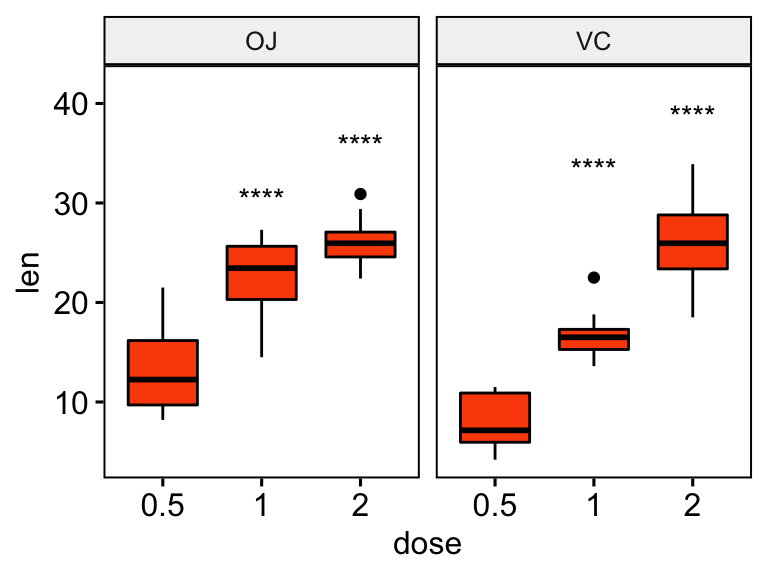
# Bar plot avec p-values
# Ajouter 10 % d'espace sur l'axe des y au-dessus des graphiques
stat.test <- stat.test %>% add_y_position(fun = "mean_sd")
ggbarplot(
df, x = "dose", y = "len", fill = "#FC4E07",
add = c("mean_sd", "jitter"), facet.by = "supp"
) +
stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.1)))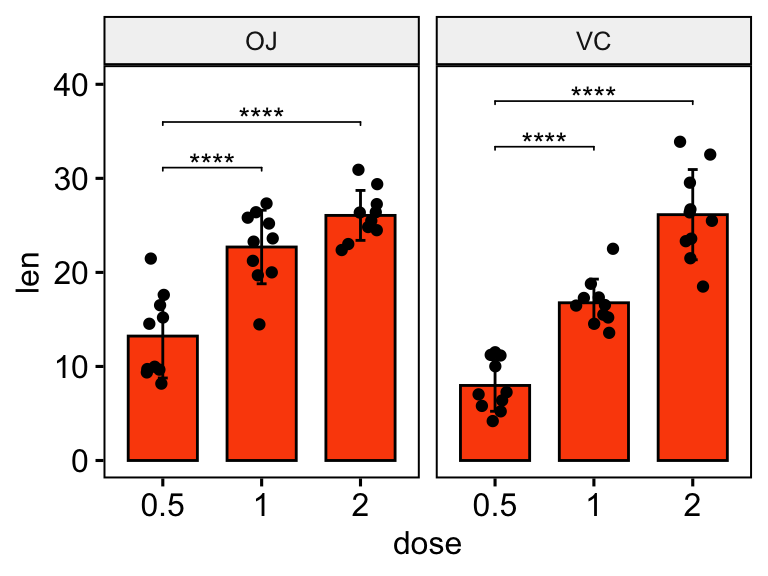
Facet grid avec deux variables
Test statistique
Faire un ggplot avec les variables supp et group, et comparer les niveaux de la variable dose sur l’axe des x.
stat.test <- df %>%
group_by(group, supp) %>%
t_test(len ~ dose) %>%
adjust_pvalue(method = "bonferroni") %>%
add_significance()
stat.test ## # A tibble: 12 x 12
## supp group .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## <fct> <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 OJ grp1 len 0.5 1 5 5 -2.50 7.80 0.038 0.456 ns
## 2 OJ grp1 len 0.5 2 5 5 -5.66 7.13 0.000717 0.00860 **
## 3 OJ grp1 len 1 2 5 5 -2.22 6.49 0.064 0.768 ns
## 4 VC grp1 len 0.5 1 5 5 -5.33 7.54 0.000855 0.0103 *
## 5 VC grp1 len 0.5 2 5 5 -8.81 6.75 0.0000606 0.000727 ***
## 6 VC grp1 len 1 2 5 5 -3.84 7.67 0.005 0.06 ns
## # … with 6 more rowsBox plots
# Créer des box plots avec les niveaux de significativité
# Cacher les ns (non significatif)
# Ajoutez 15 % d'espace entre les étiquettes et la bordure supérieure du graphique
stat.test <- stat.test %>% add_xy_position(x = "dose")
ggboxplot(
df, x = "dose", y = "len", fill = "#FC4E07",
facet = c("group", "supp")
) +
stat_pvalue_manual(stat.test, hide.ns = TRUE) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.15)))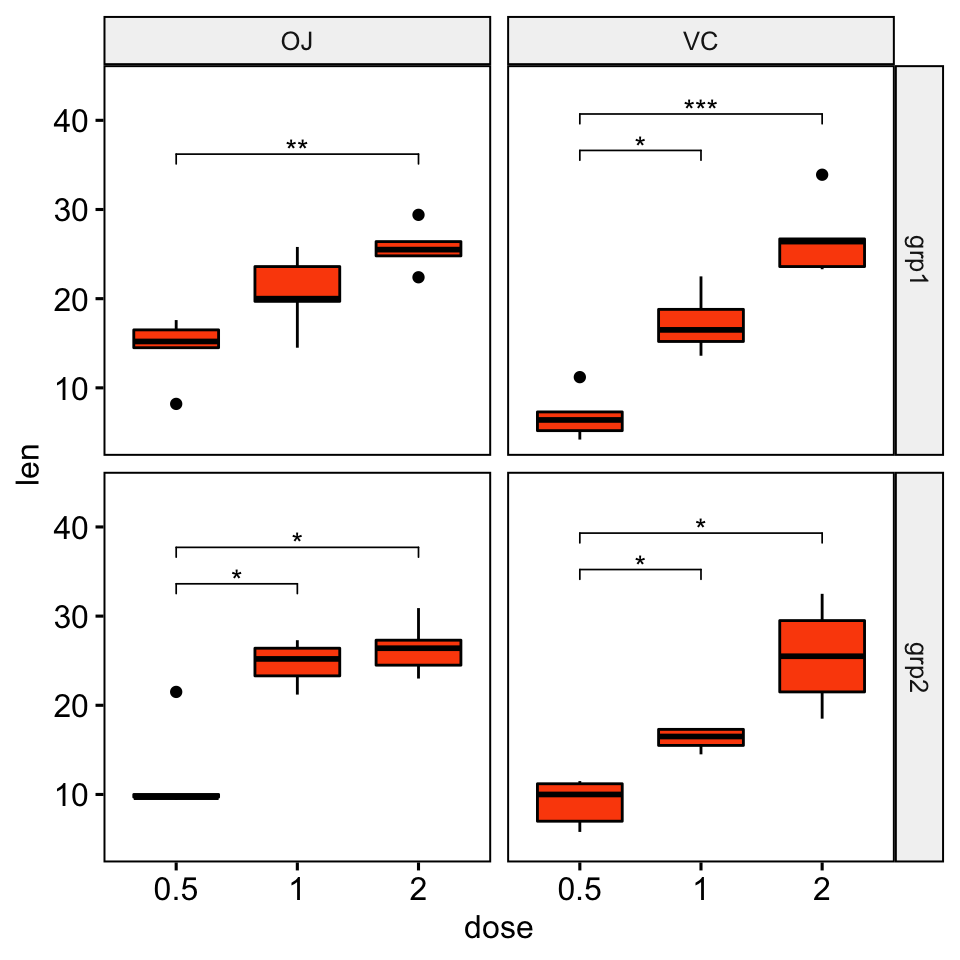
Bar plots
# Créer des bar plots avec les niveaux de significativité
# Cacher les ns (non significatif)
# Ajoutez 15 % d'espace entre les étiquettes et la bordure supérieure du graphique
stat.test <- stat.test %>% add_xy_position(x = "dose", fun = "mean_sd")
ggbarplot(
df, x = "dose", y = "len", fill = "#FC4E07",
add = c("mean_sd", "jitter"), facet = c("group", "supp")
) +
stat_pvalue_manual(stat.test, hide.ns = TRUE) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.15)))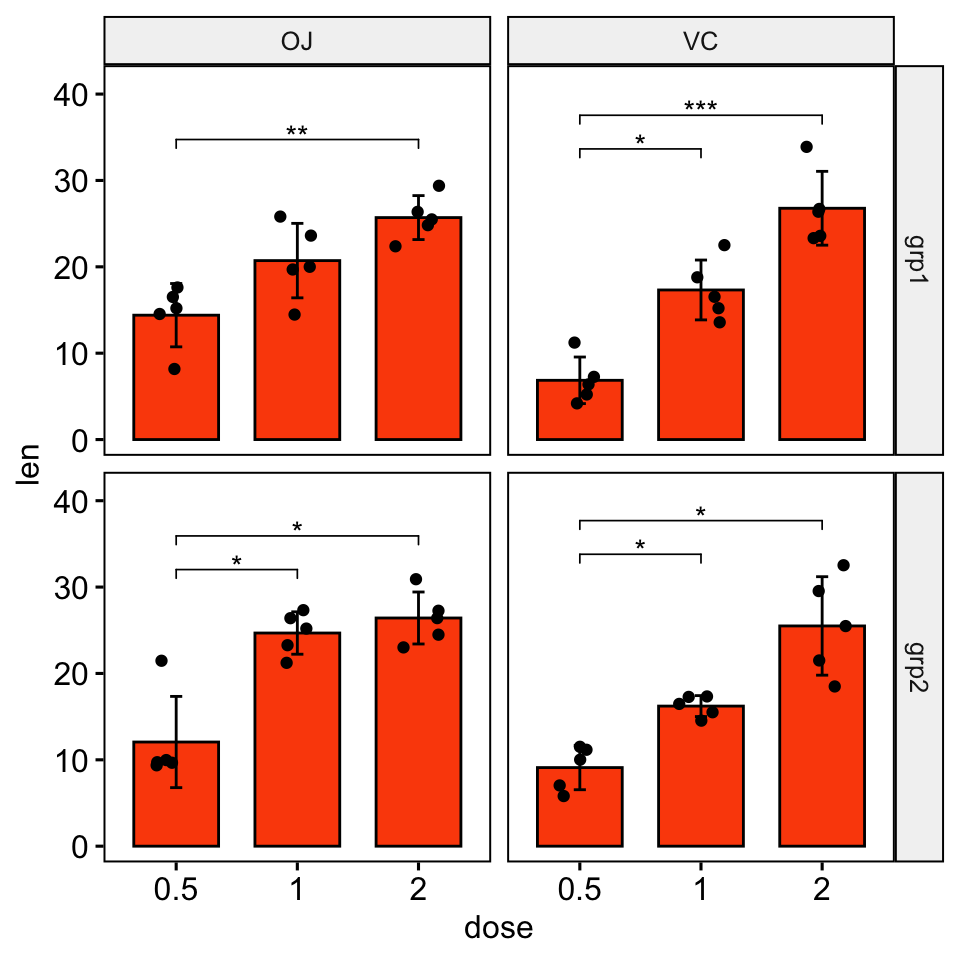
Des graphiques groupés à facet
Graphiques simples
# Box plots
bxp <- ggboxplot(
df, x = "supp", y = "len", color = "dose",
palette = "jco", facet.by = "group"
)
bxp 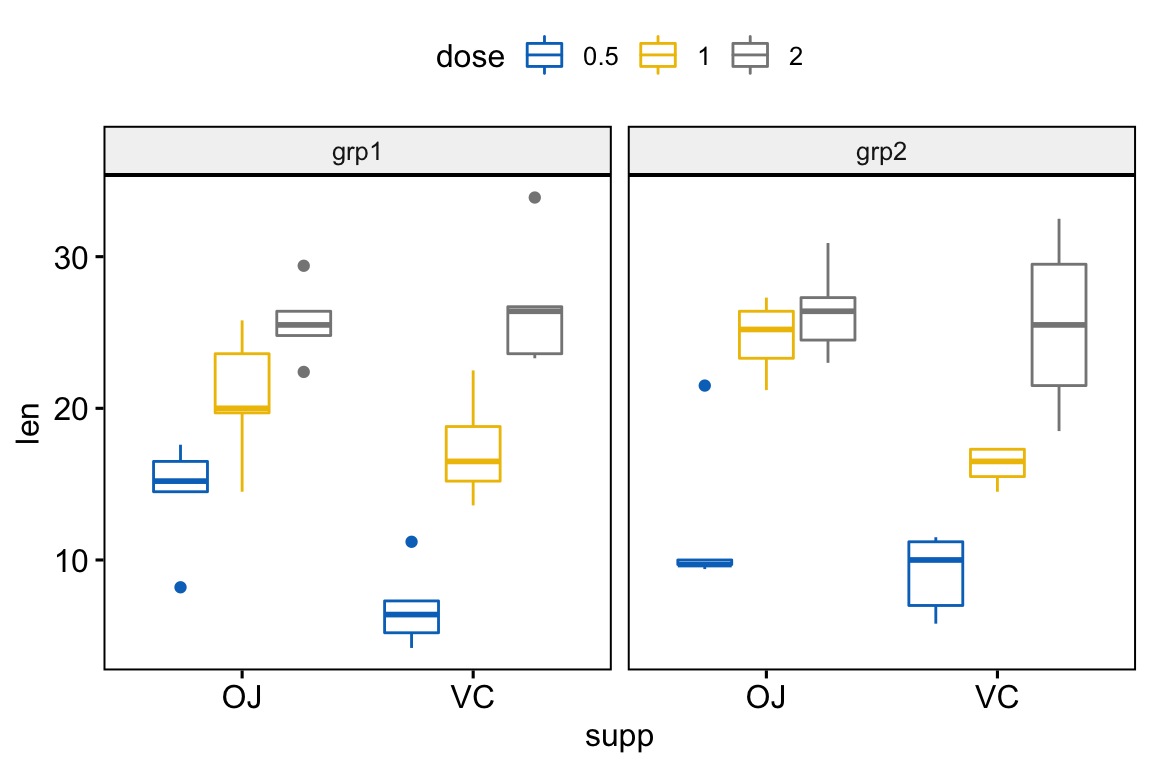
# Bar plots
bp <- ggbarplot(
df, x = "supp", y = "len", color = "dose",
palette = "jco", add = "mean_sd",
position = position_dodge(0.8),
facet.by = "group"
)
bp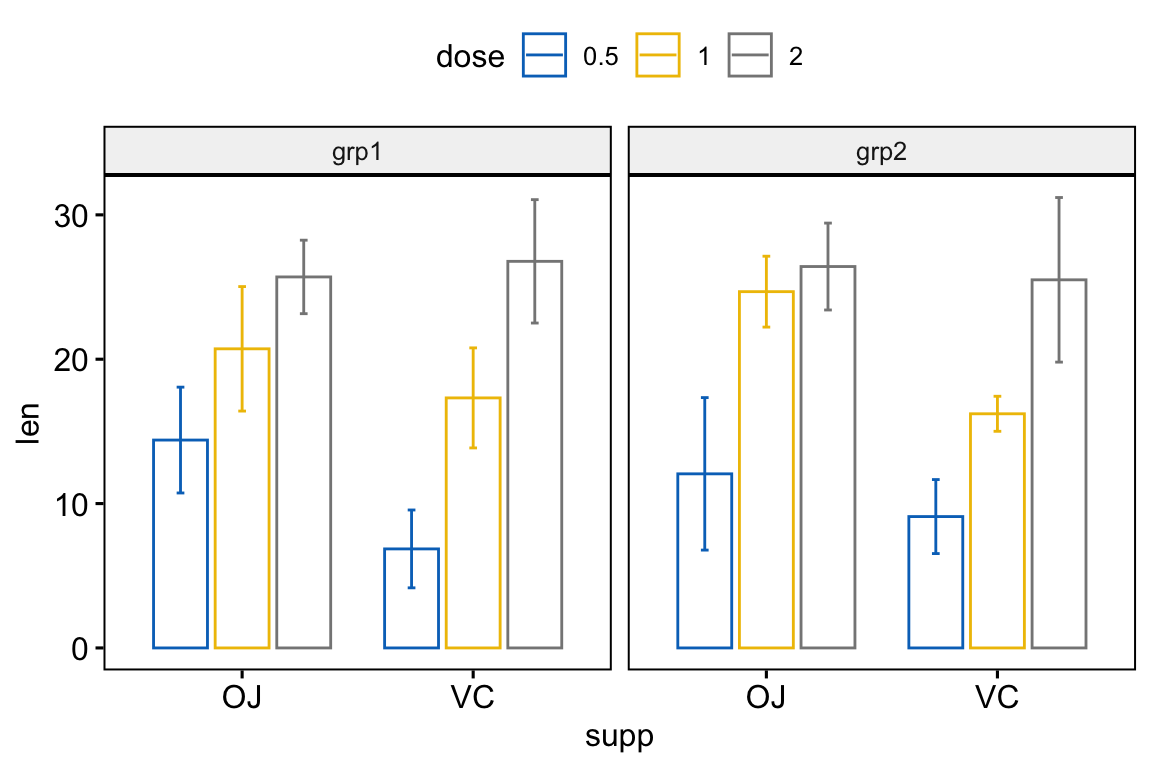
Effectuer toutes les comparaisons par paires
Test statistique:
stat.test <- df %>%
group_by(supp, group) %>%
t_test(len ~ dose)
stat.test## # A tibble: 12 x 12
## supp group .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <fct> <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 OJ grp1 len 0.5 1 5 5 -2.50 7.80 0.038 0.076 ns
## 2 OJ grp1 len 0.5 2 5 5 -5.66 7.13 0.000717 0.002 **
## 3 OJ grp1 len 1 2 5 5 -2.22 6.49 0.064 0.076 ns
## 4 OJ grp2 len 0.5 1 5 5 -4.85 5.65 0.003 0.007 **
## 5 OJ grp2 len 0.5 2 5 5 -5.28 6.35 0.002 0.005 **
## 6 OJ grp2 len 1 2 5 5 -1.00 7.69 0.347 0.347 ns
## # … with 6 more rowsAjouter des p-values sur les graphiques:
# Box plot avec p-values
# Cacher les ns (non significatif)
stat.test <- stat.test %>%
add_xy_position(x = "supp", dodge = 0.8)
bxp +
stat_pvalue_manual(
stat.test, label = "p.adj.signif", tip.length = 0.01,
hide.ns = TRUE
) +
scale_y_continuous(expand = expansion(mult = c(0.01, 0.1)))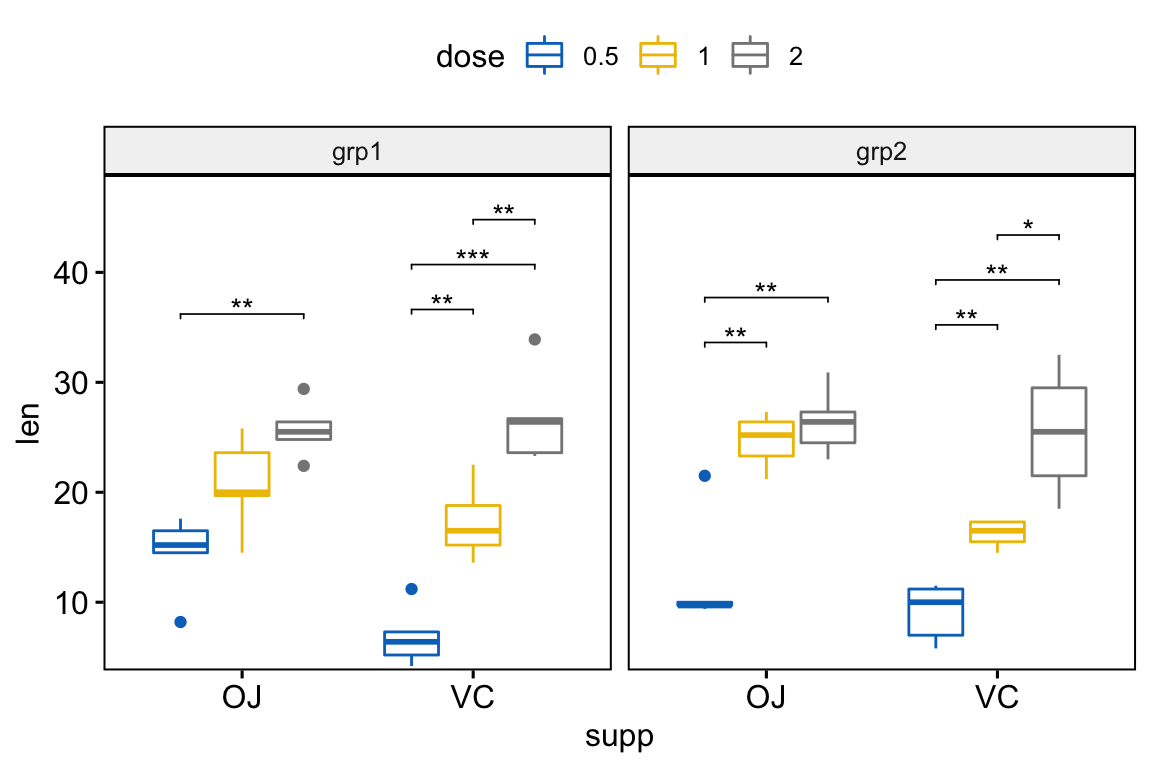
# Bar plot avec p-values
stat.test <- stat.test %>%
add_xy_position(x = "supp", fun = "mean_sd", dodge = 0.8)
bp +
stat_pvalue_manual(
stat.test, label = "p.adj.signif", tip.length = 0.01,
hide.ns = TRUE
) +
scale_y_continuous(expand = expansion(mult = c(0.01, 0.1)))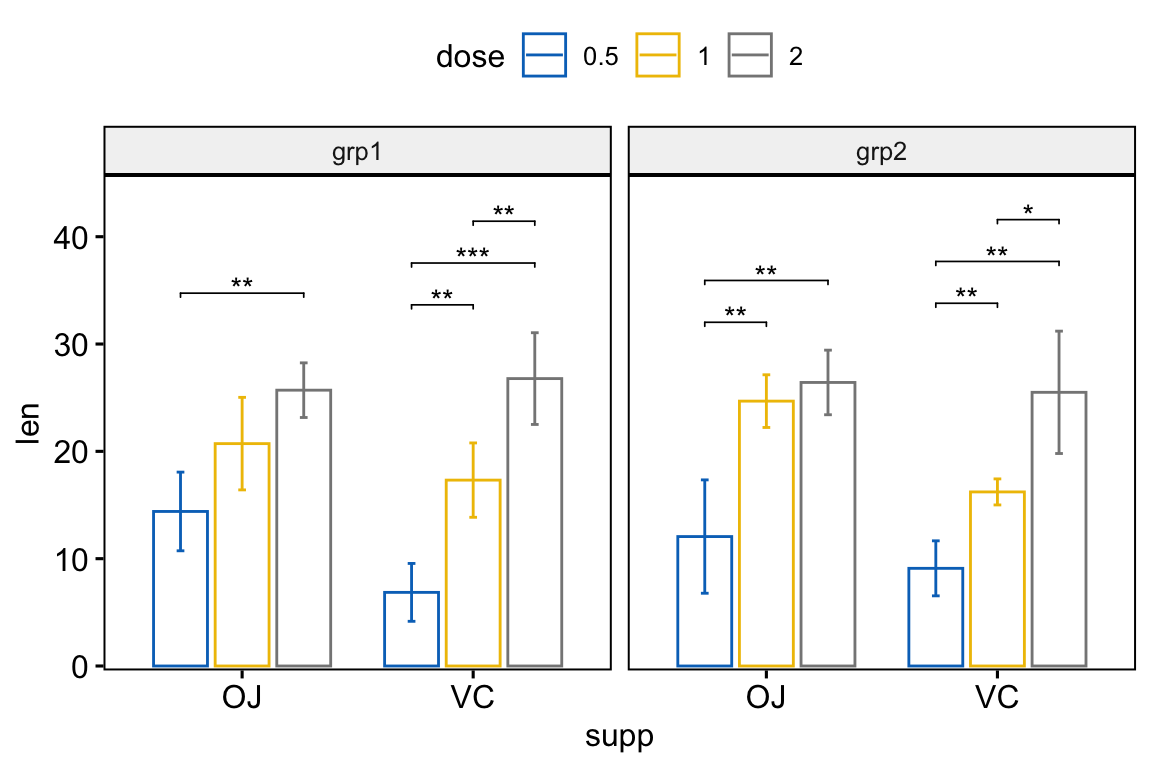
Comparaisons par paires par rapport à un groupe de référence
Test statistique:
stat.test <- df %>%
group_by(supp, group) %>%
t_test(len ~ dose, ref.group = "0.5")
stat.test## # A tibble: 8 x 12
## supp group .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <fct> <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 OJ grp1 len 0.5 1 5 5 -2.50 7.80 0.038 0.038 *
## 2 OJ grp1 len 0.5 2 5 5 -5.66 7.13 0.000717 0.001 **
## 3 OJ grp2 len 0.5 1 5 5 -4.85 5.65 0.003 0.003 **
## 4 OJ grp2 len 0.5 2 5 5 -5.28 6.35 0.002 0.003 **
## 5 VC grp1 len 0.5 1 5 5 -5.33 7.54 0.000855 0.000855 ***
## 6 VC grp1 len 0.5 2 5 5 -8.81 6.75 0.0000606 0.000121 ***
## # … with 2 more rowsAjouter des p-values sur les graphiques:
# Box plot avec p-values
stat.test <- stat.test %>%
add_xy_position(x = "supp", dodge = 0.8)
bxp +
stat_pvalue_manual(
stat.test, label = "p.adj.signif", tip.length = 0.01
) +
scale_y_continuous(expand = expansion(mult = c(0.01, 0.1)))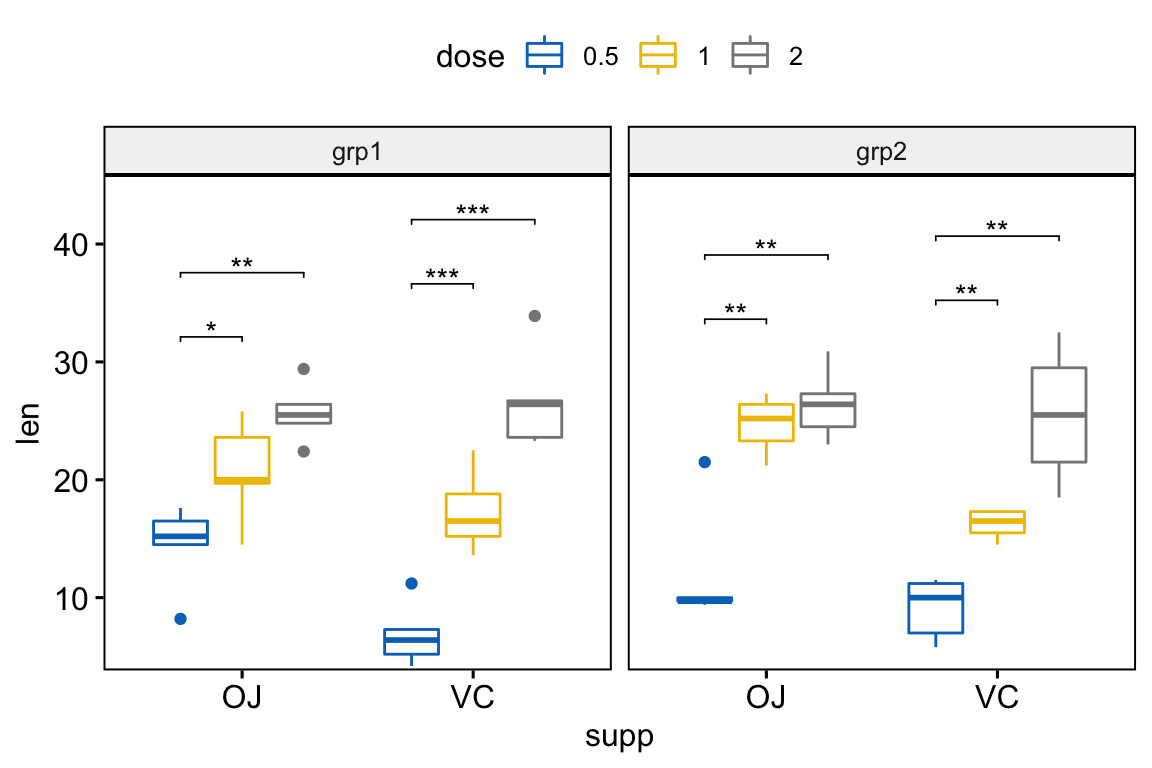
# Bar plot avec p-values
stat.test <- stat.test %>%
add_xy_position(x = "supp", fun = "mean_sd", dodge = 0.8)
bp +
stat_pvalue_manual(
stat.test, label = "p.adj.signif", tip.length = 0.01
) +
scale_y_continuous(expand = expansion(mult = c(0.01, 0.1)))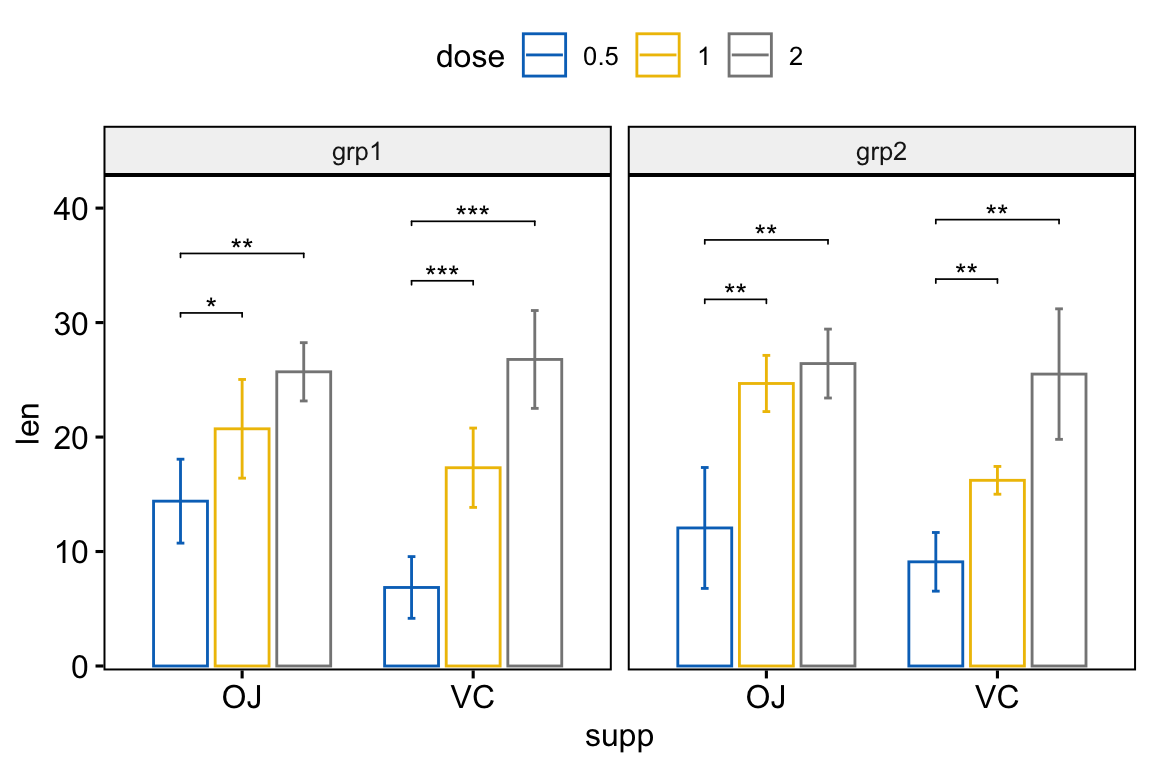
Conclusion
Cet article décrit comment ajouter des p-values sur des ggplots facets, telles que les box plots et les bar plots. Voir les autres questions fréquemment posées : ggpubr FAQ.
Version:
 English
English






No Comments