Le test ANOVA (ou Analyse de variance) est utilisé pour comparer la moyenne de plusieurs groupes. Le terme ANOVA est un peu trompeur. Bien que le nom de la technique fasse référence aux variances, l’objectif principal de l’ANOVA est d’étudier les différences de moyennes.
Ce chapitre décrit les différents types d’ANOVA pour comparer des groupes indépendants, notamment:
- ANOVA à un facteur : une extension du test t pour échantillons indépendants comparant les moyennes dans une situation où il y a plus de deux groupes. C’est le cas le plus simple du test ANOVA où les données sont organisées en plusieurs groupes en fonction d’une seule variable de groupement (aussi appelée facteur). D’autres synonymes sont : ANOVA à 1 facteur, ANOVA à un facteur et ANOVA inter-sujets.
- ANOVA à deux facteurs utilisée pour évaluer simultanément l’effet de deux variables de groupement différentes sur une variable-réponse continue. D’autres synonymes sont : plans factoriels à deux niveaux, Anova factoriels ou ANOVA à deux facteurs-inter-sujets.
- ANOVA à trois facteurs utilisée pour évaluer simultanément l’effet de trois variables de groupement différentes sur une variable-réponse continue. D’autres synonymes sont : ANOVA factorielle ou ANOVA inter-sujets à trois facteurs.
Il est à noter que les variables de regroupement indépendantes sont également connues sous le nom de facteurs inter-sujets.
Le but principal de l’ANOVA à deux facteurs et de l’ANOVA à trois facteurs est, respectivement, d’évaluer s’il existe un effet d’interaction statistiquement significatif entre deux et trois facteurs pour expliquer une variable-réponse continue.
Vous apprendrez à:
- Calculer et interpréter les différents types d’ANOVA dans R pour comparer des groupes indépendants.
- Vérifier les hypothèses du test ANOVA
- Effectuer des tests post-hoc, de multiples comparaisons par paires entre les groupes pour identifier les groupes qui sont différents
- Visualiser les données avec des boxplots, ajouter au graphique, les p-values de l’ANOVA et celles des comparaisons multiples par paires
Sommaire:
Livre Apparenté
Pratique des Statistiques dans R II - Comparaison de Groupes: Variables NumériquesNotions de base
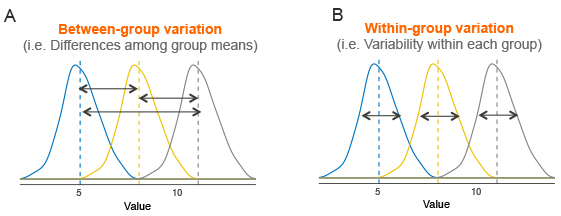
Supposons que nous ayons 3 groupes à comparer, comme illustré dans l’image ci-dessous. La ligne en pointillés indique la moyenne du groupe. La figure montre la variation entre les moyennes des groupes (panel A) et la variation au sein de chaque groupe (panel B), également appelée variance résiduelle.
L’idée qui sous-tend le test ANOVA est très simple : si la variation moyenne entre les groupes est suffisamment importante par rapport à la variation moyenne au sein des groupes, on peut conclure qu’au moins la moyenne d’un des groupes n’est pas égale aux autres.
Il est donc possible d’évaluer si les différences entre les moyennes des groupes sont significatives en comparant les deux estimations de variance. C’est pourquoi la méthode est appelée analyse de variance alors que l’objectif principal est de comparer les moyennes des groupes.
Brièvement, la procédure mathématique du test ANOVA est la suivante:
- Calculer la variance intra-groupe, aussi appelée variance résiduelle. Cela nous indique à quel point chaque participant est différent de la moyenne de son propre groupe (voir figure, panel B).
- Calculer la variance entre les moyennes des groupes (voir figure, pannel A).
- Calculez la statistique F comme étant :
variance.inter.groups/variance.intra.groups.
Notez qu’une valeur petite de F (F < 1) indique qu’il n’y a pas de différence significative entre les moyennes des échantillons comparés.
Cependant, un ratio plus élevé signifie que les variations entre les moyennes des groupes sont très différentes les unes des autres par rapport à la variation des observations individuelles dans chaque groupe.
Hypothèses
Le test ANOVA pose les hypothèses suivantes au sujet des données:
- Indépendance des observations. Chaque sujet ne doit appartenir qu’à un seul groupe. Il n’y a aucun lien entre les observations de chaque groupe. Il n’est pas permis d’avoir des mesures répétées pour les mêmes participants.
- Aucune valeur aberrante significative dans aucune cellule du plan
- Normalité. les données de chaque cellule du plan devraient être distribuées approximativement normalement.
- Homogénéité des variances. La variance de la variable-réponse doit être égale dans chaque cellule du plan expérimental.
Avant de calculer le test ANOVA, vous devez effectuer quelques tests préliminaires pour vérifier si les hypothèses sont remplies.
Notez que, si les hypothèses ci-dessus ne sont pas satisfaites, il existe une alternative non paramétrique (test de Kruskal-Wallis) à l’ANOVA à un facteur.
Malheureusement, il n’existe pas d’alternative non paramétrique à l’ANOVA à deux et à trois facteurs. Ainsi, dans le cas où les hypothèses ne sont pas satisfaites, vous pourriez envisager d’exécuter l’ANOVA à deux/trois facteurs sur les données transformées et non transformées pour voir s’il y a des différences significatives.
Si les deux tests vous amènent aux mêmes conclusions, vous pourriez choisir de ne pas transformer la variable-réponse et de poursuivre l’ANOVA à deux/trois facteurs sur les données initiales.
Il est également possible d’effectuer un test ANOVA robuste à l’aide du package R WRS2.
Quel que soit votre choix, vous devez rapporter ce que vous avez fait dans vos résultats.
Prérequis
Assurez-vous d’avoir les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixoffre des fonctions R conviviales pour des analyses statistiques faciles à réaliserdatarium: contient les jeux de données requis pour ce chapitre
Charger les packages R requis:
library(tidyverse)
library(ggpubr)
library(rstatix)Fonctions R clés : anova_test() [paquet rstatix], wrapper autour de la fonction car::Anova().
ANOVA à un facteur
Préparation des données
Ici, nous utiliserons le jeu de données intégré à R nommé PlantGrowth. Il contient le poids des plantes obtenues sous un contrôle et deux conditions de traitement différentes.
Charger et inspecter les données en utilisant la fonction sample_n_by() pour afficher une ligne aléatoire par groupes:
data("PlantGrowth")
set.seed(1234)
PlantGrowth %>% sample_n_by(group, size = 1)## # A tibble: 3 x 2
## weight group
## <dbl> <fct>
## 1 5.58 ctrl
## 2 6.03 trt1
## 3 4.92 trt2Afficher les niveaux de la variable de groupement:
levels(PlantGrowth$group)## [1] "ctrl" "trt1" "trt2"Si les niveaux ne sont pas automatiquement dans le bon ordre, réorganisez-les comme suit:
PlantGrowth <- PlantGrowth %>%
reorder_levels(group, order = c("ctrl", "trt1", "trt2"))L’ANOVA à un facteur peut être utilisée pour déterminer si les moyennes de croissance des plantes sont significativement différentes entre les trois conditions.
Statistiques descriptives
Calculer quelques statistiques sommaires (nombre, moyenne et sd) de la variable weight (poids) organisée par groupes:
PlantGrowth %>%
group_by(group) %>%
get_summary_stats(weight, type = "mean_sd")## # A tibble: 3 x 5
## group variable n mean sd
## <fct> <chr> <dbl> <dbl> <dbl>
## 1 ctrl weight 10 5.03 0.583
## 2 trt1 weight 10 4.66 0.794
## 3 trt2 weight 10 5.53 0.443Visualisation
Créez un boxplot de weight par group:
ggboxplot(PlantGrowth, x = "group", y = "weight")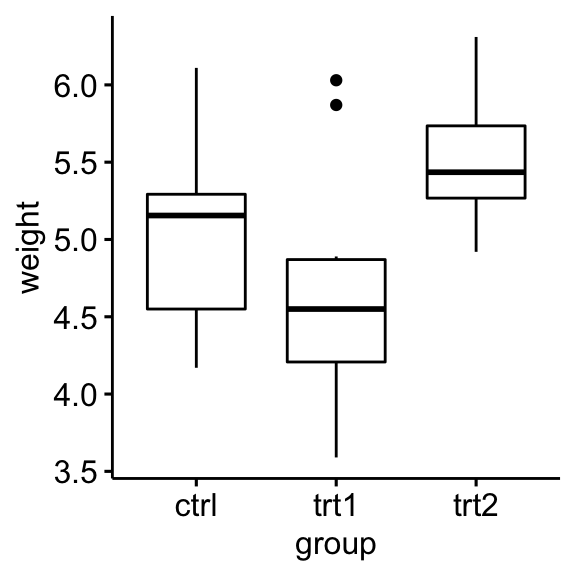
Vérifier les hypothèses
Valeurs aberrantes
Les valeurs aberrantes peuvent être facilement identifiées à l’aide de méthodes des boxplots, implémentées dans la fonction R identify_outliers() [paquet rstatix].
PlantGrowth %>%
group_by(group) %>%
identify_outliers(weight)## # A tibble: 2 x 4
## group weight is.outlier is.extreme
## <fct> <dbl> <lgl> <lgl>
## 1 trt1 5.87 TRUE FALSE
## 2 trt1 6.03 TRUE FALSEIl n’y avait pas de valeurs extrêmes aberrantes.
Notez que, dans le cas où vous avez des valeurs extrêmes aberrantes, cela peut être dû à : 1) erreurs de saisie de données, erreurs de mesure ou valeurs inhabituelles.
Vous pouvez de toute façon inclure la valeur aberrante dans l’analyse si vous ne croyez pas que le résultat sera affecté de façon substantielle. Ceci peut être évalué en comparant le résultat du test ANOVA avec et sans la valeur aberrante.
Il est également possible de conserver les valeurs aberrantes dans les données et d’effectuer un test ANOVA robuste en utilisant le package WRS2.
Hypothèse de normalité
L’hypothèse de normalité peut être vérifiée en utilisant l’une des deux approches suivantes:
- Analyse des résidus du modèle ANOVA pour vérifier la normalité pour tous les groupes ensemble. Cette approche est plus facile et très pratique lorsque vous avez plusieurs groupes ou s’il y a peu de points de données par groupe.
- Vérifier la normalité pour chaque groupe séparément. Cette approche peut être utilisée lorsque vous n’avez que quelques groupes et plusieurs points de données par groupe.
Dans cette section, nous vous montrerons comment procéder pour les options 1 et 2.
Vérifier l’hypothèse de normalité en analysant les résidus du modèle. Le QQ plot et le test de normalité de Shapiro-Wilk sont utilisés. Le graphique QQ plot dessine la corrélation entre une donnée définie et la distribution normale.
# Construire le modèle linéaire
model <- lm(weight ~ group, data = PlantGrowth)
# Créer un QQ plot des résidus
ggqqplot(residuals(model))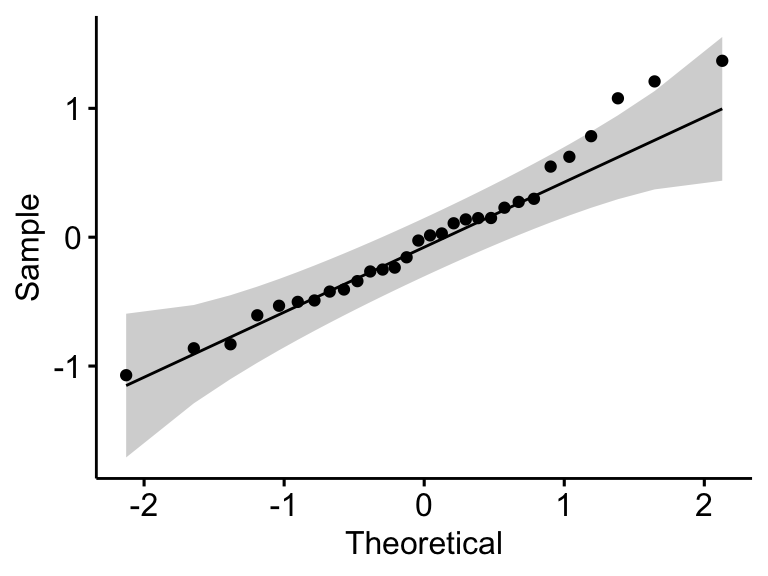
# Calculer le test de normalité de Shapiro-Wilk
shapiro_test(residuals(model))## # A tibble: 1 x 3
## variable statistic p.value
## <chr> <dbl> <dbl>
## 1 residuals(model) 0.966 0.438Dans le QQ plot, comme tous les points se situent approximativement le long de la ligne de référence, nous pouvons supposer une normalité. Cette conclusion est étayée par le test Shapiro-Wilk. La p-value n’est pas significative (p = 0,13), on peut donc supposer une normalité.
Vérifier l’hypothèse de normalité par groupe. Calcul du test Shapiro-Wilk pour chaque niveau de groupe. Si les données sont normalement distribuées, la p-value doit être supérieure à 0,05.
PlantGrowth %>%
group_by(group) %>%
shapiro_test(weight)## # A tibble: 3 x 4
## group variable statistic p
## <fct> <chr> <dbl> <dbl>
## 1 ctrl weight 0.957 0.747
## 2 trt1 weight 0.930 0.452
## 3 trt2 weight 0.941 0.564Le score était normalement distribué (p > 0,05) pour chaque groupe, tel qu’évalué par le test de normalité de Shapiro-Wilk.
Notez que, si la taille de votre échantillon est supérieure à 50, le graphique de normalité QQ plot est préféré parce qu’avec des échantillons de plus grande taille, le test de Shapiro-Wilk devient très sensible même à un écart mineur par rapport à la normale.
Le graphique QQ plot dessine la corrélation entre une donnée définie et la distribution normale. Créer des QQ plots pour chaque niveau de groupe:
ggqqplot(PlantGrowth, "weight", facet.by = "group")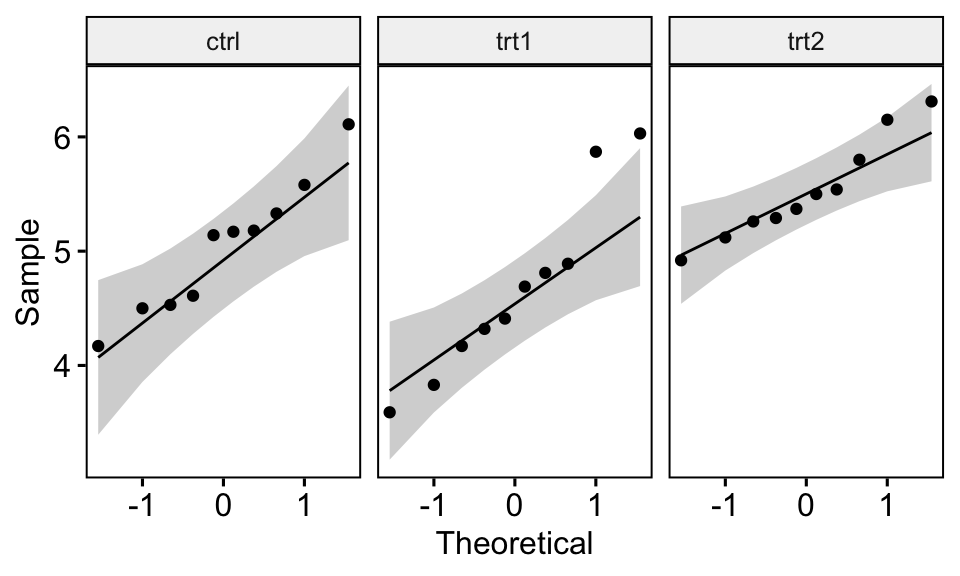
Tous les points se situent approximativement le long de la ligne de référence, pour chaque cellule. Nous pouvons donc supposer la normalité des données.
Si vous avez des doutes sur la normalité des données, vous pouvez utiliser le test Kruskal-Wallis, qui est l’alternative non paramétrique au test ANOVA à un facteur.
L’hypothèse d’homogénéité des variances
- Le graphique residuals versus fits (résidus versus prédictions) peut être utilisé pour vérifier l’homogénéité des variances.
plot(model, 1)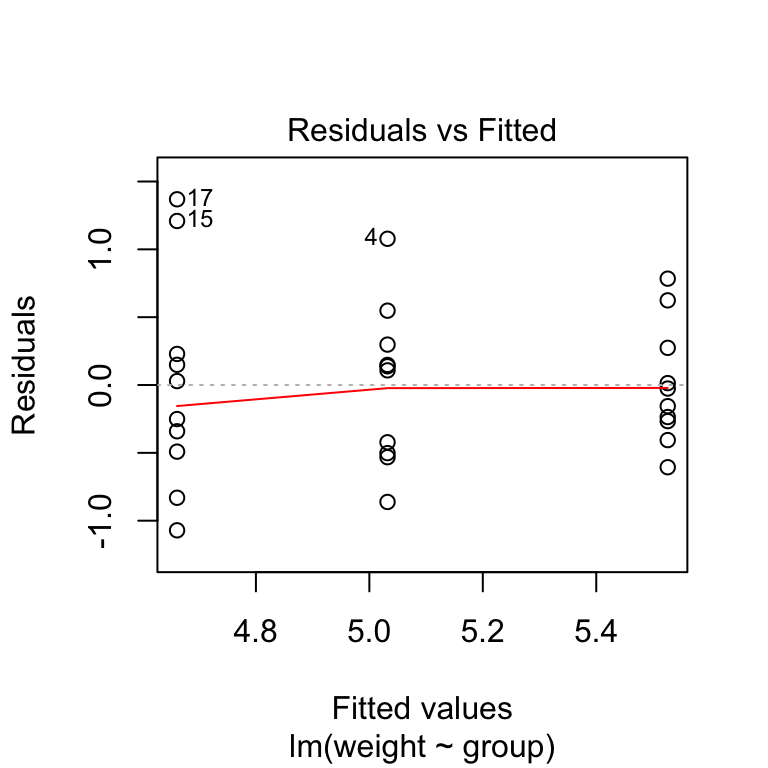
Dans le graphique ci-dessus, il n’y a pas de relations évidentes entre les résidus et les valeurs calculées (fitted) (la moyenne de chaque groupe), ce qui est bon. On peut donc supposer l’homogénéité des variances.
- Il est également possible d’utiliser le test de Levene pour vérifier l’homogénéité des variances:
PlantGrowth %>% levene_test(weight ~ group)## # A tibble: 1 x 4
## df1 df2 statistic p
## <int> <int> <dbl> <dbl>
## 1 2 27 1.12 0.341Du résultat ci-dessus, nous pouvons voir que la p-value est > 0,05, ce qui n’est pas significatif. Cela signifie qu’il n’y a pas de différence significative entre les variances d’un groupe à l’autre. Par conséquent, nous pouvons supposer l’homogénéité des variances dans les différents groupes de traitement.
Dans une situation où l’hypothèse d’homogénéité de la variances n’est pas satisfaite, vous pouvez calculer le test ANOVA de Welch à un facteur en utilisant la fonction welch_anova_test() [rstatix package]. Ce test n’exige pas l’hypothèse de variances égales.
Calculs
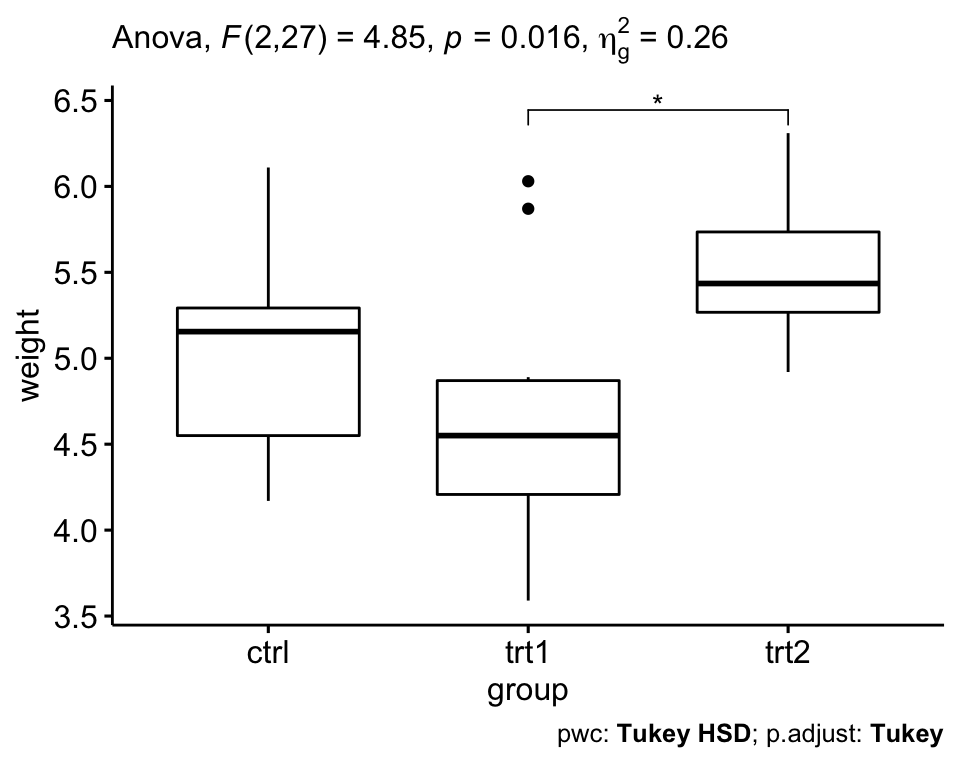
res.aov <- PlantGrowth %>% anova_test(weight ~ group)
res.aov## ANOVA Table (type II tests)
##
## Effect DFn DFd F p p<.05 ges
## 1 group 2 27 4.85 0.016 * 0.264Dans le tableau ci-dessus, la colonne ges correspond l’eta-carré généralisé (taille de l’effet). Il mesure la proportion de la variabilité de la variable-réponse (ici weight pour poids des plantes) qui peut être expliquée par le prédicteur (ici group pour groupe de traitement). Une valeur de l’effet de 0,26 (26 %) signifie que 26 % de la variation du poids (weight) peut être attribuable aux conditions de traitement.
Le tableau ANOVA ci-dessus montre qu’il existe des différences significatives entre les groupes (p = 0,016), qui sont mis en évidence par “*“, F(2, 27) = 4,85, p = 0,16, eta2[g] = 0,26.
là où,
Findique que nous comparons à une distribution F (test F) ;(2, 27)indique les degrés de liberté du numérateur (DFn) et du dénominateur (DFd), respectivement ;4.85indique la valeur statistique F obtenuepspécifie la p-valuegesest la taille de l’effet généralisé (taille de la variabilité due au facteur)
Tests post-hoc
Une ANOVA, à un facteur, significative est généralement suivie de tests post-hoc de Tukey pour effectuer de multiples comparaisons par paires entre les groupes. Fonction R clé: tukey_hsd() [rstatix].
# Comparaisons par paires
pwc <- PlantGrowth %>% tukey_hsd(weight ~ group)
pwc## # A tibble: 3 x 8
## term group1 group2 estimate conf.low conf.high p.adj p.adj.signif
## * <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 group ctrl trt1 -0.371 -1.06 0.320 0.391 ns
## 2 group ctrl trt2 0.494 -0.197 1.19 0.198 ns
## 3 group trt1 trt2 0.865 0.174 1.56 0.012 *Le résultat contient les colonnes suivantes:
estimate: estimation de la différence entre les moyennes des deux groupesconf.low,conf.high: les bornes inférieure et supérieure de l’intervalle de confiance à 95 % (par défaut)p.adj: p-value après ajustement pour les comparaisons multiples.
Il ressort du résultat que seule la différence entre trt2 et trt1 est significative (p-value ajustée = 0,012).
Rapporter
Nous pourrions présenter les résultats de l’analyse de variance à un facteur comme suit:
L’ANOVA à un facteur a été réalisée pour évaluer si la croissance des plantes était différente pour les 3 groupes de traitement différents : ctr (n = 10), trt1 (n = 10) et trt2 (n = 10).
Les données sont présentées sous forme de moyenne +/- écart type. La croissance des plantes était statistiquement significativement différente entre les différents groupes de traitement, F(2, 27) = 4,85, p = 0,016, eta-carré généralisé = 0,26.
La croissance des plantes a diminué dans le groupe trt1 (4,66 +/- 0,79) par rapport au groupe ctr (5,03 +/- 0,58). Il a augmenté dans le groupe trt2 (5,53 +/- 0,44) par rapport au groupe trt1 et ctr.
Les analyses post-hoc de Tukey ont révélé que l’augmentation de trt1 à trt2 (0,87, IC à 95 % (0,17 à 1,56)) était statistiquement significative (p = 0,012), mais qu’aucune autres différences inter-groupes n’était statistiquement significative.
# Visualisation : Boxplots avec p-values
pwc <- pwc %>% add_xy_position(x = "group")
ggboxplot(PlantGrowth, x = "group", y = "weight") +
stat_pvalue_manual(pwc, hide.ns = TRUE) +
labs(
subtitle = get_test_label(res.aov, detailed = TRUE),
caption = get_pwc_label(pwc)
)
Relaxer l’hypothèse d’homogénéité de la variance
Le test classique d’ANOVA à un facteur nécessite l’hypothèse d’égalité des variances pour tous les groupes. Dans notre exemple, l’hypothèse d’homogénéité de la variance s’est révélée bonne : le test de Levene n’est pas significatif.
Comment sauver notre test ANOVA, dans une situation où l’hypothèse d’homogénéité de la variance est violée ?
- Le test ANOVA de Welch à 1 facteur est une alternative à l’ANOVA standard, à 1 facteur, dans les situations où l’homogénéité de la variance ne peut être supposée (c.-à-d. le test de Levene est significatif).
- Dans ce cas, le test post-hoc de Games-Howell ou le test t par paires (sans l’hypothèse de variances égales) peut être utilisé pour comparer toutes les combinaisons possibles de différences de groupes.
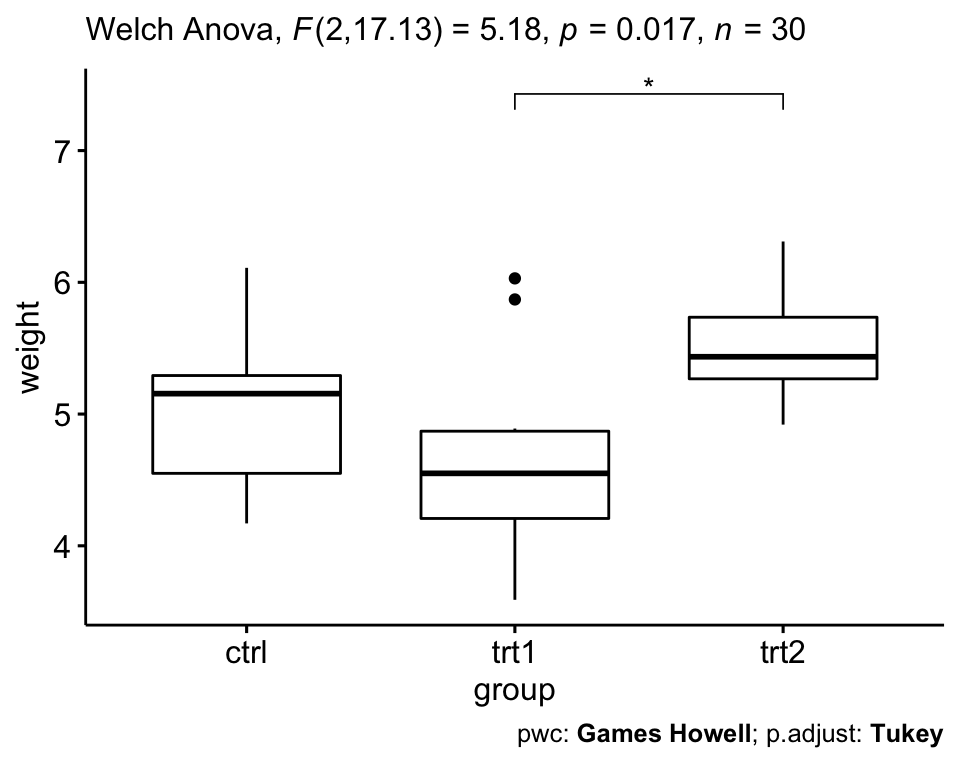
# Test ANOVA de Welch à un facteur
res.aov2 <- PlantGrowth %>% welch_anova_test(weight ~ group)
# Comparaisons par paires (Games-Howell)
pwc2 <- PlantGrowth %>% games_howell_test(weight ~ group)
# Visualisation : Boxplots avec p-values
pwc2 <- pwc2 %>% add_xy_position(x = "group", step.increase = 1)
ggboxplot(PlantGrowth, x = "group", y = "weight") +
stat_pvalue_manual(pwc2, hide.ns = TRUE) +
labs(
subtitle = get_test_label(res.aov2, detailed = TRUE),
caption = get_pwc_label(pwc2)
)
Vous pouvez également effectuer des comparaisons par paires à l’aide du test t sans hypothèse d’égalité des variances:
pwc3 <- PlantGrowth %>%
pairwise_t_test(
weight ~ group, pool.sd = FALSE,
p.adjust.method = "bonferroni"
)
pwc3ANOVA à deux facteurs
Préparation des données
Nous utiliserons le jeu de données sur la satisfaction au travail [package datarium], qui contient le score de satisfaction au travail organisé selon le sexe et le niveau de scolarité.
Dans cette étude, une recherche vise à évaluer s’il existe une interaction significative entre le sexe (gender) et le niveau d’instruction (education_level) pour expliquer le score de satisfaction au travail. Un effet d’interaction se produit lorsque l’effet d’une variable indépendante sur une variable-réponse dépend du niveau des autres variables indépendantes. S’il n’existe pas d’effet d’interaction, les effets principaux pourraient être rapportés.
Charger les données et inspecter une ligne aléatoire par groupes:
set.seed(123)
data("jobsatisfaction", package = "datarium")
jobsatisfaction %>% sample_n_by(gender, education_level, size = 1)## # A tibble: 6 x 4
## id gender education_level score
## <fct> <fct> <fct> <dbl>
## 1 3 male school 5.07
## 2 17 male college 6.3
## 3 23 male university 10
## 4 37 female school 5.51
## 5 48 female college 5.65
## 6 49 female university 8.26Dans cet exemple, l’effet de “education_level” est notre variable focale, c’est-à-dire notre première cible. On pense que l’effet de “education_level” (niveau d’éducation) dépendra d’un autre facteur, “gender” (sexe), que l’on appelle variable modératrice.
Statistiques descriptives
Calculer la moyenne et l’écart-type (écart-type) de score par groupe:
jobsatisfaction %>%
group_by(gender, education_level) %>%
get_summary_stats(score, type = "mean_sd")## # A tibble: 6 x 6
## gender education_level variable n mean sd
## <fct> <fct> <chr> <dbl> <dbl> <dbl>
## 1 male school score 9 5.43 0.364
## 2 male college score 9 6.22 0.34
## 3 male university score 10 9.29 0.445
## 4 female school score 10 5.74 0.474
## 5 female college score 10 6.46 0.475
## 6 female university score 10 8.41 0.938Visualisation
Créez un boxplot du score par niveau de genre, coloré par niveau d’éducation:
bxp <- ggboxplot(
jobsatisfaction, x = "gender", y = "score",
color = "education_level", palette = "jco"
)
bxp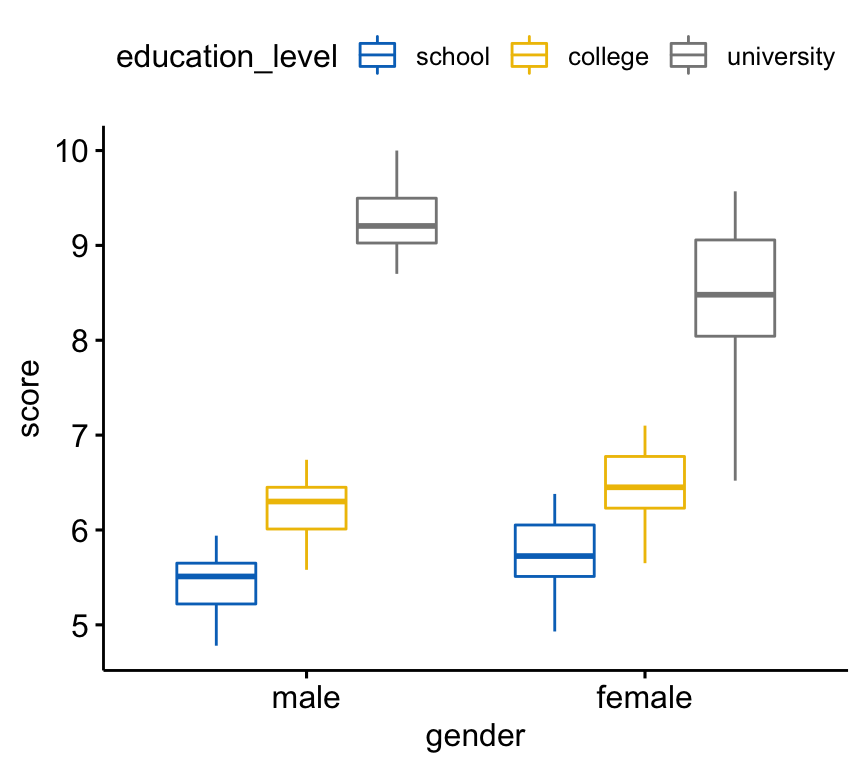
Vérifier les hypothèses
Valeurs aberrantes
Identifier les valeurs aberrantes dans chaque cellule du plan:
jobsatisfaction %>%
group_by(gender, education_level) %>%
identify_outliers(score)Il n’y avait pas de valeurs extrêmes aberrantes.
Hypothèse de normalité
Vérifier l’hypothèse de normalité en analysant les résidus du modèle. Le QQ plot et le test de normalité de Shapiro-Wilk sont utilisés.
# Construire le modèle linéaire
model <- lm(score ~ gender*education_level,
data = jobsatisfaction)
# Créer un QQ plot des résidus
ggqqplot(residuals(model))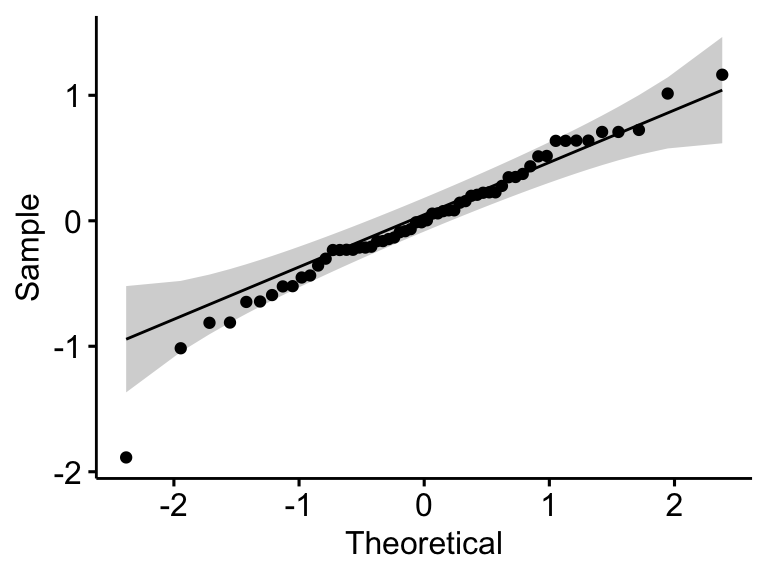
# Calculer le test de normalité de Shapiro-Wilk
shapiro_test(residuals(model))## # A tibble: 1 x 3
## variable statistic p.value
## <chr> <dbl> <dbl>
## 1 residuals(model) 0.968 0.127Dans le QQ plot, comme tous les points se situent approximativement le long de la ligne de référence, nous pouvons supposer une normalité. Cette conclusion est étayée par le test Shapiro-Wilk. La p-value n’est pas significative (p = 0,13), on peut donc supposer une normalité.
Vérifier l’hypothèse de normalité par groupe. Calcul du test de Shapiro-Wilk pour chaque combinaison de niveaux des facteurs:
jobsatisfaction %>%
group_by(gender, education_level) %>%
shapiro_test(score)## # A tibble: 6 x 5
## gender education_level variable statistic p
## <fct> <fct> <chr> <dbl> <dbl>
## 1 male school score 0.980 0.966
## 2 male college score 0.958 0.779
## 3 male university score 0.916 0.323
## 4 female school score 0.963 0.819
## 5 female college score 0.963 0.819
## 6 female university score 0.950 0.674Le score était normalement distribué (p > 0,05) pour chaque cellule, tel qu’évalué par le test de normalité de Shapiro-Wilk.
Créer des graphiques QQ plots pour chaque cellule du plan:
ggqqplot(jobsatisfaction, "score", ggtheme = theme_bw()) +
facet_grid(gender ~ education_level)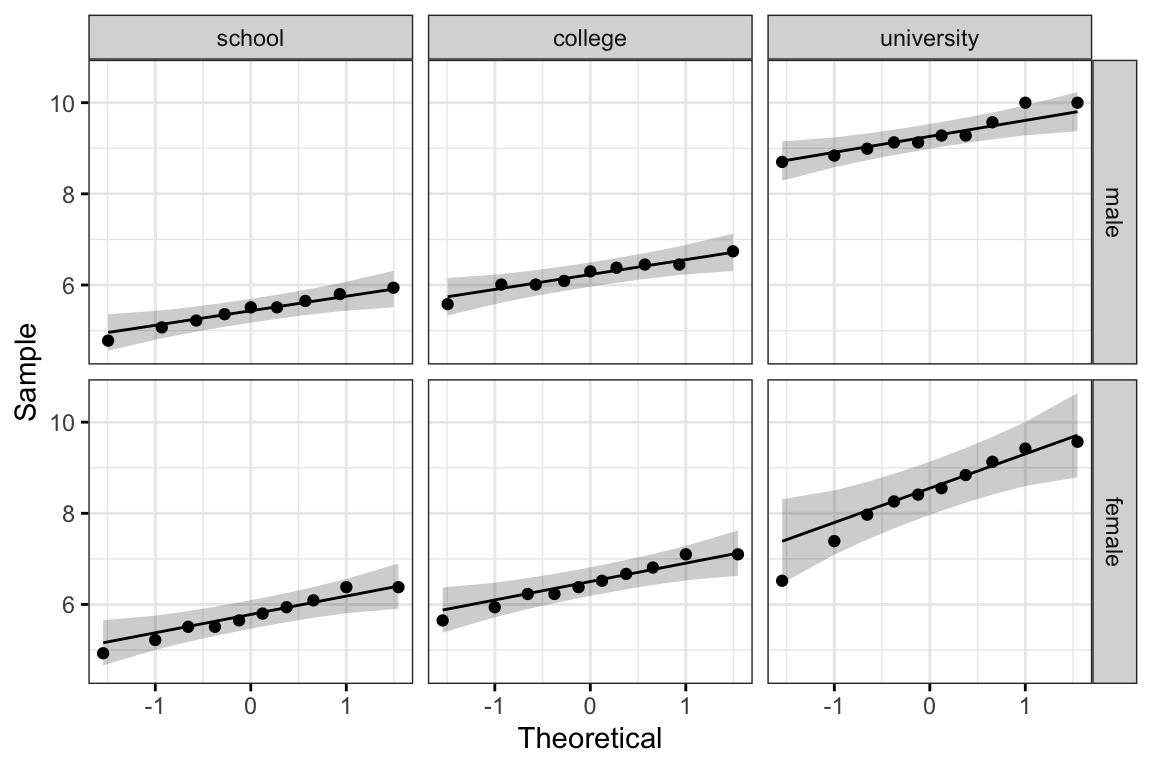
Tous les points se situent approximativement le long de la ligne de référence, pour chaque cellule. Nous pouvons donc supposer la normalité des données.
L’hypothèse d’homogénéité des variances
Ceci peut être vérifié à l’aide du test de Levene:
jobsatisfaction %>% levene_test(score ~ gender*education_level)## # A tibble: 1 x 4
## df1 df2 statistic p
## <int> <int> <dbl> <dbl>
## 1 5 52 2.20 0.0686Le test de Levene n’est pas significatif (p > 0,05). Par conséquent, nous pouvons supposer l’homogénéité des variances dans les différents groupes.
Calculs
Dans le code R ci-dessous, l’astérisque représente l’effet d’interaction et l’effet principal de chaque variable (et de toutes les interactions d’ordre inférieur).
res.aov <- jobsatisfaction %>% anova_test(score ~ gender * education_level)
res.aov## ANOVA Table (type II tests)
##
## Effect DFn DFd F p p<.05 ges
## 1 gender 1 52 0.745 3.92e-01 0.014
## 2 education_level 2 52 187.892 1.60e-24 * 0.878
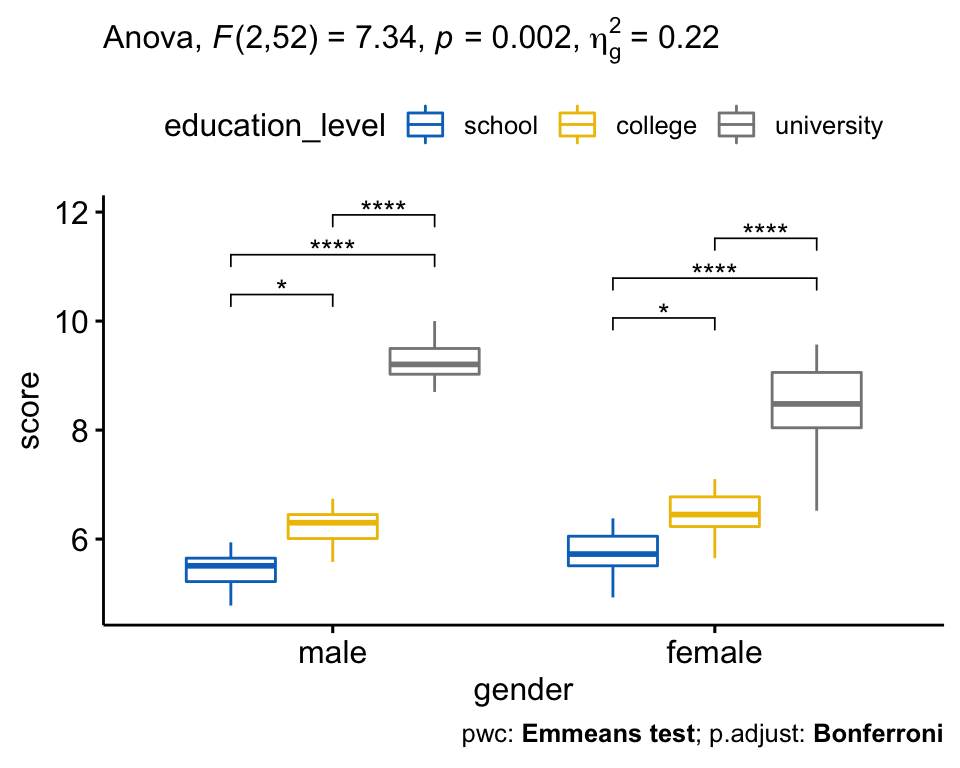
## 3 gender:education_level 2 52 7.338 2.00e-03 * 0.220Il y avait une interaction statistiquement significative entre le sexe et le niveau de scolarité pour le score de satisfaction au travail, F(2, 52) = 7,34, p = 0,002.
Tests post-hoc
Une interaction, à 2 facteurs, significative indique que l’impact d’un facteur (p. ex., le niveau de scolarité, education_level) sur la variable-réponse (p. ex., le score de satisfaction professionnelle) dépend du niveau de l’autre facteur (p. ex., sexe) (et vice versa). Ainsi, vous pouvez décomposer une interaction significative, à deux facteurs, en:
- Effet principal simple : exécuter un modèle à un facteur de la première variable à chaque niveau de la deuxième variable,
- Comparaisons par paires : si l’effet principal est significatif, effectuez plusieurs comparaisons par paires pour déterminer quels groupes sont différents.
Dans le cas d’une interaction à deux facteurs non significative, vous devez déterminer si vous avez des effets principaux statistiquement significatifs dans le résultat de l’ANOVA. Un effet principal significatif peut être suivi par des comparaisons par paires entre les groupes.
Procédure pour une interaction significative à deux facteurs
Calculer des effets principaux
Dans notre exemple, vous pourriez donc étudier l’effet de education_level à chaque niveau de gender ou étudier l’effet de gender à chaque niveau de la variable education_level.
Ici, nous allons exécuter une ANOVA à un facteur de education_level à chaque niveau de gender.
Il est à noter que, si vous avez satisfait aux hypothèses de l’ANOVA à deux facteurs (p. ex. homogénéité des variances), il est préférable d’utiliser “l’erreur globale” (provenant de l’ANOVA à deux facteurs) comme entrée dans le modèle ANOVA à un facteur. Il sera ainsi plus facile de déceler toute différence statistiquement significative s’il en existe (Keppel et Wickens, 2004 ; Maxwell et Delaney, 2004).
Lorsque l’hypothèse d’homogénéité des variances n’est pas satisfaite, vous pourriez envisager d’exécuter des ANOVA à un facteur utilisant des termes d’erreur distincts.
Dans le code R ci-dessous, nous allons regrouper les données par sexe et analyser les effets principaux simples du niveau de scolarité sur le score de satisfaction au travail. L’argument error est utilisé pour spécifier le modèle ANOVA à partir duquel l’erreur globale et les degrés de liberté doivent être calculés.
# Regrouper les données par sexe et calculer l'anova
model <- lm(score ~ gender * education_level, data = jobsatisfaction)
jobsatisfaction %>%
group_by(gender) %>%
anova_test(score ~ education_level, error = model)## # A tibble: 2 x 8
## gender Effect DFn DFd F p `p<.05` ges
## <fct> <chr> <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 male education_level 2 52 132. 3.92e-21 * 0.836
## 2 female education_level 2 52 62.8 1.35e-14 * 0.707L’effet principal simple de education_level (niveau_de_scolarité) sur le score de satisfaction au travail était statistiquement significatif tant pour les hommes que pour les femmes (p < 0,0001).
En d’autres termes, il y a une différence statistiquement significative dans le score moyen de satisfaction au travail entre hommes de niveau scolaire, collégial ou universitaire, F(2, 52) = 132, p < 0.0001. La même conclusion s’applique aux femmes, F(2, 52) = 62,8, p < 0,0001.
Il est à noter que la significativité statistique des analyses simples des effets principaux a été acceptée à un niveau alpha ajusté de Bonferroni de 0,025. Cela correspond au niveau actuel auquel vous déclarez une signification statistique (c.-à-d. p < 0,05) divisé par le nombre d’effets principaux simples que vous calculez (c.-à-d. 2).
Calculer des comparaisons par paires
Un effet principal statistiquement significatif peut être suivi de multiples comparaisons par paires pour déterminer quelles moyennes de groupe sont différentes. Nous allons maintenant effectuer de multiples comparaisons par paires entre les différents groupes de niveau d’éducation (education_level) par gender.
Vous pouvez exécuter et interpréter toutes les comparaisons par paires possibles en utilisant l’ajustement de Bonferroni. Ceci peut être fait facilement en utilisant la fonction emmeans_test() [paquet rstatix], un wrapper autour du paquet emmeans, qui doit être installé. Emmeans (estimated marginal means en anglais) signifie moyennes marginales estimées (c’est-à-dire moyennes des moindres carrés ou moyennes ajustées).
Comparez le score des différents niveaux d’éducation par niveau de gender (genre):
# comparaisons par paires
library(emmeans)
pwc <- jobsatisfaction %>%
group_by(gender) %>%
emmeans_test(score ~ education_level, p.adjust.method = "bonferroni")
pwc## # A tibble: 6 x 9
## gender .y. group1 group2 df statistic p p.adj p.adj.signif
## * <fct> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 male score school college 52 -3.07 3.37e- 3 1.01e- 2 *
## 2 male score school university 52 -15.3 6.87e-21 2.06e-20 ****
## 3 male score college university 52 -12.1 8.42e-17 2.53e-16 ****
## 4 female score school college 52 -2.94 4.95e- 3 1.49e- 2 *
## 5 female score school university 52 -10.8 6.07e-15 1.82e-14 ****
## 6 female score college university 52 -7.90 1.84e-10 5.52e-10 ****Il y avait une différence significative du score de satisfaction professionnelle entre tous les groupes pour les hommes et les femmes (p < 0,05).
Procédure pour une interaction non significative à deux facteurs
Inspecter les effets principaux
Si l’interaction à deux facteurs n’est pas statistiquement significative, vous devez consulter l’effet principal pour chacune des deux variables [gender (sexe) et education_level (niveau d’éducation)] dans le résultat de l’ANOVA.
res.aov## ANOVA Table (type II tests)
##
## Effect DFn DFd F p p<.05 ges
## 1 gender 1 52 0.745 3.92e-01 0.014
## 2 education_level 2 52 187.892 1.60e-24 * 0.878
## 3 gender:education_level 2 52 7.338 2.00e-03 * 0.220Dans notre exemple, il y avait un effet principal statistiquement significatif du niveau de scolarité (F(2, 52) = 187,89, p < 0,0001) sur le score de satisfaction professionnelle. Cependant, l’effet principal du genre n’était pas significatif, F (1, 52) = 0,74, p = 0,39.
Calculer des comparaisons par paires
Effectuer des comparaisons par paires entre les groupes de niveau d’éducation pour déterminer quels groupes sont significativement différents. L’ajustement de Bonferroni est appliqué. Cette analyse peut être faite en utilisant simplement la fonction de base R pairwise_t_test() ou en utilisant la fonction emmeans_test().
- Test t par paire:
jobsatisfaction %>%
pairwise_t_test(
score ~ education_level,
p.adjust.method = "bonferroni"
)Toutes les différences par paires étaient statistiquement significatives (p < 0,05).
- Comparaisons par paires à l’aide du test Emmeans. Vous devez spécifier le modèle global à partir duquel les degrés de liberté globaux doivent être calculés. Il sera ainsi plus facile de déceler toute différence statistiquement significative, si elle existe.
model <- lm(score ~ gender * education_level, data = jobsatisfaction)
jobsatisfaction %>%
emmeans_test(
score ~ education_level, p.adjust.method = "bonferroni",
model = model
)Rapporter
Une ANOVA à deux facteurs a été menée pour examiner les effets du sexe et du niveau des études sur le score de satisfaction professionnelle.
L’analyse des résidus a été effectuée pour vérifier les hypothèses de l’ANOVA à deux facteurs. Les valeurs aberrantes ont été déterminées à l’aide de la méthode des Box plots, la normalité a été déterminée à l’aide du test de normalité de Shapiro-Wilk et l’homogénéité des variances a été déterminée par le test de Levene.
Il n’y avait pas de valeurs extrêmes aberrantes, les résidus étaient normalement distribués (p > 0,05) et les variances étaient homogènes (p > 0,05).
Il y avait une interaction statistiquement significative entre le sexe et le niveau des études sur le score de satisfaction au travail, F(2, 52) = 7.33, p = 0.0016, eta2[g] = 0.22.
Par conséquent, une analyse des effets principaux pour le niveau d’éducation a été effectuée avec une signification statistique recevant un ajustement de Bonferroni. Il y avait une différence statistiquement significative dans les scores moyens de “satisfaction au travail” pour les hommes (F(2, 52) = 132, p < 0,0001) et les femmes (F(2, 52) = 62,8, p < 0,0001) ayant un niveau scolaire, collégial ou universitaire.
Toutes les comparaisons par paires ont été analysées entre les différents education_level groups organized by gender. Il y avait une différence significative du score de satisfaction au travail entre tous les groupes pour à la fois les hommes et les femmes (p < 0,05).
# Visualisation : Boxplots avec p-values
pwc <- pwc %>% add_xy_position(x = "gender")
bxp +
stat_pvalue_manual(pwc) +
labs(
subtitle = get_test_label(res.aov, detailed = TRUE),
caption = get_pwc_label(pwc)
)
ANOVA à trois facteurs
L’ANOVA à trois facteurs est une extension de l’analyse de variance à deux facteurs pour évaluer s’il y a un effet d’interaction entre trois variables catégorielles indépendantes sur une variable-réponse continue.
Préparation des données
Nous utiliserons le jeu de données headache [package datarium], qui contient les mesures du score de douleur d’épisode migraineux chez 72 participants traités avec trois traitements différents. Les participants incluent 36 hommes et 36 femmes. Les hommes et les femmes ont été subdivisés selon qu’ils présentaient un risque faible ou élevé de migraine.
Nous voulons comprendre comment chaque variable indépendante (type de traitement, risque de migraine et sexe) interagit pour prédire le score de douleur.
Charger les données et inspecter une ligne aléatoire par combinaison de groupes:
set.seed(123)
data("headache", package = "datarium")
headache %>% sample_n_by(gender, risk, treatment, size = 1)## # A tibble: 12 x 5
## id gender risk treatment pain_score
## <int> <fct> <fct> <fct> <dbl>
## 1 20 male high X 100
## 2 29 male high Y 91.2
## 3 33 male high Z 81.3
## 4 6 male low X 73.1
## 5 12 male low Y 67.9
## 6 13 male low Z 75.0
## # … with 6 more rowsDans cet exemple, l’effet des types de traitement est notre variable focale, c’est-à-dire notre principale cible. On pense que l’effet des traitements dépendra de deux autres facteurs, le sexe et le niveau de risque de migraine, qui sont appelés variables modératrices.
Statistiques descriptives
Calculer la moyenne et l’écart-type (SD) de pain_score ( score_de_la_douleur) par groupe:
headache %>%
group_by(gender, risk, treatment) %>%
get_summary_stats(pain_score, type = "mean_sd")## # A tibble: 12 x 7
## gender risk treatment variable n mean sd
## <fct> <fct> <fct> <chr> <dbl> <dbl> <dbl>
## 1 male high X pain_score 6 92.7 5.12
## 2 male high Y pain_score 6 82.3 5.00
## 3 male high Z pain_score 6 79.7 4.05
## 4 male low X pain_score 6 76.1 3.86
## 5 male low Y pain_score 6 73.1 4.76
## 6 male low Z pain_score 6 74.5 4.89
## # … with 6 more rowsVisualisation
Créez un boxplot de pain_score (score_douleur) par treatment (traitement), colorez les traits par risk (groupes de risque) et faire un facet du graphique par gender (sexe):
bxp <- ggboxplot(
headache, x = "treatment", y = "pain_score",
color = "risk", palette = "jco", facet.by = "gender"
)
bxp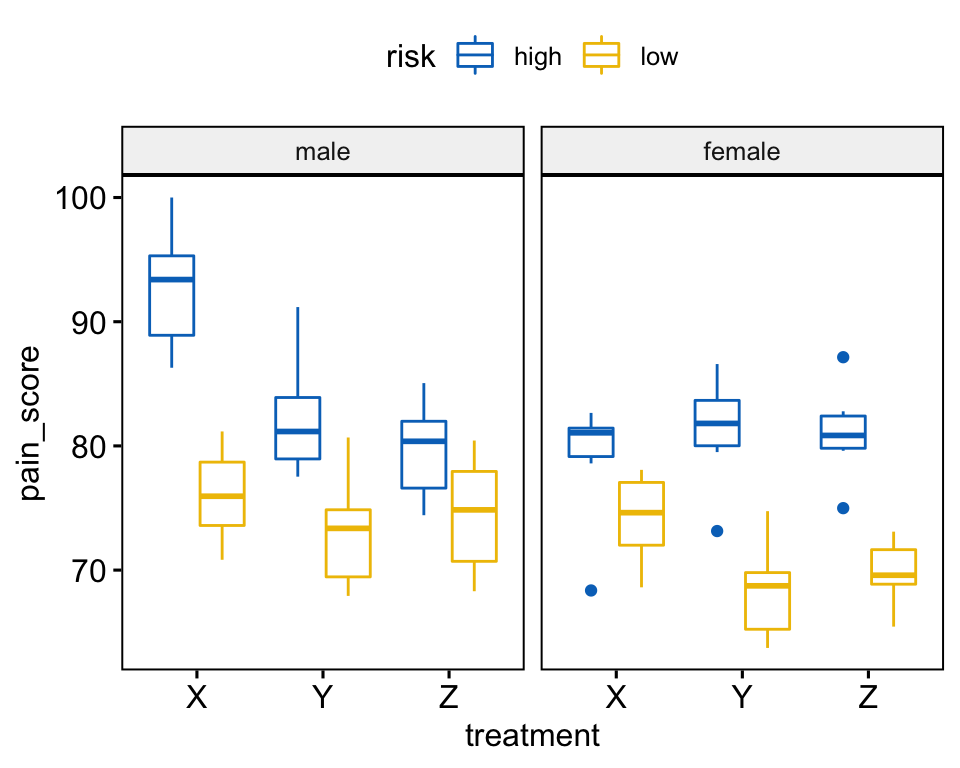
Vérifier les hypothèses
Valeurs aberrantes
Identifier les valeurs aberrantes par groupe:
headache %>%
group_by(gender, risk, treatment) %>%
identify_outliers(pain_score)## # A tibble: 4 x 7
## gender risk treatment id pain_score is.outlier is.extreme
## <fct> <fct> <fct> <int> <dbl> <lgl> <lgl>
## 1 female high X 57 68.4 TRUE TRUE
## 2 female high Y 62 73.1 TRUE FALSE
## 3 female high Z 67 75.0 TRUE FALSE
## 4 female high Z 71 87.1 TRUE FALSEOn peut voir que les données contiennent une valeur aberrante extrême (id = 57, femme à risque élevé de migraine prenant le médicament X)
Les valeurs aberrantes peuvent être dues à : 1) erreurs de saisie des données, 2) erreurs de mesure ou 3) valeurs inhabituelles.
Vous pouvez de toute façon inclure la valeur aberrante dans l’analyse si vous ne croyez pas que le résultat sera affecté de façon substantielle. Ceci peut être évalué en comparant le résultat du test ANOVA avec et sans la valeur aberrante.
Il est également possible de conserver les valeurs aberrantes dans les données et d’effectuer un test ANOVA robuste en utilisant le package WRS2.
Hypothèse de normalité
Vérifier l’hypothèse de normalité en analysant les résidus du modèle. Le QQ plot et le test de normalité de Shapiro-Wilk sont utilisés.
model <- lm(pain_score ~ gender*risk*treatment, data = headache)
# Créer un QQ plot des résidus
ggqqplot(residuals(model))
# Calculer le test de normalité de Shapiro-Wilk
shapiro_test(residuals(model))## # A tibble: 1 x 3
## variable statistic p.value
## <chr> <dbl> <dbl>
## 1 residuals(model) 0.982 0.398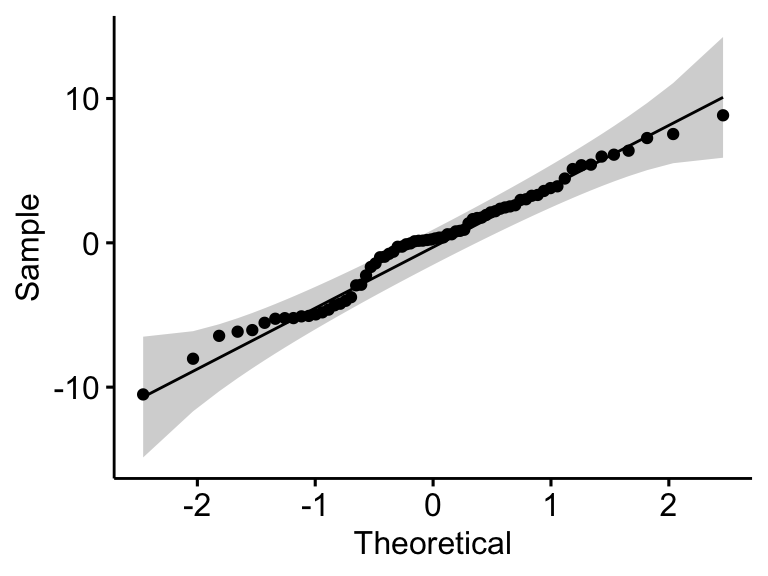
Dans le QQ plot, comme tous les points se situent approximativement le long de la ligne de référence, nous pouvons supposer une normalité. Cette conclusion est étayée par le test Shapiro-Wilk. La p-value n’est pas significative (p = 0,4), on peut donc supposer une normalité.
Vérifier l’hypothèse de normalité par groupe. Calcul du test de Shapiro-Wilk pour chaque combinaison de niveaux des facteurs.
headache %>%
group_by(gender, risk, treatment) %>%
shapiro_test(pain_score)## # A tibble: 12 x 6
## gender risk treatment variable statistic p
## <fct> <fct> <fct> <chr> <dbl> <dbl>
## 1 male high X pain_score 0.958 0.808
## 2 male high Y pain_score 0.902 0.384
## 3 male high Z pain_score 0.955 0.784
## 4 male low X pain_score 0.982 0.962
## 5 male low Y pain_score 0.920 0.507
## 6 male low Z pain_score 0.924 0.535
## # … with 6 more rowsLes scores de douleur étaient normalement distribués (p > 0,05) sauf pour un groupe (femme à risque élevé de migraine prenant le médicament X, p = 0,0086), tel qu’évalué par le test de normalité de Shapiro-Wilk.
Créer un QQ plot pour chaque cellule du plan:
ggqqplot(headache, "pain_score", ggtheme = theme_bw()) +
facet_grid(gender + risk ~ treatment, labeller = "label_both")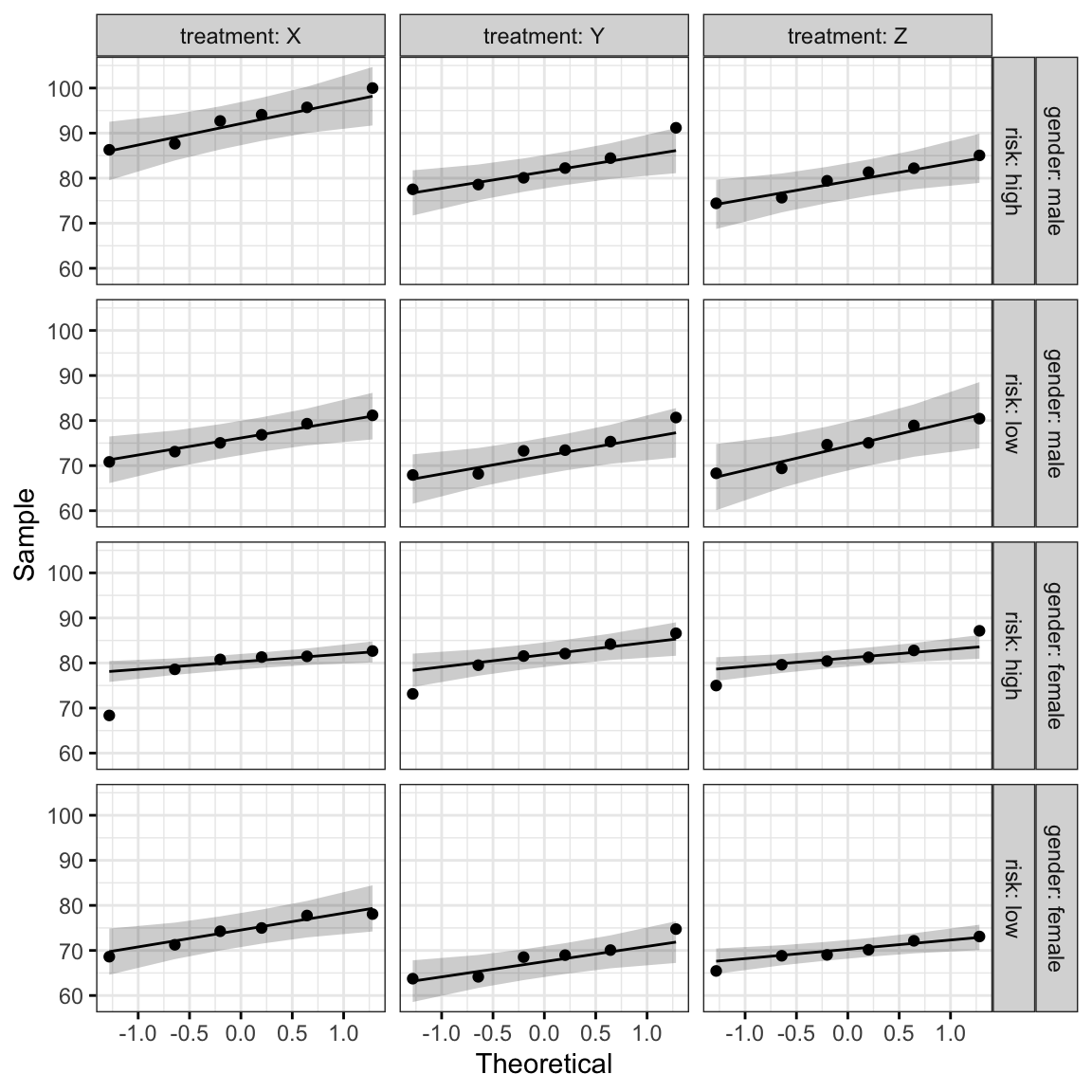
Tous les points se situent approximativement le long de la ligne de référence, à l’exception d’un groupe (femmes à risque élevé de migraine prenant le médicament X), où nous avons déjà identifié une valeur aberrante extrême.
L’hypothèse d’homogénéité des variances
Ceci peut être vérifié à l’aide du test de Levene:
headache %>% levene_test(pain_score ~ gender*risk*treatment)## # A tibble: 1 x 4
## df1 df2 statistic p
## <int> <int> <dbl> <dbl>
## 1 11 60 0.179 0.998Le test de Levene n’est pas significatif (p > 0,05). Par conséquent, nous pouvons supposer l’homogénéité des variances dans les différents groupes.
Calculs
res.aov <- headache %>% anova_test(pain_score ~ gender*risk*treatment)
res.aov## ANOVA Table (type II tests)
##
## Effect DFn DFd F p p<.05 ges
## 1 gender 1 60 16.196 1.63e-04 * 0.213
## 2 risk 1 60 92.699 8.80e-14 * 0.607
## 3 treatment 2 60 7.318 1.00e-03 * 0.196
## 4 gender:risk 1 60 0.141 7.08e-01 0.002
## 5 gender:treatment 2 60 3.338 4.20e-02 * 0.100
## 6 risk:treatment 2 60 0.713 4.94e-01 0.023
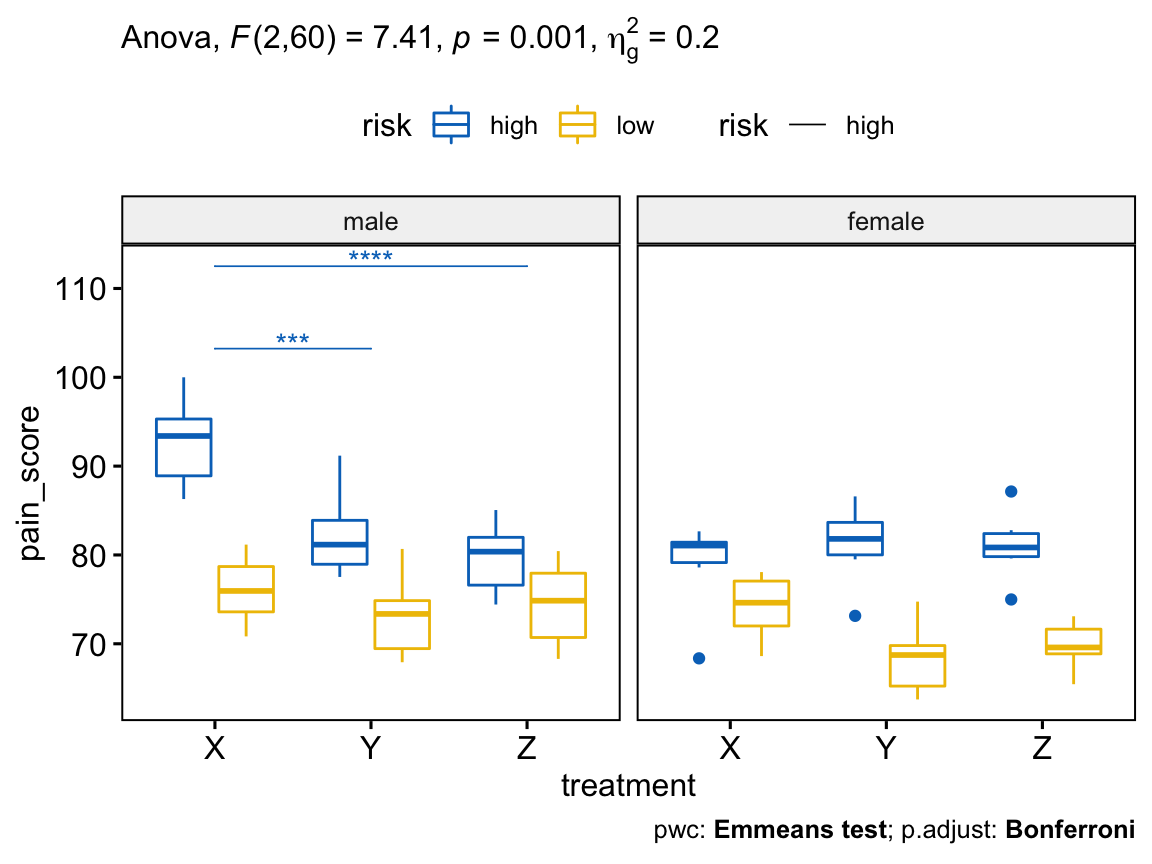
## 7 gender:risk:treatment 2 60 7.406 1.00e-03 * 0.198Il y avait une interaction, à trois facteurs, statistiquement significative entre le sexe, le risque et le traitement, F(2, 60) = 7,41, p = 0,001.
Tests post-hoc
S’il y a un effet significatif d’interaction à trois facteurs, vous pouvez le décomposer en:
- Interaction à deux facteurs : exécuter l’interaction, à deux facteurs, à chaque niveau de la troisième variable,
- Effet principal : exécuter un modèle, à un facteur, à chaque niveau de la deuxième variable, et
- Comparaisons par paires : effectuer des comparaisons par paires ou d’autres comparaisons post-hoc si nécessaire.
Si vous n’avez pas d’interaction à trois facteurs statistiquement significative, vous devez déterminer si vous avez une interaction à deux facteurs statistiquement significative à partir du résultat de l’ANOVA. Vous pouvez suivre une interaction significative à deux facteurs par des analyses simples des effets principaux et des comparaisons par paires entre les groupes si nécessaire.
Dans cette section, nous décrirons la procédure à suivre pour une interaction significative à trois facteurs.
Calculer des interactions à deux facteurs
Vous êtes libre de décider des deux variables qui formeront les interactions à deux facteurs et quelle variable agira comme troisième variable (modératrice). Dans notre exemple, nous voulons évaluer l’effet de l’interaction risque*traitement sur pain_score (score de douleur) à chaque niveau du genre (gender).
Notez que, lors de l’analyse d’interaction à deux facteurs, il est préférable d’utiliser le terme “erreur globale” (ou résidus) du résultat d’ANOVA à trois facteurs, obtenu précédemment en utilisant l’ensemble des données. Cela est particulièrement recommandé lorsque l’hypothèse d’homogénéité des variances est respectée (Keppel et Wickens, 2004).
L’utilisation de l’erreur spécifique au groupe est “plus prudent” contre toute violation des hypothèses. Toutefois, le terme “erreur global” a plus de puissance - en particulier pour les petits échantillons - mais sont susceptibles de poser des problèmes s’il y a des violations des hypothèses.
Dans le code R ci-dessous, nous allons regrouper les données par sexe et calculer l’interaction à deux facteurs treatment*risk. L’argument error est utilisé pour spécifier le modèle ANOVA à trois facteurs à partir duquel l’erreur globale et les degrés de liberté doivent être calculés.
# Regrouper les données selon le sexe et l'âge
# calculer l'interaction à deux facteurs
model <- lm(pain_score ~ gender*risk*treatment, data = headache)
headache %>%
group_by(gender) %>%
anova_test(pain_score ~ risk*treatment, error = model)## # A tibble: 6 x 8
## gender Effect DFn DFd F p `p<.05` ges
## <fct> <chr> <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 male risk 1 60 50.0 0.00000000187 * 0.455
## 2 male treatment 2 60 10.2 0.000157 * 0.253
## 3 male risk:treatment 2 60 5.25 0.008 * 0.149
## 4 female risk 1 60 42.8 0.0000000150 * 0.416
## 5 female treatment 2 60 0.482 0.62 "" 0.016
## 6 female risk:treatment 2 60 2.87 0.065 "" 0.087On a observé une interaction statistiquement significative entre le risque et le traitement (risk:treatment) chez les hommes, F(2, 60) = 5,25, p = 0,008, mais pas chez les femmes, F(2, 60) = 2,87, p = 0,065.
Pour les hommes, ce résultat suggère que l’effet du traitement sur le “score_douleur” (pain_score) dépend de leur “risque” (risk) au migraine. En d’autres termes, le risque modère l’effet du type de traitement sur le score de la douleur.
Il est à noter que la significativité statistique d’une interaction simple, à deux facteurs, a été acceptée à un niveau alpha ajusté de Bonferroni de 0,025. Cela correspond au niveau actuel auquel vous déclarez une significativité statistique (c.-à-d. p < 0,05) divisé par le nombre d’interactions à deux facteurs que vous analysées (c.-à-d. 2).
Calculer les effets principaux
Une interaction à deux facteurs statistiquement significative peut être suivie par une analyse des effets principaux. Dans notre exemple, vous pourriez donc étudier l’effet du traitement sur le pain_score (score de la douleur) à chaque niveau de risk (risque) ou étudier l’effet du risque à chaque niveau de treatment.
Vous n’aurez besoin de le faire que pour l’interaction à deux facteurs chez les “hommes”, car c’était la seule interaction à deux facteurs qui était statistiquement significative. Le terme d’erreur provient encore une fois de l’ANOVA à trois facteurs.
Regrouper les données par “sexe” (gender) et “risque” (risk) et analyser les effets principaux du traitement sur le “score_douleur” (pain_score):
# Regrouper les données par sexe et risque, et calculer l'anova
treatment.effect <- headache %>%
group_by(gender, risk) %>%
anova_test(pain_score ~ treatment, error = model)
treatment.effect %>% filter(gender == "male")## # A tibble: 2 x 9
## gender risk Effect DFn DFd F p `p<.05` ges
## <fct> <fct> <chr> <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 male high treatment 2 60 14.8 0.0000061 * 0.33
## 2 male low treatment 2 60 0.66 0.521 "" 0.022Dans le tableau ci-dessus, nous n’avons besoin des résultats que pour les effets principaux du traitement pour : (1) “hommes” à risque “faible” ; et (2) “hommes” à risque “élevé.
La significativité statistique a été acceptée à un niveau alpha ajusté de Bonferroni de 0,025, soit 0,05 divisé par le nombre d’effets principaux évalués (c.-à-d. 2).
Il y avait un effet principal statistiquement significatif du traitement chez les hommes à risque élevé de migraine, F(2, 60) = 14,8, p < 0,0001), mais pas chez les hommes à faible risque de migraine, F(2, 60) = 0,66, p = 0,521.
Cette analyse indique que le type de traitement pris a un effet statistiquement significatif sur le score de la douleur chez les hommes qui sont à risque élevé.
En d’autres termes, le score moyen de la douleur dans les groupes de traitement X, Y et Z était statistiquement significativement différent pour les hommes à risque élevé, mais pas pour les hommes à faible risque.
Calculer les comparaisons entre groupes
Un effet principal statistiquement significatif peut être suivi de multiples comparaisons par paires pour déterminer quelles moyennes de groupe sont différentes. Ceci peut être fait facilement en utilisant la fonction emmeans_test() [paquet rstatix] décrite dans la section précédente.
Comparer les différents traitements par gender (genre) et risk (risque):
# Comparaisons par paires
library(emmeans)
pwc <- headache %>%
group_by(gender, risk) %>%
emmeans_test(pain_score ~ treatment, p.adjust.method = "bonferroni") %>%
select(-df, -statistic, -p) # Supprimer les détails
# Montrer les résultats de la comparaison pour les hommes à risque élevé
pwc %>% filter(gender == "male", risk == "high")## # A tibble: 3 x 7
## gender risk .y. group1 group2 p.adj p.adj.signif
## <fct> <fct> <chr> <chr> <chr> <dbl> <chr>
## 1 male high pain_score X Y 0.000386 ***
## 2 male high pain_score X Z 0.00000942 ****
## 3 male high pain_score Y Z 0.897 ns# Moyennes marginales estimées (c.-à-d. moyenne ajustée)
# avec un intervalle de confiance à 95%
get_emmeans(pwc) %>% filter(gender == "male", risk == "high")## # A tibble: 3 x 9
## gender risk treatment emmean se df conf.low conf.high method
## <fct> <fct> <fct> <dbl> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 male high X 92.7 1.80 60 89.1 96.3 Emmeans test
## 2 male high Y 82.3 1.80 60 78.7 85.9 Emmeans test
## 3 male high Z 79.7 1.80 60 76.1 83.3 Emmeans testDans le tableau de comparaisons par paires ci-dessus, nous ne nous intéressons qu’aux comparaisons simples pour les hommes à risque élevé de migraines. Dans notre exemple, il existe trois combinaisons possibles de différences de groupe.
Chez les hommes à risque élevé, la différence moyenne statistiquement significative entre le traitement X et le traitement Y était de 10,4 (p.adj < 0,001), et de 13,1 (p.adj < 0,0001) entre le traitement X et le traitement Z.
Cependant, la différence entre le traitement Y et le traitement Z (2,66) n’était pas statistiquement significative, p.adj = 0,897.
Rapporter
Une ANOVA à trois facteurs a été menée pour déterminer les effets du sexe, du risque et du traitement sur les épisodes migraineux pain_score.
L’analyse des résidus a été effectuée pour vérifier les hypothèses de l’ANOVA à trois facteurs. La normalité a été évaluée à l’aide du test de normalité de Shapiro-Wilk et l’homogénéité des variances a été évaluée par le test de Levene.
Les résidus étaient normalement distribués (p > 0,05) et les variances étaient homogènes (p > 0,05).
Il y avait une interaction, à trois facteurs, statistiquement significative entre le sexe, le risque et le traitement, F(2, 60) = 7.41, p = 0.001.
La significativité statistique a été acceptée au niveau p < 0,025 pour les interactions à deux facteurs et les effets principaux. Il y avait une interaction statistiquement significative entre le risque et le traitement chez les hommes, F(2, 60) = 5,2, p = 0,008, mais pas chez les femmes, F(2, 60) = 2,8, p = 0,065.
Il y avait un effet principal statistiquement significatif du traitement chez les hommes à risque élevé de migraine, F(2, 60) = 14,8, p < 0,0001), mais pas chez les hommes à faible risque de migraine, F(2, 60) = 0,66, p = 0,521.
Toutes les comparaisons par paires, entre les différents groupes de traitement, ont été effectuées pour les hommes présentant un risque élevé de migraine avec un ajustement de Bonferroni.
Il y avait une différence moyenne statistiquement significative entre le traitement X et le traitement Y. Cependant, la différence entre le traitement Y et le traitement Z n’était pas statistiquement significative.
# Visualisation : Boxplots avec p-values
pwc <- pwc %>% add_xy_position(x = "treatment")
pwc.filtered <- pwc %>% filter(gender == "male", risk == "high")
bxp +
stat_pvalue_manual(
pwc.filtered, color = "risk", linetype = "risk", hide.ns = TRUE,
tip.length = 0, step.increase = 0.1, step.group.by = "gender"
) +
labs(
subtitle = get_test_label(res.aov, detailed = TRUE),
caption = get_pwc_label(pwc)
)
Résumé
Cet article décrit comment calculer et interpréter l’ANOVA dans R. Nous expliquons également les hypothèses faites par les tests ANOVA et fournissons des exemples pratiques de codes R pour vérifier si les hypothèses des tests sont respectées.
Version:
 English
English



« Si vous n’avez pas d’interaction à trois facteurs statistiquement significative, vous devez déterminer si vous avez une interaction à deux facteurs statistiquement significative à partir du résultat de l’ANOVA. Vous pouvez suivre une interaction significative à deux facteurs par des analyses simples des effets principaux et des comparaisons par paires entre les groupes si nécessaire. »
Auriez-vous un exemple de la marche à suivre lorsque l’on a une (et surtout deux) interaction(s) significative(s) à deux facteurs dans le tableau d’analyse de la variance à trois facteurs ???
Merci d’avance ,
Merci pour le commentaire. Si vous avez une interaction significative à deux facteurs, vous pouvez continuer par des analyses Post-Hoc comme dans le cas de l’ANOVA à deux facteur.
Bonjour,
Vous avez utilisé quelle version de R pour installer les packages : tidyverse et rstatix ?
Avec la version R.3.6.1 celà ne semble pas fonctionner.
Merci
J’ai utilisé les deux packages sur R3.6.0. Pourriez vous envoyer l’erreur que vous obtenez à la fin en essayant d’installer tidyverse?
Error: package or namespace load failed for ‘tidyverse’ in loadNamespace(j = 0.4.1 est requis
In addition: Warning message:
le package ‘tidyverse’ a été compilé avec la version R 3.6.2
Pareil pour Rstatix :
The following object is masked from ‘package:stats’:
filter
Warning message:
le package ‘rstatix’ a été compilé avec la version R 3.6.2
Exemple très bien détaillés. Merci!
La derniÈre ligne de commande pour la création des box plots finaux avec valeur de p génère systématiquement l’erreur (can’t find the label variable ‘p’ in the data).
Error in stat_pvalue_manual(pwc.filtered, color = « risk », linetype = « risk », : can’t find the label variable ‘p’ in the data
J’utilise R studio Version 1.2.5033 avec R 3.6
Je vous remercie pour votre commentaire positive.
Pour la dernière commande, assurez vous d’avoir une version de ggpubr >= 0.2.5
Merci!
C’est magnifique ! Merci beaucoup pour votre contribution qui me fait gagner un temps précieux dans mes analyses.
Je vous remercie pour votre commentaire positif et je suis content que ce tutoriel vous soit utile.
Merci beaucoup pour cette aide précieuse et efficace! Tout fonctionnait très bien pour moi en le reproduisant avec mes données, à part dans l’Anova à 2 facteurs, lorsque j’ai voulu reproduire le test de comparaison par paires avec emmeans_test, j’obtiens ce message d’erreur :
« Error in contrast.emmGrid(res.emmeans, by = grouping.vars, method = method, :
Nonconforming number of contrast coefficients »
Auriez-vous une solution?
Je ne trouve pas la solution sur d’autres forums =(
Merci beaucoup pour votre travail!
excellent article. Cependant, voulez-vous donner un exemple de ANOVA avec le package WRS2 pour le cas des données aberrantes!
une difficulté rencotrée avec mes données est que lorsque je fais anova, p-value = 0,002 est significative. Mais quand je fais le post-hock de Tukey (pwc), aucune différence significative n’est révélée entre groupes. Ce qui complique la compréhension de p -value significative de ANOVA. Merci donc de m’aider.
Excellent travail, merci pour votre explication détaillée
Bonjour.
Dans « ANOVA à un facteur » — « Hypothèse de normalité » — « Vérifier l’hypothèse de normalité en analysant les résidus du modèle ».
Vous réalisez un test résiduel. Pouvez-vous expliquer dans l’article ce que c’est s’il vous plaît ? Cela m’aiderait à mieux comprendre cette démarche de recherche de normalité. Le mieux serait de l’incorporer directement dans l’article original pour que cela serve à tous.
Merci pour votre travail mais j’ai vu sur plusieurs sites qu’il fallait prêter attention au type de fonction anova sur R qui parfois prend en compte l’ordre d’apparition et parfois non.. Que préconisez-vous pour une ANOVA II dans ce cas ?
merci bcp cet article est très pédagogique . bonne continuation.