L’Analyse de la covariance (ANCOVA) sert à comparer les moyennes d’une variable-réponse entre deux groupes ou plus en tenant compte (ou en corrigeant) la variabilité d’autres variables, appelées covariables. En d’autres termes, l’ANCOVA permet de comparer les moyennes ajustées de deux ou plusieurs groupes indépendants.
Par exemple, vous pouvez comparer le “score au test” par “niveau d’éducation” en tenant compte du “nombre d’heures passées à étudier”. Dans cet exemple : 1) le score du test est notre variable-réponse (ou variable dépendante) ; 2) le niveau de scolarité (secondaire, collégial ou supérieur) est notre variable de groupement ; 3) le temps de travail est notre covariable.
L’ANCOVA, à un facteur, peut être considérée comme une extension de l’ANOVA, à un facteur, qui incorpore une covariable. L’ANCOVA à deux facteurs sert à évaluer simultanément l’effet de deux variables de regroupement indépendantes (A et B) sur une variable-réponse, après ajustement pour une ou plusieurs variables continues, appelées covariables.
Dans cet article, vous apprendrez à:
- Calculer et interpréter l’ANCOVA à un facteur et deux facteurs dans R
- Vérifier les hypothèses ANCOVA
- Effectuer des tests post-hoc, de multiples comparaisons par paires entre les groupes pour identifier les groupes qui sont différents
- Visualiser les données à l’aide de boxplots, ajouter sur le graphique les p-values de l’ANCOVA et des comparaisons par paires
Sommaire:
Livre Apparenté
Pratique des Statistiques dans R II - Comparaison de Groupes: Variables NumériquesHypothèses
L’ANCOVA fait plusieurs hypothèses au sujet des données, telles que:
- Linéarité entre la covariable et la variable-réponse à chaque niveau de la variable de groupement. Ceci peut être vérifié en créant un diagramme de dispersion groupé de la covariable et de la variable-réponse.
- Homogénéité des pentes de régression. Les pentes des droites de régression, formées par la covariable et la variable-réponse, devraient être les mêmes pour chaque groupe. Cette hypothèse évalue qu’il n’y a pas d’interaction entre le résultat et la covariable. Les lignes de régression tracées par groupes doivent être parallèles.
- La variable-réponse doit être approximativement distribuée normalement. Ceci peut être vérifié à l’aide du test de normalité Shapiro-Wilk sur les résidus du modèle.
- Homoscedasticité ou homogénéité de la variance des résidus pour tous les groupes. Les résidus sont supposés avoir une variance constante (homoscédasticité)
- Aucune valeur aberrante significative dans les groupes
Bon nombre de ces hypothèses et problèmes potentiels peuvent être vérifiés en analysant les erreurs résiduelles. Dans le cas où l’hypothèse ANCOVA n’est pas satisfaite, vous pouvez effectuer un test ANCOVA robuste en utilisant le package WRS2.
Prérequis
Assurez-vous d’avoir installé les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixpour des analyses statistiques facilesbroompour l’affichage d’un beau résumé des tests statistiques sous forme de data framedatarium: contient les jeux de données requis pour ce chapitre
Commencez par charger les packages requis suivants:
library(tidyverse)
library(ggpubr)
library(rstatix)
library(broom)ANCOVA à un facteur
Préparation des données
Nous préparerons nos données de démonstration à partir du jeu de données sur l’anxiété disponible dans le package datarium.
Les chercheurs ont étudié l’effet des exercices sur la réduction du niveau d’anxiété. Par conséquent, ils ont mené une expérience où ils ont mesuré le score d’anxiété de trois groupes d’individus pratiquant des exercices physiques à différents niveaux (grp1 : faible, grp2 : modéré et grp3 : élevé).
Le score d’anxiété a été mesuré avant et six mois après les programmes d’exercices. On s’attend à ce que toute réduction de l’anxiété par les programmes d’exercices dépende également du niveau basal d’anxiété du participant.
Dans cette analyse, nous utilisons le score d’anxiété pré-test comme covariable et nous nous intéressons aux différences possibles entre les groupes par rapport aux scores d’anxiété post-test.
# Charger et préparer les données
data("anxiety", package = "datarium")
anxiety <- anxiety %>%
select(id, group, t1, t3) %>%
rename(pretest = t1, posttest = t3)
anxiety[14, "posttest"] <- 19
# Inspectez les données en affichant une ligne aléatoire par groupe
set.seed(123)
anxiety %>% sample_n_by(group, size = 1)## # A tibble: 3 x 4
## id group pretest posttest
## <fct> <fct> <dbl> <dbl>
## 1 5 grp1 16.5 15.7
## 2 27 grp2 17.8 16.9
## 3 37 grp3 17.1 14.3Vérifier les hypothèses
Hypothèse de linéarité
- Créer un diagramme de dispersion entre la covariable (
prétest) et la variable-réponse (post-test) - Ajouter des lignes de régression, montrer les équations correspondantes et le R2 par groupes
ggscatter(
anxiety, x = "pretest", y = "posttest",
color = "group", add = "reg.line"
)+
stat_regline_equation(
aes(label = paste(..eq.label.., ..rr.label.., sep = "~~~~"), color = group)
)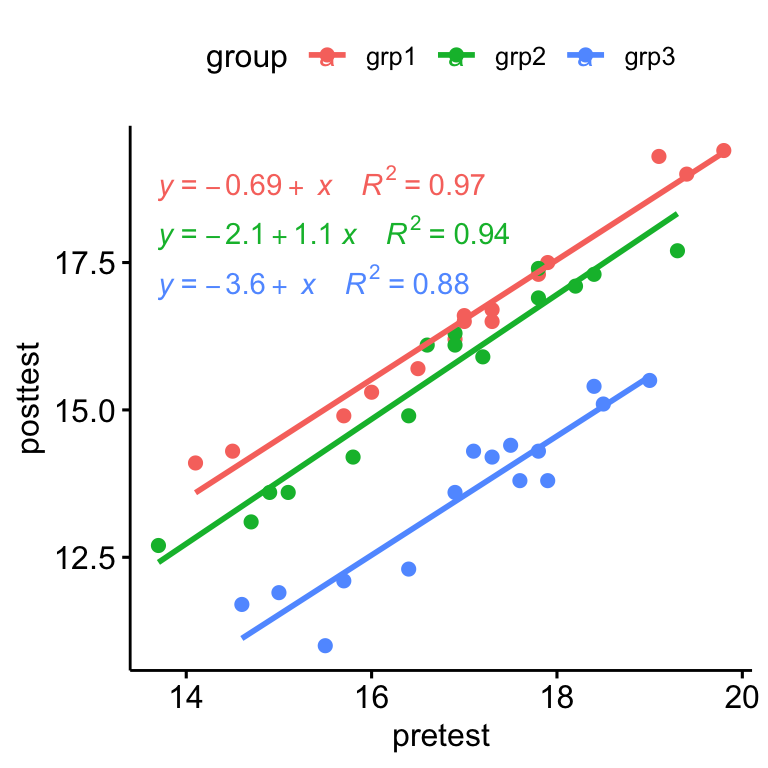
Il y avait une relation linéaire entre le score d’anxiété pré-test et post-test pour chaque groupe d’entraînement, tel qu’évalué par l’inspection visuelle du nuage de points.
Homogénéité des pentes de régression
Cette hypothèse vérifie qu’il n’y a pas d’interaction significative entre la covariable et la variable de groupement. Ceci peut être évalué comme suit:
anxiety %>% anova_test(posttest ~ group*pretest)## ANOVA Table (type II tests)
##
## Effect DFn DFd F p p<.05 ges
## 1 group 2 39 209.314 1.40e-21 * 0.915
## 2 pretest 1 39 572.828 6.36e-25 * 0.936
## 3 group:pretest 2 39 0.127 8.81e-01 0.006Il y avait homogénéité des pentes de régression puisque le terme d’interaction n’était pas statistiquement significatif, F(2, 39) = 0,13, p = 0,88.
Normalité des résidus
Vous devez d’abord calculer le modèle en utilisant lm(). Dans R, vous pouvez facilement augmenter vos données pour ajouter des valeurs prédites et des résidus en utilisant la fonction augment(model) [package broom]. Appelons le résultat model.metrics car elle contient plusieurs métriques utiles pour les diagnostics de régression.
# Calculer le modèle, la covariable passe en premier
model <- lm(posttest ~ pretest + group, data = anxiety)
# Inspecter les paramètres de diagnostic du modèle
model.metrics <- augment(model) %>%
select(-.hat, -.sigma, -.fitted, -.se.fit) # Supprimer les détails
head(model.metrics, 3)## # A tibble: 3 x 6
## posttest pretest group .resid .cooksd .std.resid
## <dbl> <dbl> <fct> <dbl> <dbl> <dbl>
## 1 14.1 14.1 grp1 0.550 0.101 1.46
## 2 14.3 14.5 grp1 0.338 0.0310 0.885
## 3 14.9 15.7 grp1 -0.295 0.0133 -0.750# Évaluer la normalité des résidus à l'aide du test de Shapiro-Wilk
shapiro_test(model.metrics$.resid)## # A tibble: 1 x 3
## variable statistic p.value
## <chr> <dbl> <dbl>
## 1 model.metrics$.resid 0.975 0.444Le test de Shapiro-Wilk n’était pas significatif (p > 0,05), on peut donc supposer une normalité des résidus
Homogénéité des variances
L’ANCOVA suppose que la variance des résidus est égale pour tous les groupes. Ceci peut être vérifié à l’aide du test de Levene:
model.metrics %>% levene_test(.resid ~ group)## # A tibble: 1 x 4
## df1 df2 statistic p
## <int> <int> <dbl> <dbl>
## 1 2 42 2.27 0.116Le test de Levene n’était pas significatif (p > 0,05), nous pouvons donc supposer l’homogénéité des variances résiduelles pour tous les groupes.
Valeurs aberrantes
Une valeur aberrante est un point qui a une valeur extrême de variable-réponse. La présence de valeurs aberrantes peut influer sur l’interprétation du modèle.
Les valeurs aberrantes peuvent être identifiées en examinant les résidus normalisé (ou résidus studentisés), qui est le résidu divisé par son erreur type estimée. Les résidus normalisés peuvent être interprétés comme le nombre d’erreurs-types en dehors de la ligne de régression.
Les observations dont les résidus normalisés sont supérieurs à 3 en valeur absolue sont des valeurs aberrantes possibles.
model.metrics %>%
filter(abs(.std.resid) > 3) %>%
as.data.frame()## [1] posttest pretest group .resid .cooksd .std.resid
## <0 rows> (or 0-length row.names)Il n’y avait pas de valeurs aberrantes dans les données, telles qu’évaluées par l’absence de cas avec des résidus normalisés supérieurs à 3 en valeur absolue.
Calculs
L’ordre des variables est important dans le calcul de l’ANCOVA. Vous voulez d’abord supprimer l’effet de la covariable - c’est-à-dire, vous voulez le contrôler - avant d’entrer votre variable principale ou votre intérêt.
La covariable passe en premier (et il n’y a pas d’interaction) ! Si vous ne le faites pas dans l’ordre, vous obtiendrez des résultats différents.
res.aov <- anxiety %>% anova_test(posttest ~ pretest + group)
get_anova_table(res.aov)## ANOVA Table (type II tests)
##
## Effect DFn DFd F p p<.05 ges
## 1 pretest 1 41 598 4.48e-26 * 0.936
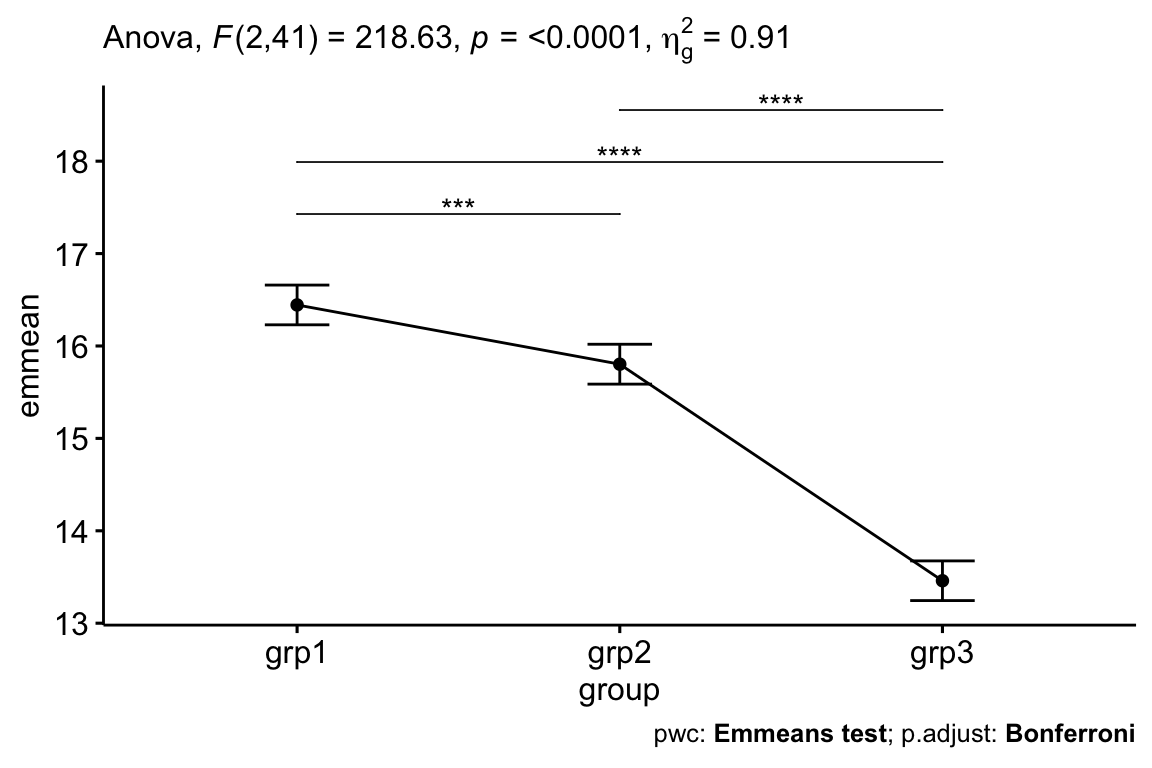
## 2 group 2 41 219 1.35e-22 * 0.914Après ajustement pour tenir compte du score d’anxiété pré-test, il y avait une différence statistiquement significative du score d’anxiété post-test entre les groupes, F(2, 41) = 218,63, p < 0,0001.
Test post-hoc
Des comparaisons par paires peuvent être effectuées pour identifier les groupes qui sont statistiquement différents. La correction de tests multiples de Bonferroni est appliquée. Ceci peut être fait facilement en utilisant la fonction emmeans_test() [paquet rstatix], un wrapper autour du paquet emmeans, qui doit être installé. Emmeans (estimated marginal means en anglais) signifie moyennes marginales estimées (c’est-à-dire moyennes des moindres carrés ou moyennes ajustées).
# Comparaisons par paires
library(emmeans)
pwc <- anxiety %>%
emmeans_test(
posttest ~ group, covariate = pretest,
p.adjust.method = "bonferroni"
)
pwc## # A tibble: 3 x 8
## .y. group1 group2 df statistic p p.adj p.adj.signif
## * <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 posttest grp1 grp2 41 4.24 1.26e- 4 3.77e- 4 ***
## 2 posttest grp1 grp3 41 19.9 1.19e-22 3.58e-22 ****
## 3 posttest grp2 grp3 41 15.5 9.21e-19 2.76e-18 ****# Afficher les moyennes ajustées de chaque groupe
# Également appelée moyenne marginale estimée (emmeans)
get_emmeans(pwc)## # A tibble: 3 x 8
## pretest group emmean se df conf.low conf.high method
## <dbl> <fct> <dbl> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 16.9 grp1 16.4 0.106 41 16.2 16.7 Emmeans test
## 2 16.9 grp2 15.8 0.107 41 15.6 16.0 Emmeans test
## 3 16.9 grp3 13.5 0.106 41 13.2 13.7 Emmeans testLes données sont des moyennes +/- erreur-type ajustées. Le score moyen d’anxiété était statistiquement significativement plus élevé dans grp1 (16,4 +/- 0,15) que dans grp2 (15,8 +/- 0,12) et grp3 (13,5 +/_ 0,11), p < 0,001.
Rapporter
Une ANCOVA a été effectuée pour déterminer l’effet des exercices sur le score d’anxiété après contrôle du score d’anxiété basale des participants.
Après ajustement pour tenir compte du score d’anxiété pré-test, il y avait une différence statistiquement significative du score d’anxiété post-test entre les groupes, F(2, 41) = 218,63, p < 0,0001.
L’analyse post-hoc a été effectuée avec un ajustement de Bonferroni. Le score moyen d’anxiété était statistiquement significativement plus élevé dans grp1 (16,4 +/- 0,15) que dans grp2 (15,8 +/- 0,12) et grp3 (13,5 +/_ 0,11), p < 0,001.
# Visualisation : Line plots avec p-values
pwc <- pwc %>% add_xy_position(x = "group", fun = "mean_se")
ggline(get_emmeans(pwc), x = "group", y = "emmean") +
geom_errorbar(aes(ymin = conf.low, ymax = conf.high), width = 0.2) +
stat_pvalue_manual(pwc, hide.ns = TRUE, tip.length = FALSE) +
labs(
subtitle = get_test_label(res.aov, detailed = TRUE),
caption = get_pwc_label(pwc)
)
ANCOVA à deux facteurs
Préparation des données
Nous utiliserons le jeu de données stress disponible dans le package datarium. Dans cette étude, un chercheur veut évaluer l’effet du traitement (treatment) et de l’ exercice (exercise) sur le score de réduction du stress après ajustement pour age.
Dans cet exemple : 1) le score de stress est notre variable-réponse (ou variable dépendante) ; 2) treatment (groups : non et oui) et exercises (groups : faible, moyen et élevé) sont nos variables de regroupement ; 3) age est notre covariable.
Charger les données et afficher quelques lignes aléatoires par groupes:
data("stress", package = "datarium")
stress %>% sample_n_by(treatment, exercise)## # A tibble: 6 x 5
## id score treatment exercise age
## <int> <dbl> <fct> <fct> <dbl>
## 1 8 83.8 yes low 61
## 2 15 86.9 yes moderate 55
## 3 29 71.5 yes high 55
## 4 40 92.4 no low 67
## 5 41 100 no moderate 75
## 6 56 82.4 no high 53Vérifier les hypothèses
Hypothèse de linéarité
- Créez un diagramme de dispersion entre la covariable (
age) et la variable-réponse (score) pour chaque combinaison des groupes des deux variables de groupement: - Ajoutez des lignes lisses de loess, ce qui aide à décider si la relation est linéaire ou non
ggscatter(
stress, x = "age", y = "score",
facet.by = c("exercise", "treatment"),
short.panel.labs = FALSE
)+
stat_smooth(method = "loess", span = 0.9)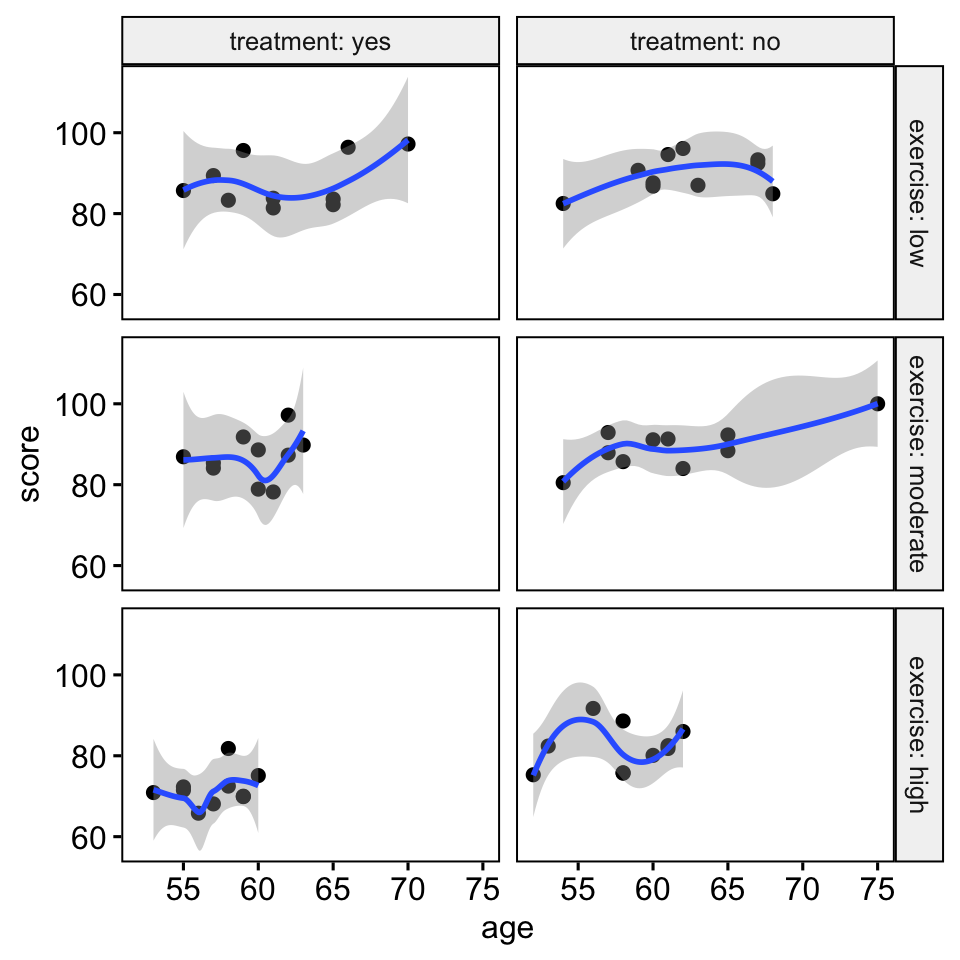
Il y a une relation linéaire entre la covariable (variable de l’âge) et la variable-réponse (score) pour chaque groupe, telle qu’évaluée par une inspection visuelle du diagramme de dispersion.
Homogénéité des pentes de régression
Cette hypothèse vérifie qu’il n’y a pas d’interaction significative entre la covariable et les variables de groupement. Ceci peut être évalué comme suit:
stress %>%
anova_test(
score ~ age + treatment + exercise +
treatment*exercise + age*treatment +
age*exercise + age*exercise*treatment
)## ANOVA Table (type II tests)
##
## Effect DFn DFd F p p<.05 ges
## 1 age 1 48 8.359 6.00e-03 * 0.148000
## 2 treatment 1 48 9.907 3.00e-03 * 0.171000
## 3 exercise 2 48 18.197 1.31e-06 * 0.431000
## 4 treatment:exercise 2 48 3.303 4.50e-02 * 0.121000
## 5 age:treatment 1 48 0.009 9.25e-01 0.000189
## 6 age:exercise 2 48 0.235 7.91e-01 0.010000
## 7 age:treatment:exercise 2 48 0.073 9.30e-01 0.003000Une autre solution simple consiste à créer une nouvelle variable de regroupement, par exemple groupe, à partir des combinaisons des variables existantes, puis à calculer le modèle ANOVA:
stress %>%
unite(col = "group", treatment, exercise) %>%
anova_test(score ~ group*age)## ANOVA Table (type II tests)
##
## Effect DFn DFd F p p<.05 ges
## 1 group 5 48 10.912 4.76e-07 * 0.532
## 2 age 1 48 8.359 6.00e-03 * 0.148
## 3 group:age 5 48 0.126 9.86e-01 0.013Les pentes de régression étaient homogènes, car les termes d’interaction entre les covariables (âge) et les variables de groupement (traitement et exercice) n’étaient pas statistiquement significatifs, p > 0,05.
Normalité des résidus
# Calculer le modèle, la covariable passe en premier
model <- lm(score ~ age + treatment*exercise, data = stress)
# Inspecter les paramètres de diagnostic du modèle
model.metrics <- augment(model) %>%
select(-.hat, -.sigma, -.fitted, -.se.fit) # Supprimer les détails
head(model.metrics, 3)## # A tibble: 3 x 7
## score age treatment exercise .resid .cooksd .std.resid
## <dbl> <dbl> <fct> <fct> <dbl> <dbl> <dbl>
## 1 95.6 59 yes low 9.10 0.0647 1.93
## 2 82.2 65 yes low -7.32 0.0439 -1.56
## 3 97.2 70 yes low 5.16 0.0401 1.14# Évaluer la normalité des résidus à l'aide du test de Shapiro-Wilk
shapiro_test(model.metrics$.resid)## # A tibble: 1 x 3
## variable statistic p.value
## <chr> <dbl> <dbl>
## 1 model.metrics$.resid 0.982 0.531Le test de Shapiro-Wilk n’était pas significatif (p > 0,05), on peut donc supposer une normalité des résidus
Homogénéité des variances
L’ANCOVA suppose que la variance des résidus est égale pour tous les groupes. Ceci peut être vérifié à l’aide du test de Levene:
levene_test(.resid ~ treatment*exercise, data = model.metrics)Le test de Levene n’était pas significatif (p > 0,05), nous pouvons donc supposer l’homogénéité des variances résiduelles pour tous les groupes.
Valeurs aberrantes
Les observations dont les résidus normalisés sont supérieurs à 3 en valeur absolue sont des valeurs aberrantes possibles.
model.metrics %>%
filter(abs(.std.resid) > 3) %>%
as.data.frame()## [1] score age treatment exercise .resid .cooksd .std.resid
## <0 rows> (or 0-length row.names)Il n’y avait pas de valeurs aberrantes dans les données, telles qu’évaluées par l’absence de cas avec des résidus normalisés supérieurs à 3 en valeur absolue.
Calculs
res.aov <- stress %>%
anova_test(score ~ age + treatment*exercise)
get_anova_table(res.aov)## ANOVA Table (type II tests)
##
## Effect DFn DFd F p p<.05 ges
## 1 age 1 53 9.11 4.00e-03 * 0.147
## 2 treatment 1 53 11.10 2.00e-03 * 0.173
## 3 exercise 2 53 20.82 2.13e-07 * 0.440
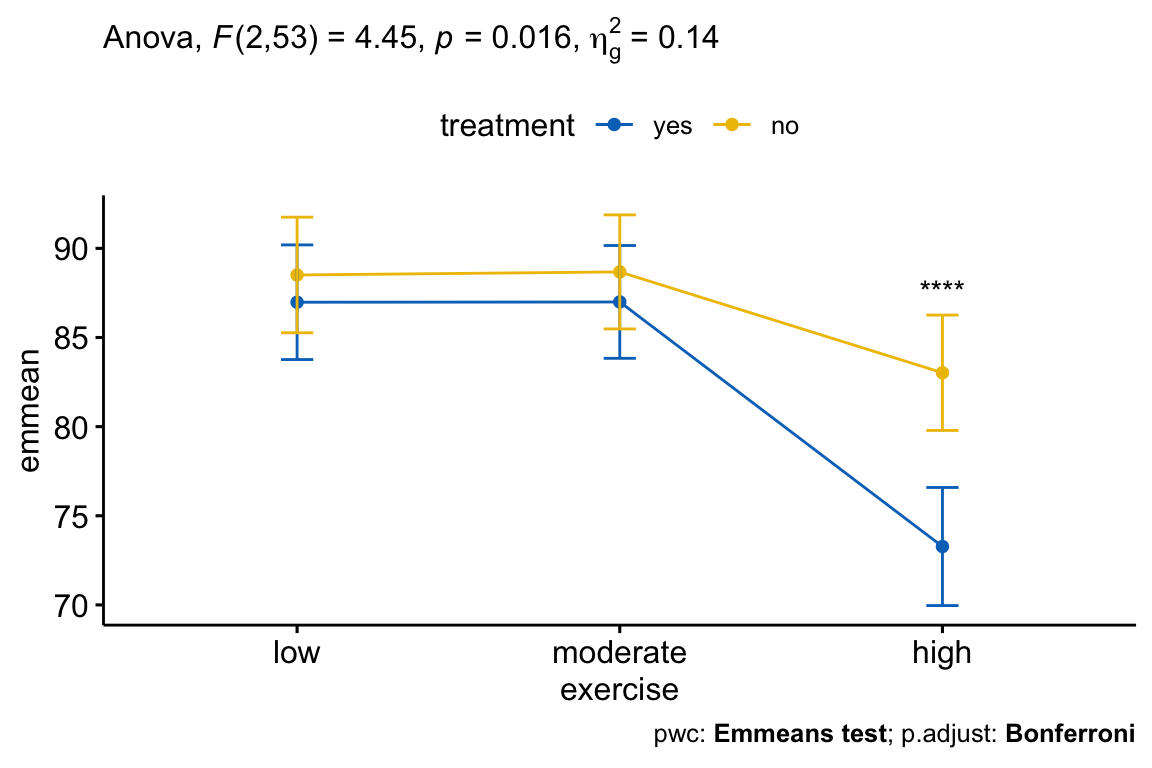
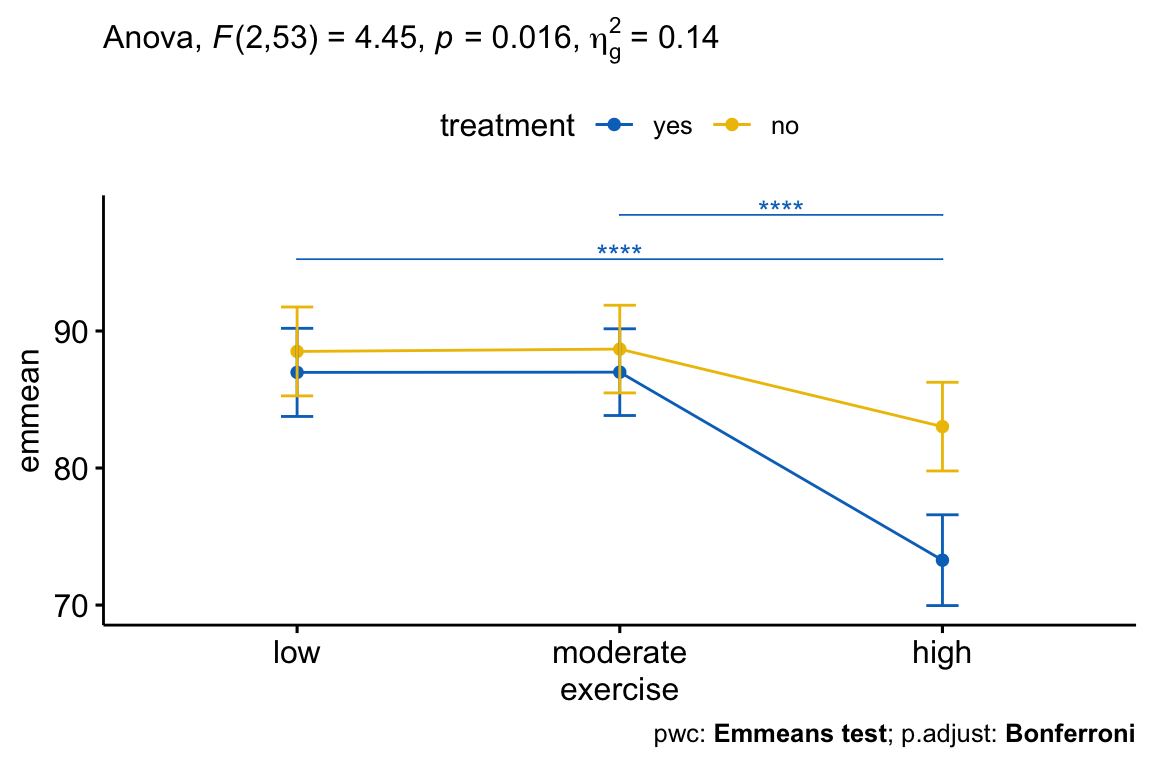
## 4 treatment:exercise 2 53 4.45 1.60e-02 * 0.144Après ajustement pour l’âge, il y avait une interaction statistiquement significative entre le traitement et l’exercice sur le score de stress, F(2, 53) = 4,45, p = 0,016. Cela indique que l’effet de l’exercice sur le score dépend du traitement, et vice-versa.
Test post-hoc
Une interaction statistiquement significative, à deux facteurs, peut être suivie d’analyses d’effets principaux, c’est-à-dire l’évaluation de l’effet d’une variable à chaque niveau de la deuxième variable, et vice-versa.
Dans le cas où l’interaction n’est pas significative, vous pouvez indiquer l’effet principal de chaque variable de regroupement.
Une interaction significative à deux facteurs indique que l’impact d’un facteur sur la variable-réponse dépend du niveau de l’autre facteur (et vice versa). Ainsi, vous pouvez décomposer une interaction significative, à deux facteurs, en:
- Effet principal : exécuter le modèle à un facteur avec la première variable (facteur A) à chaque niveau de la deuxième variable (facteur B),
- Comparaisons par paires : si l’effet principal est significatif, effectuez plusieurs comparaisons par paires pour déterminer quels groupes sont différents.
Dans le cas d’une interaction, à deux facteurs, non significative, vous devez déterminer si vous avez des effets principaux statistiquement significatifs dans le résultat de l’ANCOVA.
Dans cette section, nous décrirons la procédure à suivre pour une interaction significative à trois facteurs
Analyses de l’effet principal pour le traitement
Analyser l’effet principal simple de treatment at each level of exercise. Regroupez les données par exercice (exercise) et effectuez une ANCOVA pour le traitement (treatment) en ajustant pour age:
# Effet du traitement à chaque niveau d'exercice
stress %>%
group_by(exercise) %>%
anova_test(score ~ age + treatment)## # A tibble: 6 x 8
## exercise Effect DFn DFd F p `p<.05` ges
## <fct> <chr> <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 low age 1 17 2.25 0.152 "" 0.117
## 2 low treatment 1 17 0.437 0.517 "" 0.025
## 3 moderate age 1 17 6.65 0.02 * 0.281
## 4 moderate treatment 1 17 0.419 0.526 "" 0.024
## 5 high age 1 17 0.794 0.385 "" 0.045
## 6 high treatment 1 17 18.7 0.000455 * 0.524Notez que nous devons appliquer l’ajustement de Bonferroni pour les corrections de tests multiples. Une approche courante consiste à baisser le niveau auquel vous déclarez la significativité en divisant la valeur alpha (0,05) par le nombre de tests effectués. Dans notre exemple, c’est-à-dire 0,05/3 = 0,01666667.
La significativité statistique a été acceptée au niveau alpha ajusté de Bonferroni de 0,01667, soit 0,05/3. L’effet du traitement était statistiquement significatif dans le groupe des exercices d’intensité élevée (p = 0,00045), mais pas dans le groupe des exercices de faible intensité (p = 0,517) et dans le groupe des exercices d’intensité moyenne (p = 0,526).
Calculer des comparaisons par paires entre les groupes de traitement à chaque niveau d’exercice. La correction de tests multiples de Bonferroni est appliquée.
# Comparaisons par paires
pwc <- stress %>%
group_by(exercise) %>%
emmeans_test(
score ~ treatment, covariate = age,
p.adjust.method = "bonferroni"
)
pwc %>% filter(exercise == "high")## # A tibble: 1 x 9
## exercise .y. group1 group2 df statistic p p.adj p.adj.signif
## <fct> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 high score yes no 53 -4.36 0.0000597 0.0000597 ****Dans le tableau de comparaison par paires, vous n’aurez besoin du résultat que pour le groupe “exercices:high”, car c’était la seule condition où l’effet principal du traitement était statistiquement significatif.
Les comparaisons par paires entre le groupe treatment:no et treatment:yes étaient statistiquement significatives chez les participants qui entreprenaient des exercices à haute intensité (p < 0,0001).
Effet principal simple pour l’exercice
Vous pouvez faire les mêmes analyses post-hoc pour la variable exercice à chaque niveau de la variable traitement.
# Effet de l'exercice à chaque niveau de traitement
stress %>%
group_by(treatment) %>%
anova_test(score ~ age + exercise)## # A tibble: 4 x 8
## treatment Effect DFn DFd F p `p<.05` ges
## <fct> <chr> <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 yes age 1 26 2.37 0.136 "" 0.083
## 2 yes exercise 2 26 17.3 0.0000164 * 0.572
## 3 no age 1 26 7.26 0.012 * 0.218
## 4 no exercise 2 26 3.99 0.031 * 0.235La significativité statistique a été acceptée au niveau alpha ajusté de Bonferroni de 0,025, soit 0,05/2 (le nombre de tests). L’effet de l’exercice était statistiquement significatif dans le groupe treatment=yes (p < 0,0001), mais pas dans le groupe treatment=no (p = 0,031).
Effectuer plusieurs comparaisons par paires entre exercise groups at each level of treatment. Il n’est pas nécessaire d’interpréter les résultats pour le groupe “no treatment” (sans traitement), car l’effet de l’exercice n’était pas significatif pour ce groupe.
pwc2 <- stress %>%
group_by(treatment) %>%
emmeans_test(
score ~ exercise, covariate = age,
p.adjust.method = "bonferroni"
) %>%
select(-df, -statistic, -p) # Supprimer les détails
pwc2 %>% filter(treatment == "yes")## # A tibble: 3 x 6
## treatment .y. group1 group2 p.adj p.adj.signif
## <fct> <chr> <chr> <chr> <dbl> <chr>
## 1 yes score low moderate 1 ns
## 2 yes score low high 0.00000113 ****
## 3 yes score moderate high 0.000000466 ****Il y avait une différence statistiquement significative entre la moyenne ajustée du groupe à faible et forte activité d’exercices (p < 0,0001) et, entre le groupe modéré et le groupe élevé (p < 0,0001). La différence entre la moyenne ajustée des valeurs faibles et modérées n’était pas significative.
Rapporter
Une ANCOVA, à deux facteurs, a été effectuée pour examiner les effets du traitement et de l’exercice sur la réduction du stress, après contrôle de l’âge.
Il y avait une interaction statistiquement significative entre le traitement et l’exercice sur la concentration du score, tout en tenant compte de l’âge, F(2, 53) = 4,45, p = 0,016.
Par conséquent, une analyse des effets principaux de l’exercice et du traitement a été effectuée avec une signification statistique recevant un ajustement de Bonferroni et étant acceptée au niveau p < 0,025 pour l’exercice et p < 0,0167 pour le traitement.
L’effet principal du traitement était statistiquement significatif dans le groupe des exercices de forte intensité (p = 0,00046), mais pas dans le groupe des exercices de faible intensité (p = 0,52) et le groupe des exercices de moyenne intensité (p = 0,53).
L’effet de l’exercice était statistiquement significatif dans le groupe treatment=yes (p < 0,0001), mais pas dans le groupe treatment=no (p = 0,031).
Toutes les comparaisons par paires ont été calculées pour les effets principaux statistiquement significatifs avec des p-values rapportées ajustées de Bonferroni. Pour le groupe treatment=yes, il y avait une différence statistiquement significative entre la moyenne ajustée du groupe à faible et à forte intensité d’exercices (p < 0,0001) et, entre le groupe modéré et le groupe élevé (p < 0,0001). La différence entre les moyennes ajustées des groupes d’exercice faible et modéré n’était pas significative.
- Créer un line plot:
# Line plot
lp <- ggline(
get_emmeans(pwc), x = "exercise", y = "emmean",
color = "treatment", palette = "jco"
) +
geom_errorbar(
aes(ymin = conf.low, ymax = conf.high, color = treatment),
width = 0.1
)- Ajouter des p-values
# Comparaisons entre les groupes de traitement à chaque niveau d'exercice
pwc <- pwc %>% add_xy_position(x = "exercise", fun = "mean_se", step.increase = 0.2)
pwc.filtered <- pwc %>% filter(exercise == "high")
lp +
stat_pvalue_manual(
pwc.filtered, hide.ns = TRUE, tip.length = 0,
bracket.size = 0
) +
labs(
subtitle = get_test_label(res.aov, detailed = TRUE),
caption = get_pwc_label(pwc)
)
# Comparaisons entre les groupes d'exercices à chaque niveau de traitement
pwc2 <- pwc2 %>% add_xy_position(x = "exercise", fun = "mean_se")
pwc2.filtered <- pwc2 %>% filter(treatment == "yes")
lp +
stat_pvalue_manual(
pwc2.filtered, hide.ns = TRUE, tip.length = 0,
step.group.by = "treatment", color = "treatment"
) +
labs(
subtitle = get_test_label(res.aov, detailed = TRUE),
caption = get_pwc_label(pwc2)
)
Résumé
Cet article décrit comment calculer et interpréter l’ANCOVA à un et deux facteurs dans R. Nous expliquons également les hypothèses faites par les tests ANCOVA et fournissons des exemples pratiques de codes R pour vérifier si les hypothèses des tests sont respectées ou non.
Version:
 English
English



It is good. But tell it is not possible to use 3, 4 or 5 factors and 2 or 3 covariables ?
How Can we construct a composite indicator from 4 or 5 binar variables ?