L’ANOVA sur mesures répétées est utilisée pour l’analyse de données lorsque les mêmes sujets sont mesurés plus d’une fois. Ce test est également appelé ANOVA intra-sujets ou ANOVA sur mesures répétées. Le terme “intra-sujets” signifie que les mêmes individus sont mesurés sur la même variable-réponse à des moments ou dans des conditions différents.
Par exemple, vous pourriez avoir mesuré l’estime de soi de 10 personnes (variable-réponse ou variable-dépendante) à trois moments au cours d’un régime alimentaire particulier pour déterminer si leur estime de soi s’est améliorée.
Ce chapitre décrit les différents types d’ANOVA sur mesures répétées, notamment:
- ANOVA à un facteur sur mesures répétées, une extension du test t apparié pour comparer les moyennes de trois niveaux ou plus d’une variable intra-sujets.
- ANOVA à deux facteurs sur mesures répétées utilisée pour évaluer simultanément l’effet de deux facteurs intra-sujets sur une variable-réponse continue.
- ANOVA à trois facteurs sur mesures répétées utilisées pour évaluer simultanément l’effet de trois facteurs intra-sujets sur une variable-réponse continue.
L’objectif principal de l’ANOVA à deux et à trois facteurs sur mesures répétées est, respectivement, d’évaluer s’il existe un effet d’interaction statistiquement significatif entre deux et trois facteurs intra-sujets pour expliquer une variable-réponse continue.
Vous apprendrez à:
- Calculer et interpréter les différentes types d’ANOVA sur mesures répétées dans R.
- Vérifier les hypothèses des tests ANOVA sur mesures répétées
- Effectuer des tests post-hoc, de multiples comparaisons par paires entre les groupes pour identifier les groupes qui sont différents
- Visualiser les données avec des boxplots, ajouter au graphique, les p-values de l’ANOVA et celles des comparaisons multiples par paires
Sommaire:
Livre Apparenté
Pratique des Statistiques dans R II - Comparaison de Groupes: Variables NumériquesHypothèses
L’ANOVA sur mesures répétées reposent sur les hypothèses suivantes au sujet des données:
- Aucune valeur aberrante significative dans aucune cellule du plan. Ceci peut être vérifié en visualisant les données à l’aide de boxplots et en utilisant la fonction
identify_outliers()[package rstatix]. - Normalité : la variable-réponse (ou dépendante) doit être distribuée approximativement normalement dans chaque cellule du plan expérimental. Ceci peut être vérifié en utilisant le test de normalité de Shapiro-Wilk (
shapiro_test()[rstatix]) ou par inspection visuelle en utilisant le QQ plot (ggqqplot()[ggpubr package]). - Hypothèse de sphéricité : la variance des différences entre les groupes doit être égale. Ceci peut être vérifié à l’aide du test de sphéricité de Mauchly, qui est automatiquement rapporté en utilisant la fonction R
anova_test()[package rstatix]. Pour en savoir plus, lisez le chapitre @ref(mauchly-s-test-of-sphericity-in-r).
Avant de calculer le test ANOVA sur mesures répétées, vous devez effectuer quelques tests préliminaires pour vérifier si les hypothèses sont respectées.
Notez que, si les hypothèses ci-dessus ne sont pas satisfaites, il existe une alternative non paramétrique (test de Friedman) à l’ANOVA à un facteur sur mesures répétées.
Malheureusement, il n’existe pas d’alternatives non paramétriques à l’ANOVA à deux/trois facteurs sur mesures répétées. Ainsi, dans le cas où les hypothèses ne sont pas satisfaites, vous pourriez envisager d’exécuter l’ANOVA sur les données transformées et non transformées pour voir s’il y a des différences significatives.
Si les deux tests vous amènent aux mêmes conclusions, il se peut que vous ne choisissiez pas de transformer la variable-réponse et de continuer avec l’ANOVA sur les données originelles.
Il est également possible d’effectuer un test ANOVA robuste à l’aide du package R WRS2.
Quel que soit votre choix, vous devez rapporter ce que vous avez fait dans vos résultats.
Prérequis
Assurez-vous d’avoir installé les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixcontient des fonctions R facilitant les analyses statistiquesdatarium: contient les jeux de données requis pour ce chapitre
Commencez par charger les paquets R suivants:
library(tidyverse)
library(ggpubr)
library(rstatix)Fonctions R clés:
anova_test()[paquet rstatix], un wrapper autour decar::Anova()pour faciliter le calcul de l’ANOVA sur mesures répétées. Principaux arguments pour executer l’ANOVA sur mesures répétées:data: data framedv: (numérique) le nom de la variable dépendante (ou variable-réponse).wid: nom de la variable spécifiant l’identificateur de cas/échantillon.within: facteur ou variable de groupement intra-sujets
get_anova_table()[paquet rstatix]. Extrait le tableau ANOVA à partir du résultat deanova_test(). Elle retourne le tableau ANOVA qui est automatiquement corrigé pour tenir compte d’un éventuel écart par rapport à l’hypothèse de sphéricité. Par défaut, la correction de sphéricité de Greenhouse-Geisser est appliquée automatiquement aux seuls facteurs intra-sujets violant l’hypothèse de sphéricité (c.-à-d. la valeur p du test de Mauchly est significative, p <= 0,05). Pour en savoir plus, lisez le chapitre @ref(mauchly-s-test-of-sphericity-in-r).
ANOVA à un facteur sur mesures répétées
Préparation des données
Nous utiliserons le jeu de données sur l’estime de soi mesuré sur trois points temporels. Les données sont disponibles dans le package datarium.
# Préparation des données
# Format large
data("selfesteem", package = "datarium")
head(selfesteem, 3)## # A tibble: 3 x 4
## id t1 t2 t3
## <int> <dbl> <dbl> <dbl>
## 1 1 4.01 5.18 7.11
## 2 2 2.56 6.91 6.31
## 3 3 3.24 4.44 9.78# Rassembler les colonnes t1, t2 et t3 en format long
# Convertir l'identifiant et le temps en facteurs
selfesteem <- selfesteem %>%
gather(key = "time", value = "score", t1, t2, t3) %>%
convert_as_factor(id, time)
head(selfesteem, 3)## # A tibble: 3 x 3
## id time score
## <fct> <fct> <dbl>
## 1 1 t1 4.01
## 2 2 t1 2.56
## 3 3 t1 3.24L’ANOVA à un facteur sur mesures répétées peut être utilisée pour déterminer si les scores moyens d’estime de soi sont significativement différents entre les trois temps.
Statistiques descriptives
Calculer quelques statistiques sommaires du score d’estime de soi par groupe (temps) : moyenne et sd (écart-type)
selfesteem %>%
group_by(time) %>%
get_summary_stats(score, type = "mean_sd")## # A tibble: 3 x 5
## time variable n mean sd
## <fct> <chr> <dbl> <dbl> <dbl>
## 1 t1 score 10 3.14 0.552
## 2 t2 score 10 4.93 0.863
## 3 t3 score 10 7.64 1.14Visualisation
Créer un box plot et ajouter des points correspondant à des valeurs individuelles:
bxp <- ggboxplot(selfesteem, x = "time", y = "score", add = "point")
bxp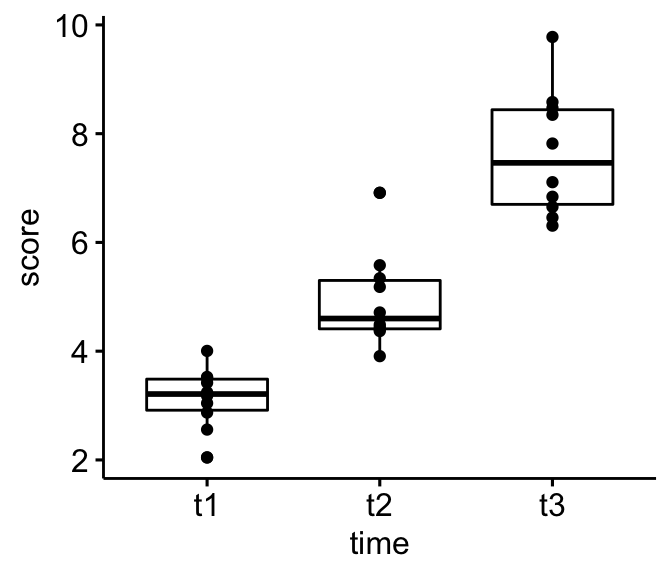
Vérifier les hypothèses
Valeurs aberrantes
Les valeurs aberrantes peuvent être facilement identifiées à l’aide de méthodes des boxplots, implémentées dans la fonction R identify_outliers() [paquet rstatix].
selfesteem %>%
group_by(time) %>%
identify_outliers(score)## # A tibble: 2 x 5
## time id score is.outlier is.extreme
## <fct> <fct> <dbl> <lgl> <lgl>
## 1 t1 6 2.05 TRUE FALSE
## 2 t2 2 6.91 TRUE FALSEIl n’y avait pas de valeurs extrêmes aberrantes.
Notez que, dans le cas où vous avez des valeurs extrêmes aberrantes, cela peut être dû à : 1) erreurs de saisie de données, erreurs de mesure ou valeurs inhabituelles.
Vous pouvez quand même inclure la valeur aberrante dans l’analyse si vous ne croyez pas que le résultat sera affecté de façon substantielle. Ceci peut être évalué en comparant le résultat de l’ANOVA avec et sans la valeur aberrante.
Il est également possible de conserver les valeurs aberrantes dans les données et d’effectuer un test ANOVA robuste en utilisant le package WRS2.
Hypothèse de normalité
L’hypothèse de normalité peut être vérifiée en calculant le test de Shapiro-Wilk pour chaque point dans le temps. Si les données sont normalement distribuées, la p-value doit être supérieure à 0,05.
selfesteem %>%
group_by(time) %>%
shapiro_test(score)## # A tibble: 3 x 4
## time variable statistic p
## <fct> <chr> <dbl> <dbl>
## 1 t1 score 0.967 0.859
## 2 t2 score 0.876 0.117
## 3 t3 score 0.923 0.380Le score d’estime de soi était normalement distribué à chaque point dans le temps, tel qu’évalué par le test de Shapiro-Wilk (p > 0,05).
Notez que, si la taille de votre échantillon est supérieure à 50, le graphique de normalité QQ plot est préféré parce qu’avec des échantillons de plus grande taille, le test de Shapiro-Wilk devient très sensible même à un écart mineur par rapport à la distribution normale.
Le graphique QQ plot dessine la corrélation entre une donnée définie et la distribution normale. Créer des QQ plots pour chaque point dans le temps:
ggqqplot(selfesteem, "score", facet.by = "time")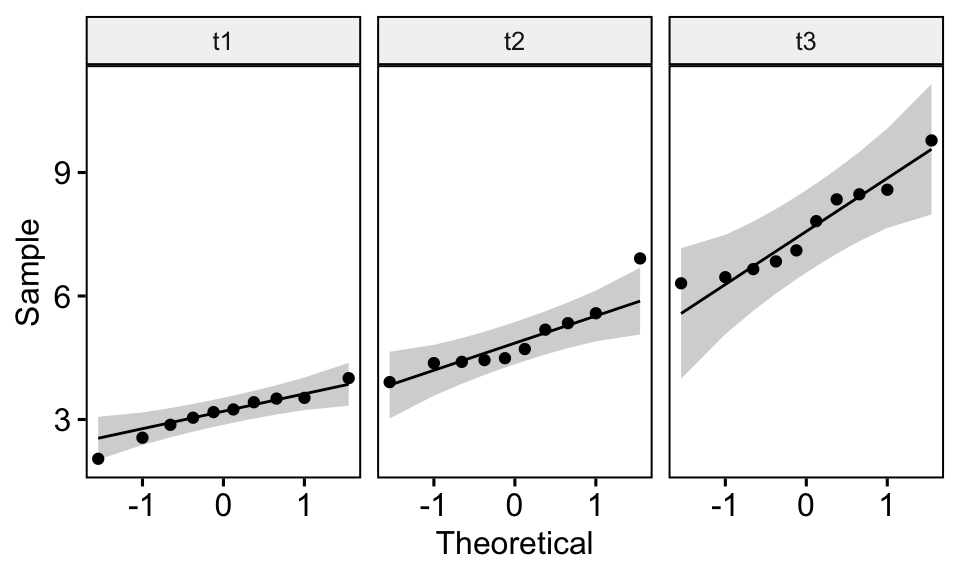
D’après le graphique ci-dessus, comme tous les points se situent approximativement le long de la ligne de référence, nous pouvons supposer une normalité.
Hypothèse de sphéricité
Comme mentionné dans les sections précédentes, l’hypothèse de sphéricité sera automatiquement vérifiée lors du calcul du test ANOVA en utilisant la fonction R anova_test() [package rstatix]. Le test de Mauchly est utilisé en interne pour évaluer l’hypothèse de sphéricité.
En utilisant la fonction get_anova_table() [rstatix] pour extraire la table ANOVA, la correction de sphéricité de Greenhouse-Geisser est automatiquement appliquée aux facteurs qui violent l’hypothèse de sphéricité.
Calculs
res.aov <- anova_test(data = selfesteem, dv = score, wid = id, within = time)
get_anova_table(res.aov)## ANOVA Table (type III tests)
##
## Effect DFn DFd F p p<.05 ges
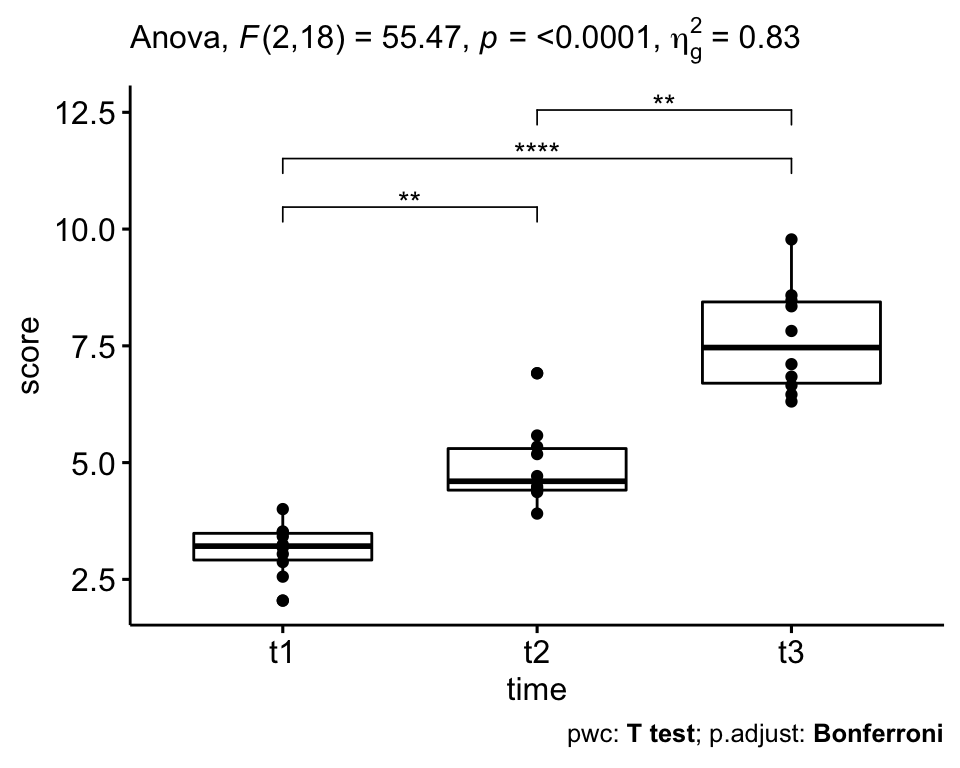
## 1 time 2 18 55.5 2.01e-08 * 0.829Le score de l’estime de soi était statistiquement significativement différent aux différents temps pendant le régime, F(2, 18) = 55,5, p < 0,0001, eta2[g] = 0,83.
où,
FIndique que nous comparons à une distribution F (test F) ;(2, 18)indique les degrés de liberté du numérateur (DFn) et du dénominateur (DFd), respectivement ;55.5indique la valeur statistique F obtenuepspécifie la p-valuegesest la taille de l’effet généralisé (taille de la variabilité due au facteur intra-sujets)
Tests post-hoc
Vous pouvez effectuer plusieurs tests t appariés par paires entre les niveaux du facteur intra-sujets (ici time). Les p-values sont ajustées à l’aide de la méthode de correction des tests multiples de Bonferroni.
# comparaisons par paires
pwc <- selfesteem %>%
pairwise_t_test(
score ~ time, paired = TRUE,
p.adjust.method = "bonferroni"
)
pwc## # A tibble: 3 x 10
## .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 score t1 t2 10 10 -4.97 9 0.000772 0.002 **
## 2 score t1 t3 10 10 -13.2 9 0.000000334 0.000001 ****
## 3 score t2 t3 10 10 -4.87 9 0.000886 0.003 **Toutes les différences par paires sont statistiquement significatives.
Rapporter
Nous pourrions rapporter le résultat comme suit:
Le score de l’estime de soi était statistiquement différent de façon significative aux différents points dans le temps, F(2, 18) = 55,5, p < 0,0001, eta-carré généralisé = 0,82.
Des analyses post-hoc avec ajustement de Bonferroni ont révélé que toutes les différences par paires, entre les différents temps, étaient statistiquement significatives (p <= 0,05).
# Visualisation : Boxplots avec p-values
pwc <- pwc %>% add_xy_position(x = "time")
bxp +
stat_pvalue_manual(pwc) +
labs(
subtitle = get_test_label(res.aov, detailed = TRUE),
caption = get_pwc_label(pwc)
)
ANOVA à deux facteurs sur mesures répétées
Préparation des données
Nous utiliserons le jeu de données selfesteem2` [package datarium] qui contient les mesures de l’estime de soi de 12 personnes incluses à 2 essais successifs de court terme (4 semaines) : essais contrôle (placebo) et essais avec un régime alimentaire spécial.
Chaque participant a effectué les deux essais. L’ordre des essais a été contrebalancé et un délai suffisant a été respecté entre les essais pour que les effets des essais précédents puissent se dissiper.
Le score d’estime de soi a été enregistré à trois moments : au début (t1), à mi-chemin (t2) et à la fin (t3) des essais.
La question est de savoir si ce traitement diététique à court terme peut induire une augmentation significative de l’estime de soi avec le temps. En d’autres termes, nous aimerions savoir s’il y a une interaction significative entre la diète et le temps sur le score de l’estime de soi.
L’ANOVA à deux facteurs sur mesures répétées peut être effectuée afin de déterminer s’il y a une interaction significative entre l’alimentation et le temps sur le score de l’estime de soi.
Charger et afficher une ligne aléatoire par groupe de traitement:
# Format large
set.seed(123)
data("selfesteem2", package = "datarium")
selfesteem2 %>% sample_n_by(treatment, size = 1)## # A tibble: 2 x 5
## id treatment t1 t2 t3
## <fct> <fct> <dbl> <dbl> <dbl>
## 1 4 ctr 92 92 89
## 2 10 Diet 90 93 95# Rassemblez les colonnes t1, t2 et t3 en format long.
# Convertir l'identifiant et le temps en facteurs
selfesteem2 <- selfesteem2 %>%
gather(key = "time", value = "score", t1, t2, t3) %>%
convert_as_factor(id, time)
# Inspecter quelques lignes aléatoires des données par groupes
set.seed(123)
selfesteem2 %>% sample_n_by(treatment, time, size = 1)## # A tibble: 6 x 4
## id treatment time score
## <fct> <fct> <fct> <dbl>
## 1 4 ctr t1 92
## 2 10 ctr t2 84
## 3 5 ctr t3 68
## 4 11 Diet t1 93
## 5 12 Diet t2 80
## 6 1 Diet t3 88Dans cet exemple, l’effet du “temps” sur l’estime de soi est notre variable focale, c’est-à-dire notre première cible.
Cependant, on pense que l’effet “temps” sera différent si le traitement est effectué ou non. Dans ce contexte, la variable “traitement” est considérée comme variable modératrice.
Statistiques descriptives
Regrouper les données par traitement (treatment) et temps (time), puis calculer quelques statistiques sommaires de la variable score: moyenne et sd (écart-type).
selfesteem2 %>%
group_by(treatment, time) %>%
get_summary_stats(score, type = "mean_sd")## # A tibble: 6 x 6
## treatment time variable n mean sd
## <fct> <fct> <chr> <dbl> <dbl> <dbl>
## 1 ctr t1 score 12 88 8.08
## 2 ctr t2 score 12 83.8 10.2
## 3 ctr t3 score 12 78.7 10.5
## 4 Diet t1 score 12 87.6 7.62
## 5 Diet t2 score 12 87.8 7.42
## 6 Diet t3 score 12 87.7 8.14Visualisation
Créez des boxplots du score colorée par groupes de traitement:
bxp <- ggboxplot(
selfesteem2, x = "time", y = "score",
color = "treatment", palette = "jco"
)
bxp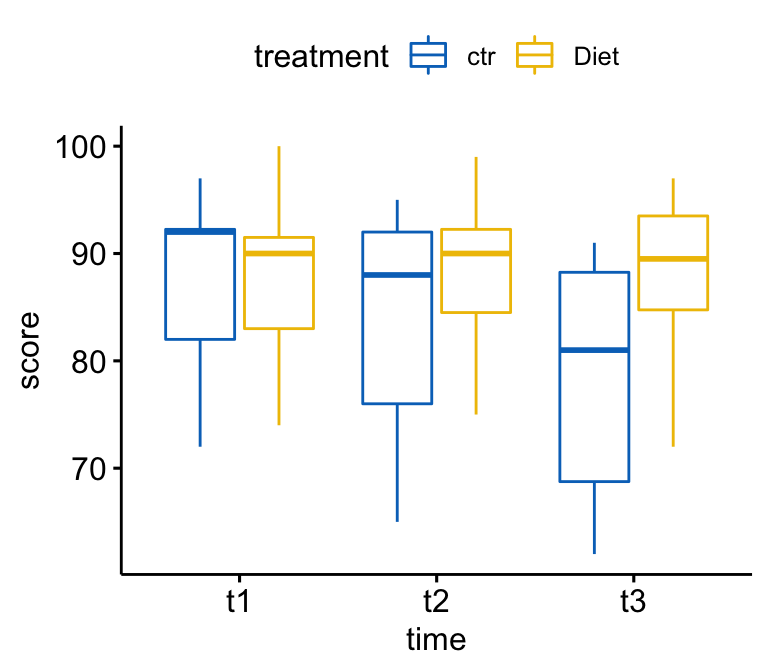
Vérifier les hypothèses
Valeurs aberrantes
selfesteem2 %>%
group_by(treatment, time) %>%
identify_outliers(score)## [1] treatment time id score is.outlier is.extreme
## <0 rows> (or 0-length row.names)Il n’y avait pas de valeurs extrêmes aberrantes.
Hypothèse de normalité
Calculer le test de Shapiro-Wilk pour chaque combinaison de niveaux des facteurs:
selfesteem2 %>%
group_by(treatment, time) %>%
shapiro_test(score)## # A tibble: 6 x 5
## treatment time variable statistic p
## <fct> <fct> <chr> <dbl> <dbl>
## 1 ctr t1 score 0.828 0.0200
## 2 ctr t2 score 0.868 0.0618
## 3 ctr t3 score 0.887 0.107
## 4 Diet t1 score 0.919 0.279
## 5 Diet t2 score 0.923 0.316
## 6 Diet t3 score 0.886 0.104Le score d’estime de soi était normalement distribué à chaque point dans le temps (p > 0,05), sauf pour le traitement ctr à t1, tel qu’évalué par le test de Shapiro-Wilk.
Créer un QQ plot pour chaque cellule du plan:
ggqqplot(selfesteem2, "score", ggtheme = theme_bw()) +
facet_grid(time ~ treatment, labeller = "label_both")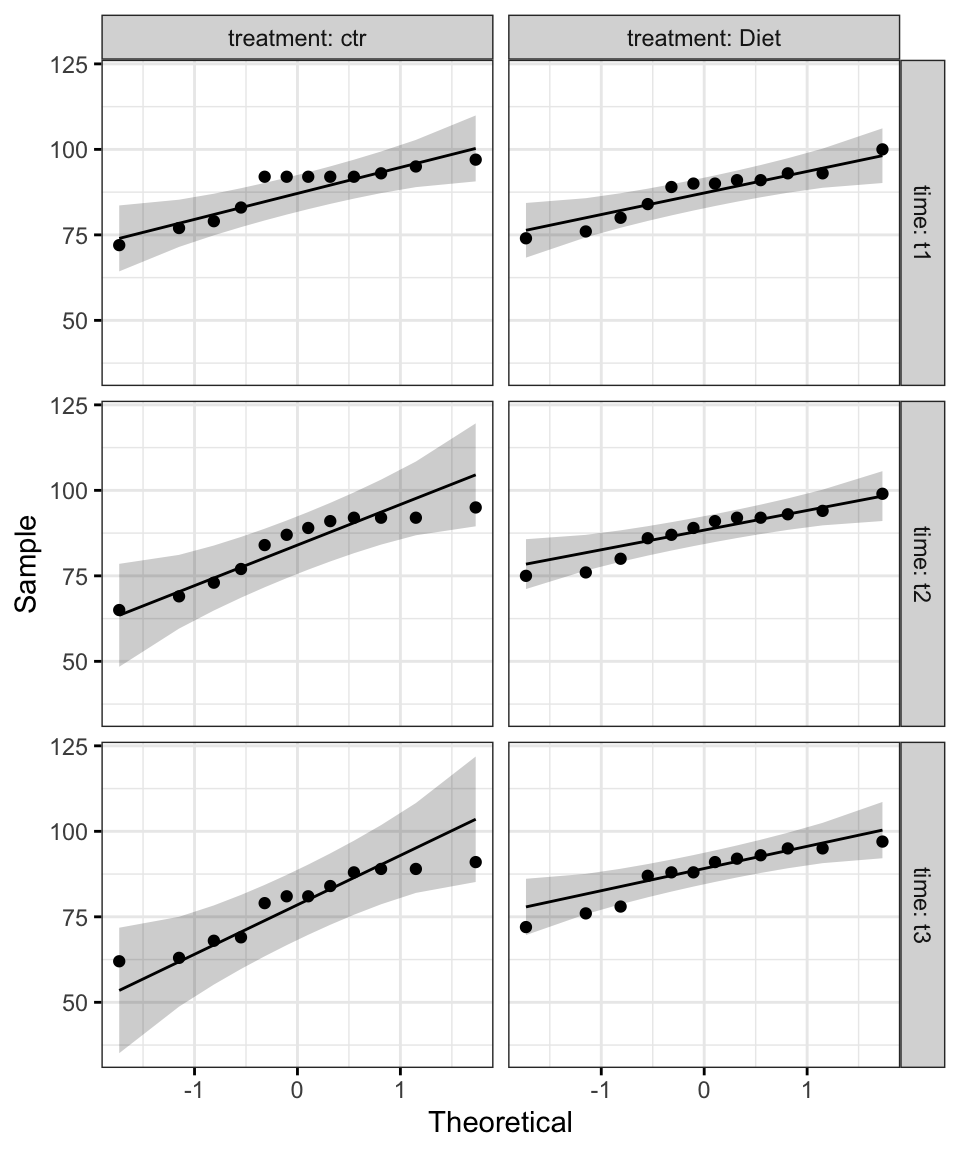
D’après le graphique ci-dessus, comme tous les points se situent approximativement le long de la ligne de référence, nous pouvons supposer une normalité.
Calculs
res.aov <- anova_test(
data = selfesteem2, dv = score, wid = id,
within = c(treatment, time)
)
get_anova_table(res.aov)## ANOVA Table (type III tests)
##
## Effect DFn DFd F p p<.05 ges
## 1 treatment 1.00 11.0 15.5 2.00e-03 * 0.059
## 2 time 1.31 14.4 27.4 5.03e-05 * 0.049
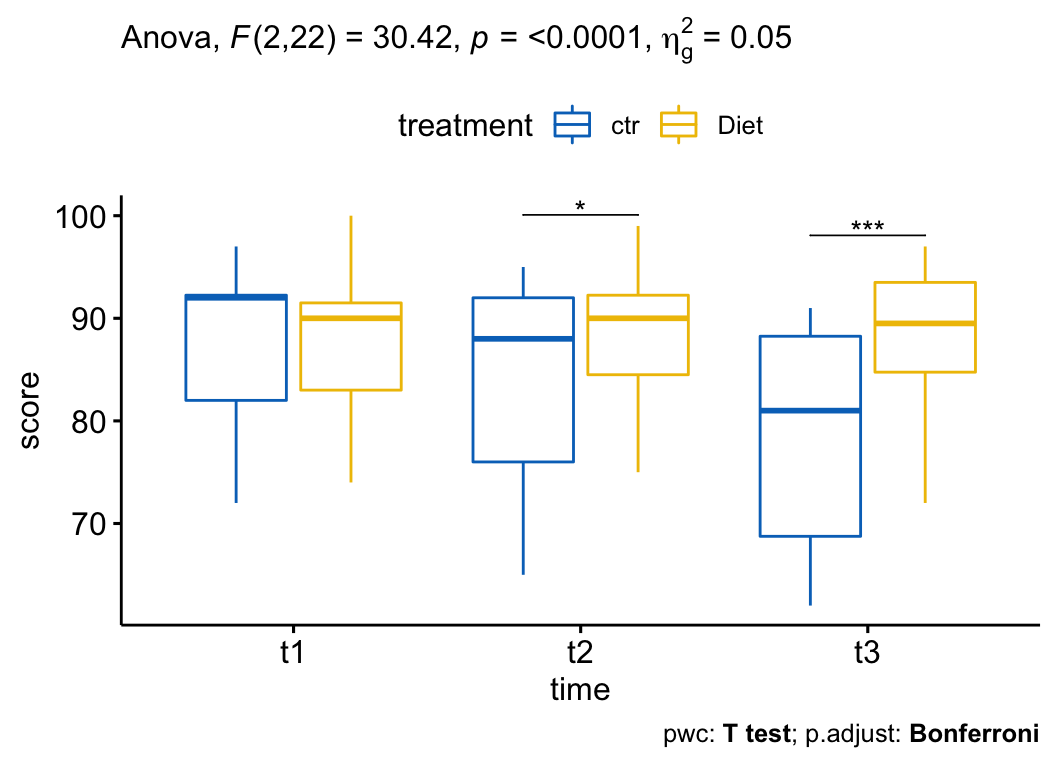
## 3 treatment:time 2.00 22.0 30.4 4.63e-07 * 0.050Il existe une interaction statistiquement significatives entre le traitement et le temps, F(2, 22) = 30,4, p < 0,0001.
Tests post-hoc
Une interaction significative à deux facteurs indique que l’impact d’un facteur (p. ex., le traitement) sur la variable-réponse (p. ex., l’estime de soi) dépend du niveau de l’autre facteur (p. ex., le temps) (et vice versa). Ainsi, vous pouvez décomposer une interaction significative, à deux facteurs, en:
- Effet principal : exécuter le modèle à un facteur avec la première variable (facteur A) à chaque niveau de la deuxième variable (facteur B),
- Comparaisons par paires : si l’effet principal est significatif, effectuez plusieurs comparaisons par paires pour déterminer quels groupes sont différents.
Dans le cas d’une interaction à deux facteurs non significative, vous devez déterminer si vous avez des effets principaux statistiquement significatifs dans le résultat de l’ANOVA.
Procédure pour une interaction significative à deux facteurs
Effet du traitement. Dans notre exemple, nous analyserons l’effet du traitement sur l’estime de soi à chaque instant.
Notez que la variable treatment n’a que deux niveaux (“ctr” et “Diet”) ; ainsi, le test ANOVA et le test t apparié donneront les mêmes p-values.
# Effet du traitement à chaque instant
one.way <- selfesteem2 %>%
group_by(time) %>%
anova_test(dv = score, wid = id, within = treatment) %>%
get_anova_table() %>%
adjust_pvalue(method = "bonferroni")
one.way## # A tibble: 3 x 9
## time Effect DFn DFd F p `p<.05` ges p.adj
## <fct> <chr> <dbl> <dbl> <dbl> <dbl> <chr> <dbl> <dbl>
## 1 t1 treatment 1 11 0.376 0.552 "" 0.000767 1
## 2 t2 treatment 1 11 9.03 0.012 * 0.052 0.036
## 3 t3 treatment 1 11 30.9 0.00017 * 0.199 0.00051# Comparaisons par paires entre les groupes de traitement
pwc <- selfesteem2 %>%
group_by(time) %>%
pairwise_t_test(
score ~ treatment, paired = TRUE,
p.adjust.method = "bonferroni"
)
pwc## # A tibble: 3 x 11
## time .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 t1 score ctr Diet 12 12 0.613 11 0.552 0.552 ns
## 2 t2 score ctr Diet 12 12 -3.00 11 0.012 0.012 *
## 3 t3 score ctr Diet 12 12 -5.56 11 0.00017 0.00017 ***Si l’on considère la p-value corrigée de Bonferroni (p.adj), on peut voir que l’effet principal du traitement n’était pas significatif à t1 (p = 1). Elle devient significative à t2 (p = 0,036) et t3 (p = 0,00051).
Les comparaisons par paires montrent que le score moyen d’estime de soi était significativement différent entre le groupe ctr et le groupe Diet à t2 (p = 0,12) et t3 (p = 0,00017) mais pas à t1 (p = 0,55).
Effet du temps. Notez qu’il est également possible d’effectuer la même analyse pour l’option time variable at each level of treatment. Vous n’avez pas nécessairement besoin de faire cette analyse.
Le code R:
# Effet du temps à chaque niveau de traitement
one.way2 <- selfesteem2 %>%
group_by(treatment) %>%
anova_test(dv = score, wid = id, within = time) %>%
get_anova_table() %>%
adjust_pvalue(method = "bonferroni")
# Comparaisons par paires entre les points dans le temps
pwc2 <- selfesteem2 %>%
group_by(treatment) %>%
pairwise_t_test(
score ~ time, paired = TRUE,
p.adjust.method = "bonferroni"
)
pwc2Après avoir exécuté le code R ci-dessus, vous pouvez voir que l’effet du temps (time) n’est significatif que pour l’essai contrôle, F(2, 22) = 39.7, p < 0.0001. Les comparaisons par paires montrent que toutes les comparaisons entre les différents temps étaient statistiquement significatives pour l’essai contrôle.
Procédure pour une interaction non significative à deux facteurs
Si l’interaction n’est pas significative, il faut interpréter les principaux effets pour chacune des deux variables: treatment et time. Un effet principal significatif peut être suivi par des comparaisons par paires.
Dans notre exemple (voir tableau ANOVA dans res.aov), il y avait un effet principal statistiquement significatif du traitement (F(1, 11) = 15,5, p = 0,002) et du temps (F(2, 22) = 27,4, p < 0,0001) sur le score de l’estime de soi.
Comparaisons par paires à l’aide du t-test apparié:
# comparaisons pour la variable traitement
selfesteem2 %>%
pairwise_t_test(
score ~ treatment, paired = TRUE,
p.adjust.method = "bonferroni"
)
# comparaisons pour la variable time
selfesteem2 %>%
pairwise_t_test(
score ~ time, paired = TRUE,
p.adjust.method = "bonferroni"
)Toutes les comparaisons par paires sont significatives.
Rapporter
Nous pourrions rapporter le résultat comme suit:
Une ANOVA à deux facteurs sur mesures répétées a été effectuée pour évaluer l’effet de différents traitements diététiques sur l’estime de soi au fil du temps.
Il y avait une interaction statistiquement significative entre le traitement et le temps sur l’estime de soi, F(2, 22) = 30,4, p < 0,0001. Par conséquent, l’effet de la variable treatment a été analysé à chaque point de time. Les p-values ont été ajustées à l’aide de la méthode de correction des tests multiples de Bonferroni. L’effet du traitement était significatif à t2 (p = 0,036) et t3 (p = 0,00051), mais pas au temps t1 (p = 1).
Des comparaisons par paires, utilisant le test t apparié, montrent que le score moyen d’estime de soi était significativement différent entre l’essai ctr et l’essai Diet aux temps t2 (p = 0,012) et t3 (p = 0,00017) mais pas à t1 (p = 0,55).
# Visualisation : Boxplots avec p-values
pwc <- pwc %>% add_xy_position(x = "time")
bxp +
stat_pvalue_manual(pwc, tip.length = 0, hide.ns = TRUE) +
labs(
subtitle = get_test_label(res.aov, detailed = TRUE),
caption = get_pwc_label(pwc)
)
ANOVA à trois facteurs sur mesures répétées
Préparation des données
Nous utiliserons le jeu de données weightloss [package datarium]. Dans cette étude, un chercheur voulait évaluer les effets de l’alimentation et de l’exercice sur la perte de poids chez 10 personnes sédentaires.
Les participants ont été inclus dans quatre essais : (1) pas de régime et pas d’exercices ; (2) régime seulement ; (3) exercices seulement ; et (4) régime et exercices combinés.
Chaque participant a effectué les quatre essais. L’ordre des essais a été contrebalancé et un délai suffisant a été respecté entre les essais pour que les effets des essais précédents puissent se dissiper.
Chaque essai a duré neuf semaines et le score de perte de poids a été mesuré au début (t1), au milieu (t2) et à la fin (t3) de chaque essai.
L’ANOVA à trois facteurs, sur mesures répétées, peut être effectuée afin de déterminer s’il y a une interaction significative entre le régime alimentaire, les exercices et le temps sur le score de perte de poids.
Charger les données et afficher quelques lignes aléatoires par groupes:
# Format large
set.seed(123)
data("weightloss", package = "datarium")
weightloss %>% sample_n_by(diet, exercises, size = 1)## # A tibble: 4 x 6
## id diet exercises t1 t2 t3
## <fct> <fct> <fct> <dbl> <dbl> <dbl>
## 1 4 no no 11.1 9.5 11.1
## 2 10 no yes 10.2 11.8 17.4
## 3 5 yes no 11.6 13.4 13.9
## 4 11 yes yes 12.7 12.7 15.1# Rassemblez les colonnes t1, t2 et t3 en format long.
# Convertir l'identifiant et le temps en facteurs
weightloss <- weightloss %>%
gather(key = "time", value = "score", t1, t2, t3) %>%
convert_as_factor(id, time)
# Inspecter quelques lignes aléatoires des données par groupes
set.seed(123)
weightloss %>% sample_n_by(diet, exercises, time, size = 1)## # A tibble: 12 x 5
## id diet exercises time score
## <fct> <fct> <fct> <fct> <dbl>
## 1 4 no no t1 11.1
## 2 10 no no t2 10.7
## 3 5 no no t3 12.3
## 4 11 no yes t1 10.2
## 5 12 no yes t2 13.2
## 6 1 no yes t3 15.8
## # … with 6 more rowsDans cet exemple, l’effet du “temps” est notre variable focale, c’est-à-dire notre première cible.
On pense que l’effet du “temps” sur le score de perte de poids dépendra de deux autres facteurs, “régime” et “exercices”, que l’on appelle variables modératrices.
Statistiques descriptives
Regroupez les données par diet, exercises et time, puis calculez quelques statistiques sommaires de la variable score : moyenne et sd (écart type)
weightloss %>%
group_by(diet, exercises, time) %>%
get_summary_stats(score, type = "mean_sd")## # A tibble: 12 x 7
## diet exercises time variable n mean sd
## <fct> <fct> <fct> <chr> <dbl> <dbl> <dbl>
## 1 no no t1 score 12 10.9 0.868
## 2 no no t2 score 12 11.6 1.30
## 3 no no t3 score 12 11.4 0.935
## 4 no yes t1 score 12 10.8 1.27
## 5 no yes t2 score 12 13.4 1.01
## 6 no yes t3 score 12 16.8 1.53
## # … with 6 more rowsVisualisation
Créer des box plots:
bxp <- ggboxplot(
weightloss, x = "exercises", y = "score",
color = "time", palette = "jco",
facet.by = "diet", short.panel.labs = FALSE
)
bxp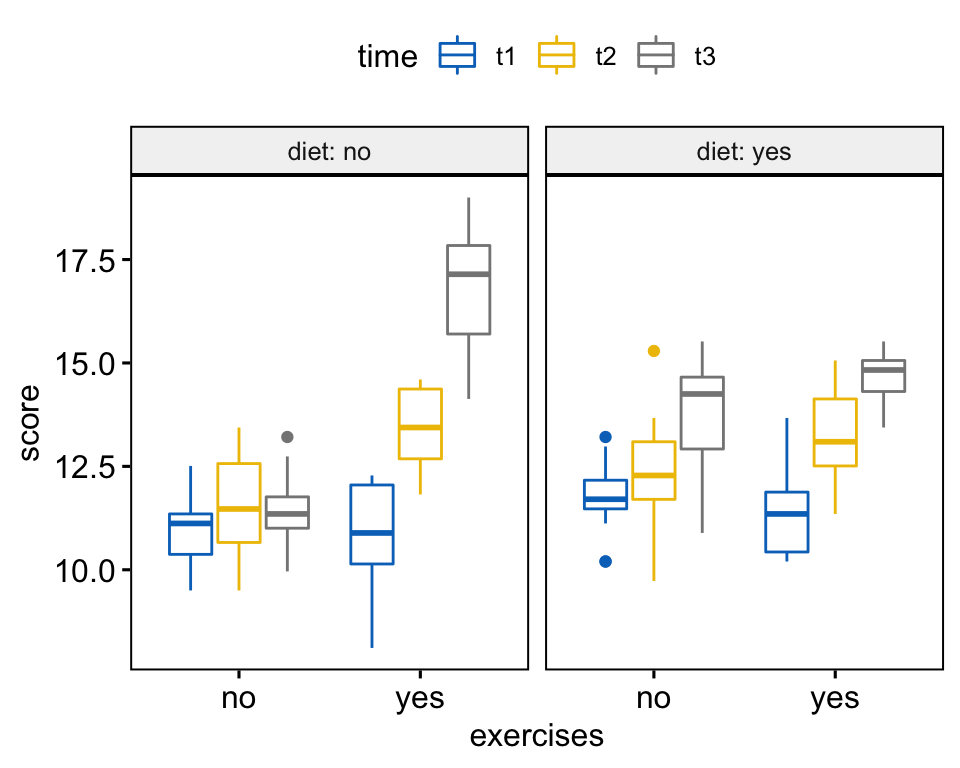
Vérifier les hypothèses
Valeurs aberrantes
weightloss %>%
group_by(diet, exercises, time) %>%
identify_outliers(score)## # A tibble: 5 x 7
## diet exercises time id score is.outlier is.extreme
## <fct> <fct> <fct> <fct> <dbl> <lgl> <lgl>
## 1 no no t3 2 13.2 TRUE FALSE
## 2 yes no t1 1 10.2 TRUE FALSE
## 3 yes no t1 3 13.2 TRUE FALSE
## 4 yes no t1 4 10.2 TRUE FALSE
## 5 yes no t2 10 15.3 TRUE FALSEIl n’y avait pas de valeurs extrêmes aberrantes.
Hypothèse de normalité
Calculer le test de Shapiro-Wilk pour chaque combinaison de niveaux des facteurs:
weightloss %>%
group_by(diet, exercises, time) %>%
shapiro_test(score)## # A tibble: 12 x 6
## diet exercises time variable statistic p
## <fct> <fct> <fct> <chr> <dbl> <dbl>
## 1 no no t1 score 0.917 0.264
## 2 no no t2 score 0.957 0.743
## 3 no no t3 score 0.965 0.851
## 4 no yes t1 score 0.922 0.306
## 5 no yes t2 score 0.912 0.229
## 6 no yes t3 score 0.953 0.674
## # … with 6 more rowsLe score de perte de poids était normalement distribué, tel qu’évalué par le test de normalité de Shapiro-Wilk (p > .05).
Créer un QQ plot pour chaque cellule du plan:
ggqqplot(weightloss, "score", ggtheme = theme_bw()) +
facet_grid(diet + exercises ~ time, labeller = "label_both")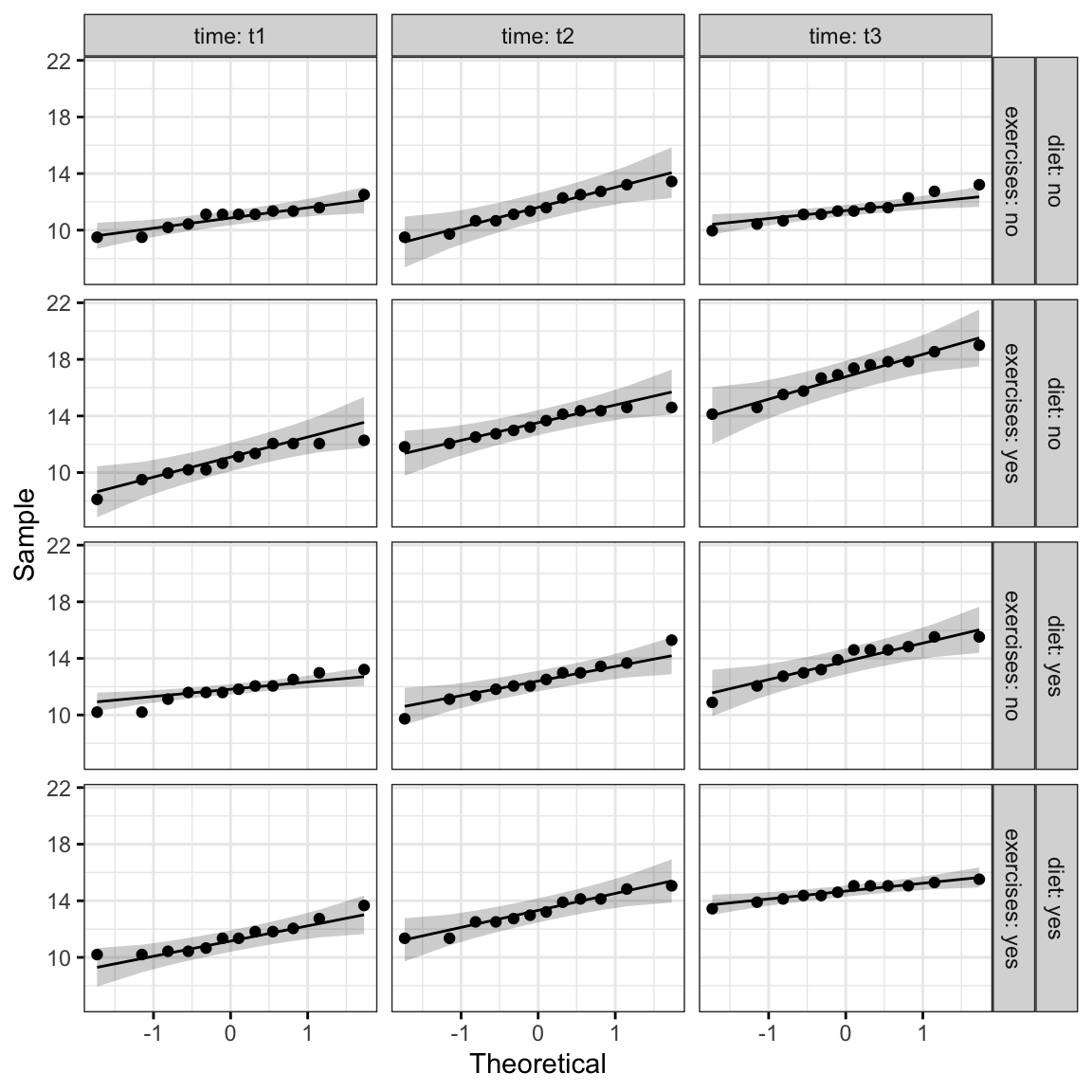
D’après le graphique ci-dessus, comme tous les points se situent approximativement le long de la ligne de référence, nous pouvons supposer une normalité.
Calculs
res.aov <- anova_test(
data = weightloss, dv = score, wid = id,
within = c(diet, exercises, time)
)
get_anova_table(res.aov)## ANOVA Table (type III tests)
##
## Effect DFn DFd F p p<.05 ges
## 1 diet 1.00 11.0 6.021 3.20e-02 * 0.028
## 2 exercises 1.00 11.0 58.928 9.65e-06 * 0.284
## 3 time 2.00 22.0 110.942 3.22e-12 * 0.541
## 4 diet:exercises 1.00 11.0 75.356 2.98e-06 * 0.157
## 5 diet:time 1.38 15.2 0.603 5.01e-01 0.013
## 6 exercises:time 2.00 22.0 20.826 8.41e-06 * 0.274
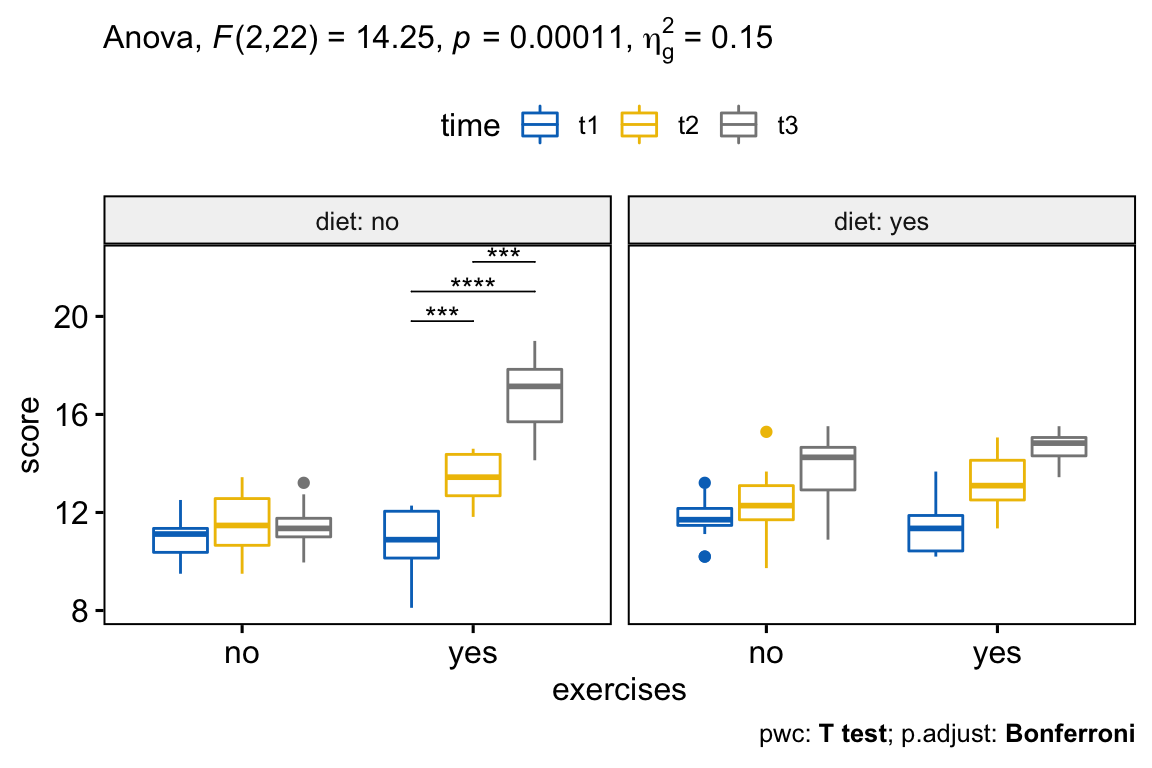
## 7 diet:exercises:time 2.00 22.0 14.246 1.07e-04 * 0.147D’après les résultats ci-dessus, on peut voir qu’il existe une interaction, à trois facteurs, statistiquement significatives entre le régime alimentaire, les exercices et le temps, F(2, 22) = 14,24, p = 0,00011.
Notez que, si l’interaction à trois facteurs n’est pas statistiquement significative, vous devez consulter les interactions à deux facteurs dans le résultat.
Dans notre exemple, il y avait des interactions statistiquement significatives: diet:exercises (p < 0,0001) et exercises:time (p < 0,0001). L’interaction diet:time (régime:temps) n’était pas statistiquement significative (p = 0,5).
Tests post-hoc
S’il y a un effet significatif d’interaction à trois facteurs, vous pouvez le décomposer en:
- Interaction à deux facteurs : exécuter l’interaction, à deux facteurs, à chaque niveau de la troisième variable,
- Effet principal : exécuter un modèle, à un facteur, à chaque niveau de la deuxième variable, et
- Comparaisons par paires : effectuer des comparaisons par paires ou d’autres comparaisons post-hoc si nécessaire.
Calculer l’interaction à deux facteurs
Vous êtes libre de décider des deux variables qui formeront les interactions à deux facteurs et quelle variable agira comme troisième variable (modératrice). Dans le code R suivant, nous avons considéré l’interaction exercices*temps à chaque niveau de diet.
Regroupez les données par diet (régime) et analysez l’interaction entre exercises et time:
# ANOVA à deux facteurs à chaque niveau de diet
two.way <- weightloss %>%
group_by(diet) %>%
anova_test(dv = score, wid = id, within = c(exercises, time))
two.way## # A tibble: 2 x 2
## diet anova
## <fct> <list>
## 1 no <anov_tst>
## 2 yes <anov_tst># Extraire le tableau anova
get_anova_table(two.way)## # A tibble: 6 x 8
## diet Effect DFn DFd F p `p<.05` ges
## <fct> <chr> <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 no exercises 1 11 72.8 3.53e- 6 * 0.526
## 2 no time 2 22 71.7 2.32e-10 * 0.587
## 3 no exercises:time 2 22 28.9 6.92e- 7 * 0.504
## 4 yes exercises 1 11 13.4 4.00e- 3 * 0.038
## 5 yes time 2 22 20.5 9.30e- 6 * 0.49
## 6 yes exercises:time 2 22 2.57 9.90e- 2 "" 0.06Il y a une interaction statistiquement significative entre les exercices et le temps pour l’essai “diet no” (sans régime), F(2, 22) = 28,9, p < 0,0001, mais pas pour l’essai “diet yes” (avec régime), F(2, 22) = 2,6, p = 0,099.
Notez qu’il est recommandé d’ajuster la p-value pour les tests multiples. Une approche courante consiste à appliquer un ajustement de Bonferroni pour réduire le niveau auquel vous déclarez la significativité statistique.
Pour ce faire, divisez le niveau actuel auquel vous déclarez une significativité statistique (p < 0,05) par le nombre d’interactions, à deux facteurs, que vous analysez (c.-à-d. 2).
Ainsi, vous ne déclarez une interaction comme statistiquement significative que lorsque p < 0,025 (c.-à-d. p < 0,05/2). En appliquant cela à notre exemple actuel, nous tirerions toujours les mêmes conclusions.
Calculer l’effet principal
Une interaction à deux facteurs statistiquement significative peut être suivie par une analyse des effets principaux.
Dans notre exemple, vous pourriez donc étudier l’effet du temps (time) sur le score de la perte de poids (score) à chaque niveau de la variable exercices ou étudier l’effet de la variable exercices à chaque niveau de time.
Vous n’aurez qu’à tenir compte du résultat des analyses des effets principaux de l’essai “diet no” (pas de régime), car il s’agit de la seule interaction, à deux facteur, statistiquement significative (voir section précédente).
Regrouper les données par diet and exercises, et analyser l’effet principal simple de time. L’ajustement de Bonferroni sera considéré, ce qui conduit à un seuil de significativité statistique p < 0,025 (soit 0,05 divisé par le nombre de tests (ici 2) considérés pour l’essai “diet:no”.
# Effet du temps à chaque cellule de régime x exercices
time.effect <- weightloss %>%
group_by(diet, exercises) %>%
anova_test(dv = score, wid = id, within = time)
time.effect## # A tibble: 4 x 3
## diet exercises anova
## <fct> <fct> <list>
## 1 no no <anov_tst>
## 2 no yes <anov_tst>
## 3 yes no <anov_tst>
## 4 yes yes <anov_tst># Extraire le tableau anova
get_anova_table(time.effect) %>%
filter(diet == "no")## # A tibble: 2 x 9
## diet exercises Effect DFn DFd F p `p<.05` ges
## <fct> <fct> <chr> <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 no no time 2 22 1.32 2.86e- 1 "" 0.075
## 2 no yes time 2 22 78.8 9.30e-11 * 0.801Il y avait un effet principal statistiquement significatif du temps sur le score de perte de poids pour le groupe “diet:no,exercises:yes” (p < 0,0001), mais pas pour les cas où ni régime ni exercices n’ont été effectués (p = 0,286).
Calculer les comparaisons entre groupes
Un effet principal statistiquement significatif peut être suivi de multiples comparaisons par paires pour déterminer quelles moyennes de groupe sont différentes.
Regroupez les données par diet et exercices, et effectuez des comparaisons par paires entre les points de time en appliquant l’ajustement de Bonferroni:
# Comparaisons par paires
pwc <- weightloss %>%
group_by(diet, exercises) %>%
pairwise_t_test(score ~ time, paired = TRUE, p.adjust.method = "bonferroni") %>%
select(-df, -statistic) # Supprimer les détails
# Afficher les résultats de la comparaison pour les groupes `diet:no,exercises:yes`
pwc %>% filter(diet == "no", exercises == "yes") %>%
select(-p) # enlever les colonnes p## # A tibble: 3 x 9
## diet exercises .y. group1 group2 n1 n2 p.adj p.adj.signif
## <fct> <fct> <chr> <chr> <chr> <int> <int> <dbl> <chr>
## 1 no yes score t1 t2 12 12 0.000741 ***
## 2 no yes score t1 t3 12 12 0.0000000121 ****
## 3 no yes score t2 t3 12 12 0.000257 ***Dans le tableau de comparaison par paires ci-dessus, nous ne nous intéressons qu’aux comparaisons pour les groupes “diet:no,exercises:yes”. Dans notre exemple, il existe trois combinaisons possibles de différences de groupe. Nous pourrions présenter les résultats de la comparaison par paires comme suit.
Toutes les comparaisons par paires ont été effectuées entre les différents temps pour l’essai “diet:no,exercises:yes”. L’ajustement Bonferroni a été appliqué. Le score moyen de perte de poids était significativement différent dans toutes les comparaisons à tous les temps lorsque des exercices sont effectués (p < 0,05).
Rapporter
L’ANOVA à trois facteurs sur mesures répétées a été effectuée pour évaluer les effets de l’alimentation, de l’exercice et du temps sur la perte de poids. Il y avait une interaction, à trois facteurs, statistiquement significative entre l’alimentation, les exercices et le temps, F(2, 22) = 14,2, p = 0,00011.
Pour les interactions à deux facteurs et les effets principaux, un ajustement de Bonferroni a été appliqué conduisant à un seuil de significativité statistique accepté de p < 0,025.
Il y a une interaction statistiquement significative entre les exercices et le temps pour l’essai “diet no” (sans régime), F(2, 22) = 28,9, p < 0,0001, mais pas pour l’essai “diet yes” (avec régime), F(2, 22) = 2,6, p = 0,099.
Il y avait un effet principal statistiquement significatif du temps sur le score de perte de poids pour l’essai “diet:no,exercises:yes” (p < 0.0001), mais pas pour les cas où ni le régime ni les exercices n’étaient effectués (p = 0.286).
Toutes les comparaisons par paires ont été effectuées entre les différents temps pour l’essai “diet:no,exercises:yes” avec un ajustement de Bonferroni appliqué. Le score moyen de perte de poids était significativement différent dans toutes les comparaisons à tous les temps lorsque des exercices sont effectués (p < 0,05).
# Visualisation : Boxplots avec p-values
pwc <- pwc %>% add_xy_position(x = "exercises")
pwc.filtered <- pwc %>%
filter(diet == "no", exercises == "yes")
bxp +
stat_pvalue_manual(pwc.filtered, tip.length = 0, hide.ns = TRUE) +
labs(
subtitle = get_test_label(res.aov, detailed = TRUE),
caption = get_pwc_label(pwc)
)
Résumé
Ce chapitre décrit comment calculer, interpréter et rapporter l’ANOVA sur mesures répétées dans R. Nous expliquons également les hypothèses faites par les tests ANOVA sur mesures répétées et fournissons des exemples pratiques de codes R pour vérifier si les hypothèses des tests sont respectées.
Version:
 English
English



Bonjour,
Tout d’abord merci beaucoup pour ce site internet, il est très instructif!
J’ai quelques problèmes avec l’étape de l’anova_test. Ce message d’erreur apparait alors que je n’ai aucune donnée manquante.
Error in lm.fit(x, y, offset = offset, singular.ok = singular.ok, …) :
0 (non-NA) cases
Avez-vous une idée pour la résolution de ce problème?
Je vous remercie pour le commentaire positif.
L’erreur obtenue ressemble à celle décrite à https://github.com/kassambara/rstatix/issues/55. Elle a été corrigée dans la dernière version de development. Vous pouvez l’installer comme suit: devtools::install_github(« kassambara/rstatix »).
Bonjour,
Merci d’avoir développé ce tutoriel. Il est très utile et pratique. Bravo!
Malheureusement, je rencontre toujours le même message d’erreur:
Error in lm.fit(x, y, offset = offset, singular.ok = singular.ok, …) :
0 (non-NA) cases
Pourtant, j’avais suivi les pas que vous avez indiqué pour résoudre le problème. Cela n’a pas marché.
Merci par avance de votre réponse.
Bonjour,
Merci beaucoup pour votre site et la qualité des ressources que vous publiez.
Tout comme ‘Andréa Bellavance » l »erreur « Error in lm.fit(x, y, offset = offset, singular.ok = singular.ok, …) :
0 (non-NA) cases » survient des que j utilise une base de donnees en .csv.
Bien que le script fonctionne parfaitement avec le dataset de votre example.
Je n ai pourtant aucune donnees manquantes dans ma table:
sapply(Viralrep,function(x) sum(is.na(x)))
Sample_ID Tank Time_hpi Family Bag ORF38
0 0 0 0 0 0
J’utilise la version 4.0.4 de R et j’ai mis à jours rstatix comme vous l’indiquez ci-dessus.
Avez-vous une idée pour resoudre ce problème?
Je vous remercie par avance,
Bonjour,
Merci pour le travail fait. J’ai cependant un souci avec la syntaxe de la fonction anova_test. Veuillez m’aider s’il vous plait.
* Considérons une anova à deux facteurs sur mesures répétées suivant seulement un des facteurs (par exemple le temps). Comment se présenterait le code ?
Dans la commande suivante, je ne vois pas comment ça pourrait être pris en compte puisqu’aucun facteur n’est spécifié pour la répétition.
res.aov <- anova_test(
data = selfesteem2, dv = score, wid = id,
within = c(treatment, time)
)
* Plus généralement, j'aimerais bien comprendre les paramètres de cette fonction s'il vous plait.
* Une autre chose : quelle commande (ainsi que la syntaxe pour différents cas : mesures répétées suivant le facteur, un des facteurs et suivant les deux facteurs) me permet d'obtenir les coefficients du modèle ?… un peu comme avec la fonction lm() dans le cas de l'anova simple à 1 ou 2 facteurs
Bonjour,
merci beaucoup pour le tutoriel, très bien expliqué.
Je voudrais savoir, comment je fais si j’ai des facteurs INTER SUJET ? C’est à dire, des facteurs qui ne concernent pas la totalité des sujets (par ex, le sexe, male/female). Dans mon cas, les mêmes sujets ont été mesurés à quatre moments differents (facteur intra sujets = temps). Par contre, ils ont subi deux traitements differents (donc facteur inter sujet).
Merci pour votre aide!