Cet article décrit les bases pour calculer et ajouter des p-values aux graphiques ggplots basiques en utilisant les packages R rstatix et ggpubr.
Vous apprendrez à:
- Effectuer des comparaisons de moyennes par paires et ajouter les p-values sur des box plots et bar plots baiques.
- Afficher les p-values ajustées et les niveaux de significativité sur les graphiques
- Formater les étiquettes de p-values
- Spécifier manuellement la position y des étiquettes de p-values et raccourcir la largeur des crochets
Nous suivrons les étapes suivantes pour ajouter des niveaux de significativité sur un ggplot:
- Calculer facilement des tests statistiques (
t_test()ouwilcox_test()) en utilisant le packagerstatix - Auto-calculez les positions des étiquettes des p-values en utilisant la fonction
add_xy_position()[dans le package rstatix]. - Ajoutez les p-values au graphique en utilisant la fonction
stat_pvalue_manual()[dans le package ggpubr]. Les options clés suivantes sont illustrées dans certains des exemples:- L’option
bracket.nudge.yest utilisée pour monter ou descendre les crochets. - L’option
step.increaseest utilisée pour ajouter de l’espace entre les crochets. - L’option
vjustest utilisée pour ajuster verticalement la position des étiquettes des p-values
- L’option
Notez que, dans certains cas, les étiquettes de p-values sont partiellement cachées par la bordure supérieure du graphique. Dans ce cas, la fonction ggplot2 scale_y_continuous(expand = expansion(mult = c(0, 0.1))) peut être utilisée pour ajouter des espaces entre les étiquettes et la bordure supérieure du graphique. L’option mult = c(0, 0.1) indique que des espaces de 0% et 10% sont respectivement ajoutés en bas et en haut du graphique.
Sommaire:
Prérequis
Assurez-vous d’avoir installé les paquets R suivants:
ggpubrpour créer facilement des graphiques prêts à la publicationrstatixcontient des fonctions R facilitant les analyses statistiques.
Commencez par charger les packages requis suivants:
library(ggpubr)
library(rstatix)Préparation des données
# Transformer `dose` en variable factorielle
df <- ToothGrowth
df$dose <- as.factor(df$dose)
head(df, 3)## len supp dose
## 1 4.2 VC 0.5
## 2 11.5 VC 0.5
## 3 7.3 VC 0.5Comparer deux moyens
Pour comparer les moyennes de deux groupes, vous pouvez utiliser soit la fonction t_test() (paramétrique) ou wilcox_test() (non-paramétrique). Dans l’exemple suivant, le test t sera illustré.
Comparer deux groupes indépendants
Box plot avec p-values
# Test statistique
stat.test <- df %>%
t_test(len ~ supp) %>%
add_significance()
stat.test## # A tibble: 1 x 9
## .y. group1 group2 n1 n2 statistic df p p.signif
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <chr>
## 1 len OJ VC 30 30 1.92 55.3 0.0606 ns# Box plot avec p-values
bxp <- ggboxplot(df, x = "supp", y = "len", fill = "#00AFBB")
stat.test <- stat.test %>% add_xy_position(x = "supp")
bxp +
stat_pvalue_manual(stat.test, label = "p") +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.1)))
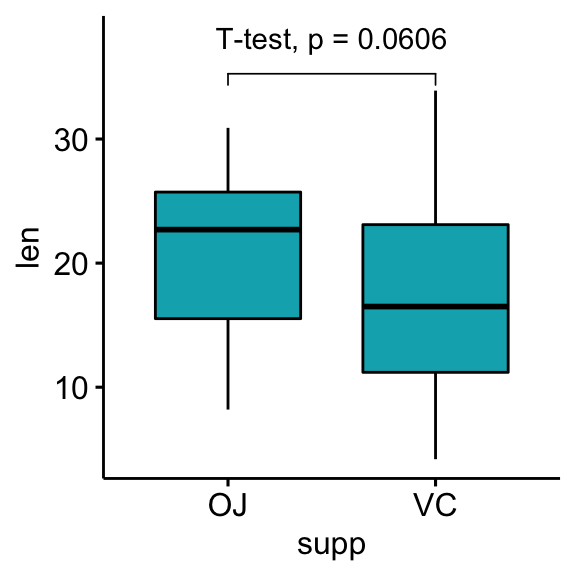
# Personnaliser les étiquettes de p-values en utilisant le format `glue`
# https://github.com/tidyverse/glue
bxp + stat_pvalue_manual(
stat.test, label = "T-test, p = {p}",
vjust = -1, bracket.nudge.y = 1
) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.15)))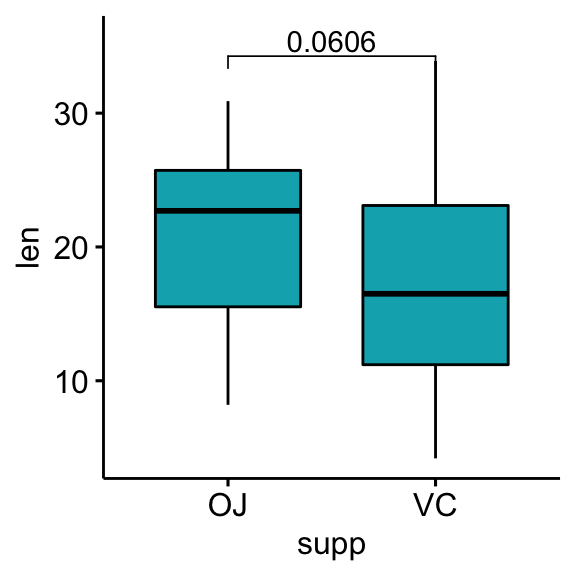
Données groupées
Regroupez les données en fonction de la variable dose et comparez ensuite les niveaux de la variable supp.
# Test statistique
stat.test <- df %>%
group_by(dose) %>%
t_test(len ~ supp) %>%
adjust_pvalue() %>%
add_significance("p.adj")
stat.test## # A tibble: 3 x 11
## dose .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 0.5 len OJ VC 10 10 3.17 15.0 0.00636 0.0127 *
## 2 1 len OJ VC 10 10 4.03 15.4 0.00104 0.00312 **
## 3 2 len OJ VC 10 10 -0.0461 14.0 0.964 0.964 ns# Box plot avec p-values
stat.test <- stat.test %>% add_xy_position(x = "supp")
bxp <- ggboxplot(df, x = "supp", y = "len", fill = "#00AFBB",
facet.by = "dose")
bxp +
stat_pvalue_manual(stat.test, label = "p.adj") +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.10)))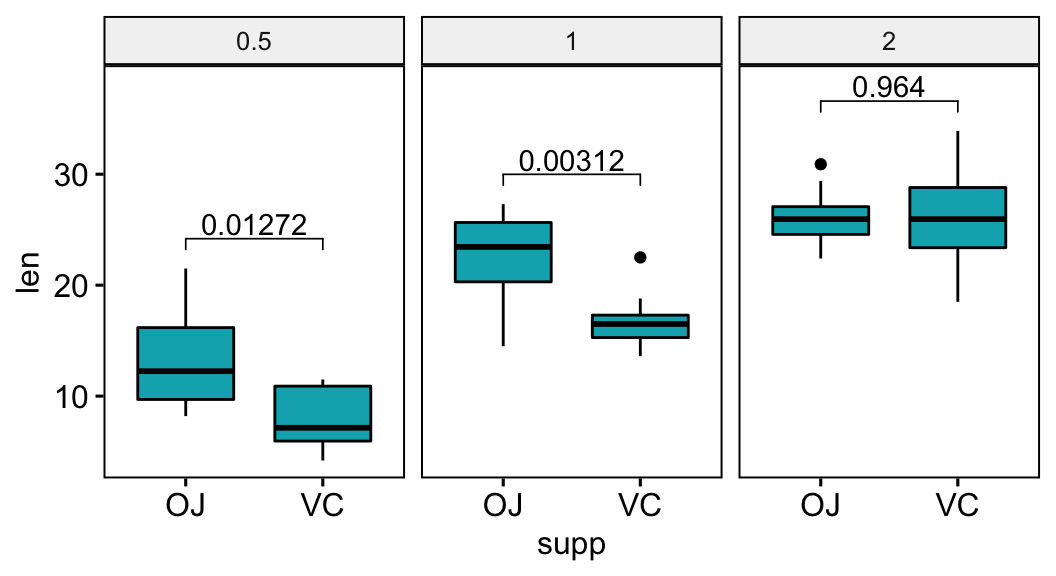
Afficher les p-values si elles sont significatives sinon afficher ns
Cette section décrit comment afficher les p-values lorsqu’elles sont significatives et montrer “ns” lorsque les p-values ne sont pas significatives.
# Ajouter une colonne d'étiquettes personnalisées
# indiquer les p-values ajustées si elles sont significatives, sinon "ns"
stat.test <- stat.test %>% add_xy_position(x = "supp")
stat.test$custom.label <- ifelse(stat.test$p.adj <= 0.05, stat.test$p.adj, "ns")
# Visualisation
bxp +
stat_pvalue_manual(stat.test, label = "custom.label") +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.10)))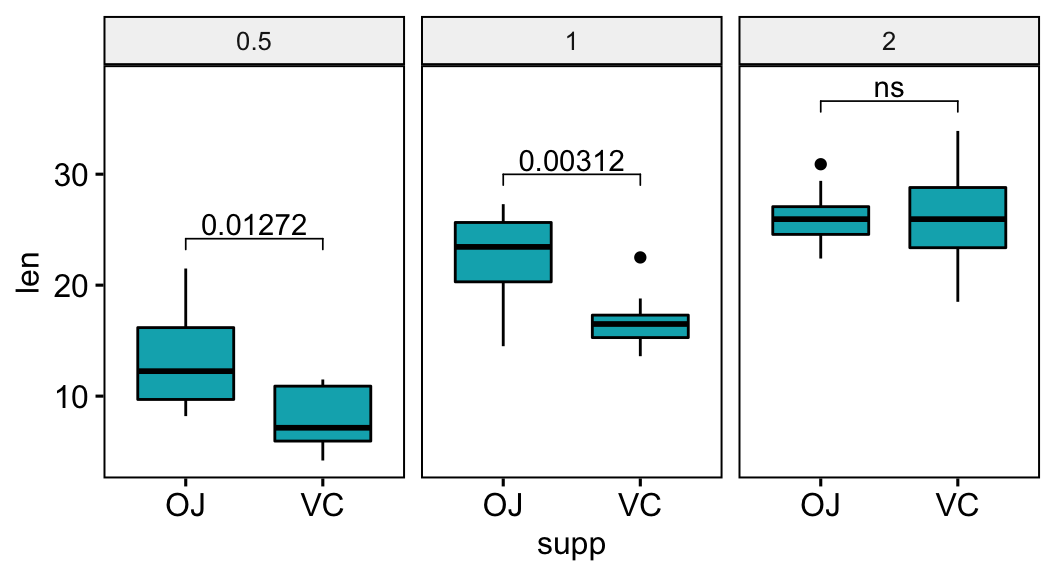
Formater les p-values en utilisant l’option de précision ou accuracy
Les p-values seront formatées en utilisant “<” et “>”.
stat.test <- stat.test %>% add_xy_position(x = "supp")
stat.test$p.format <- p_format(
stat.test$p.adj, accuracy = 0.01,
leading.zero = FALSE
)
# Visualisation
bxp +
stat_pvalue_manual(stat.test, label = "p.format") +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.10)))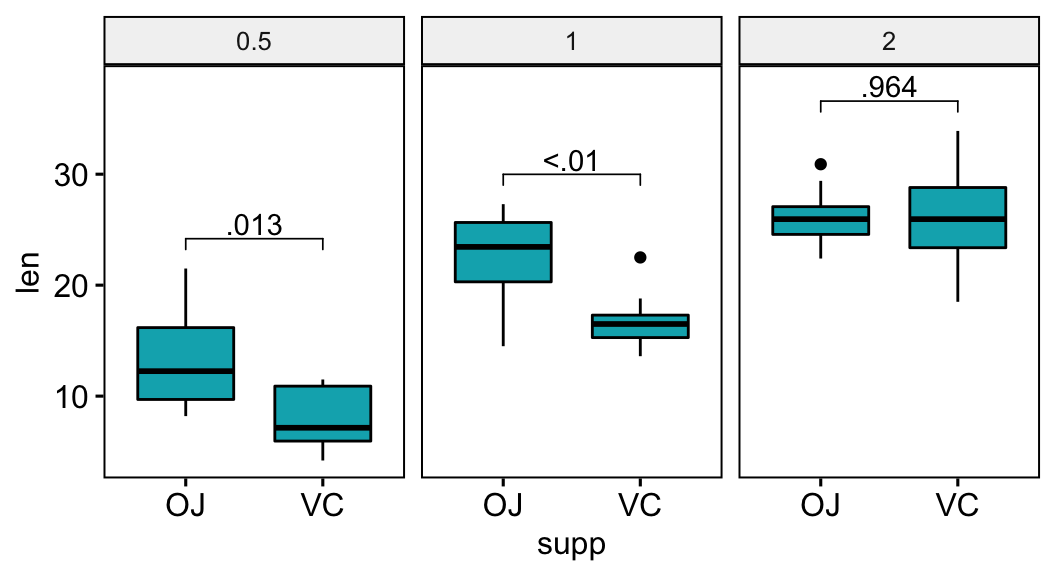
Formater les p-values en notation scientifique
# Convertir les p-values en format scientifique
stat.test <- stat.test %>% add_xy_position(x = "supp")
stat.test$p.scient <- format(stat.test$p.adj, scientific = TRUE)
bxp +
stat_pvalue_manual(stat.test, label = "p.scient") +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.15)))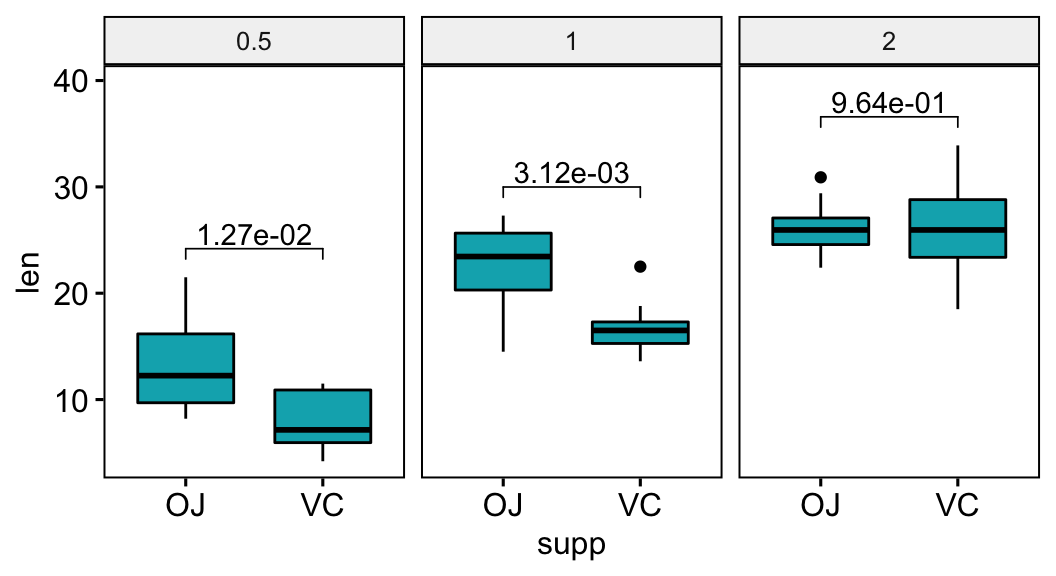
Comparer des échantillons appariés
# Test statistique
stat.test <- df %>%
t_test(len ~ supp, paired = TRUE) %>%
add_significance()
stat.test## # A tibble: 1 x 9
## .y. group1 group2 n1 n2 statistic df p p.signif
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <chr>
## 1 len OJ VC 30 30 3.30 29 0.00255 **# Box plot avec p-values
bxp <- ggpaired(df, x = "supp", y = "len", fill = "#E7B800",
line.color = "gray", line.size = 0.4)
stat.test <- stat.test %>% add_xy_position(x = "supp")
bxp + stat_pvalue_manual(stat.test, label = "p.signif")
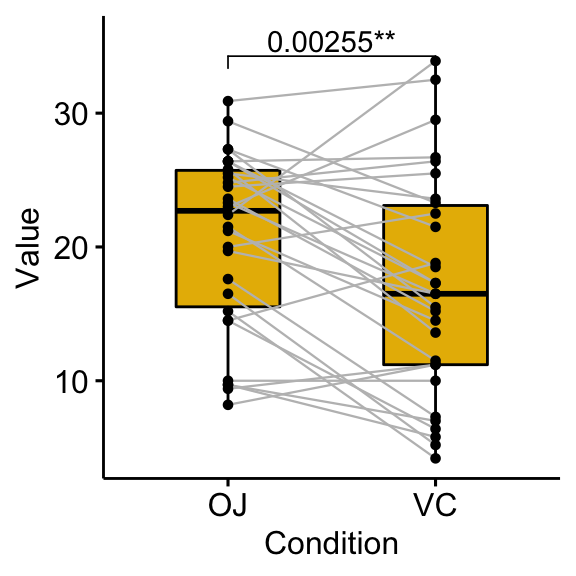
# Indiquer la p-value combinée avec le niveau de significativité
bxp +
stat_pvalue_manual(stat.test, label = "{p}{p.signif}") +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.10)))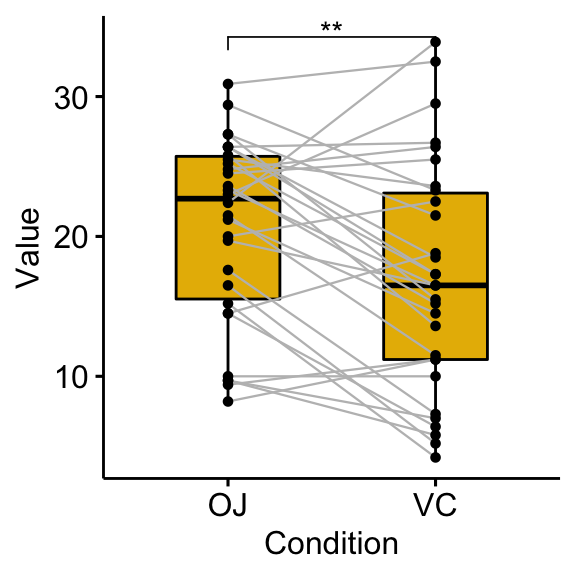
Comparaisons par paires
Créer des graphiques simples
# Box plots
bxp <- ggboxplot(df, x = "dose", y = "len", fill = "dose",
palette = c("#00AFBB", "#E7B800", "#FC4E07"))
bxp
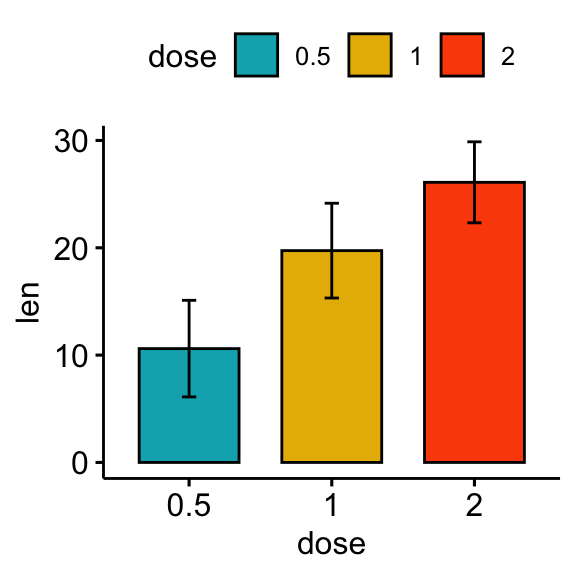
# Bar plots représentant la moyenne +/- l'écart-type
bp <- ggbarplot(df, x = "dose", y = "len", add = "mean_sd", fill = "dose",
palette = c("#00AFBB", "#E7B800", "#FC4E07"))
bp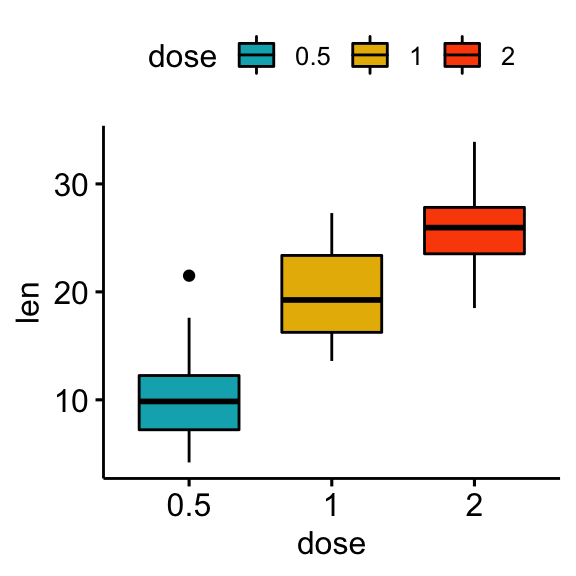
Test statistique
Dans l’exemple suivant, nous allons effectuer un test-T en utilisant la fonction t_test() [package rstatix]. Il est également possible d’utiliser la fonction wilcox_test().
stat.test <- df %>% t_test(len ~ dose)
stat.test## # A tibble: 3 x 10
## .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 len 0.5 1 20 20 -6.48 38.0 1.27e- 7 2.54e- 7 ****
## 2 len 0.5 2 20 20 -11.8 36.9 4.40e-14 1.32e-13 ****
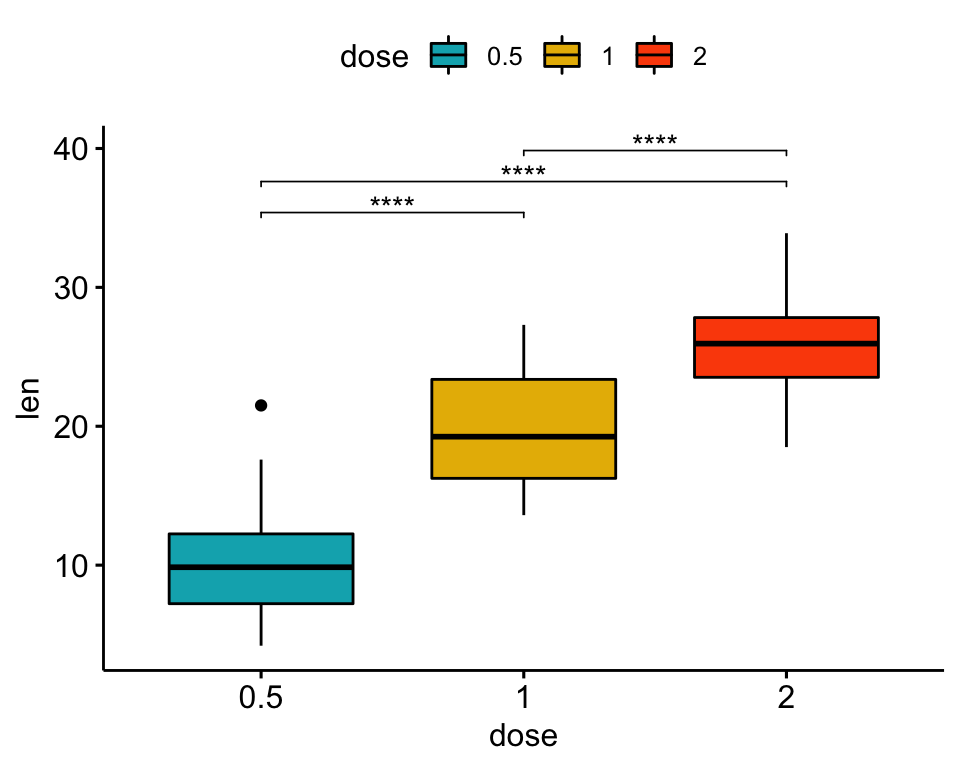
## 3 len 1 2 20 20 -4.90 37.1 1.91e- 5 1.91e- 5 ****Créer des graphiques avec les niveaux de significativité
# Box plot
stat.test <- stat.test %>% add_xy_position(x = "dose")
bxp + stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01)
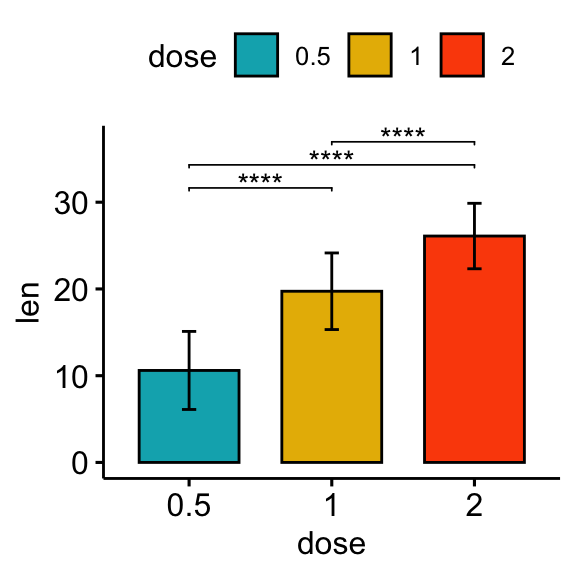
# Bar plot
stat.test <- stat.test %>% add_xy_position(fun = "mean_sd", x = "dose")
bp + stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01)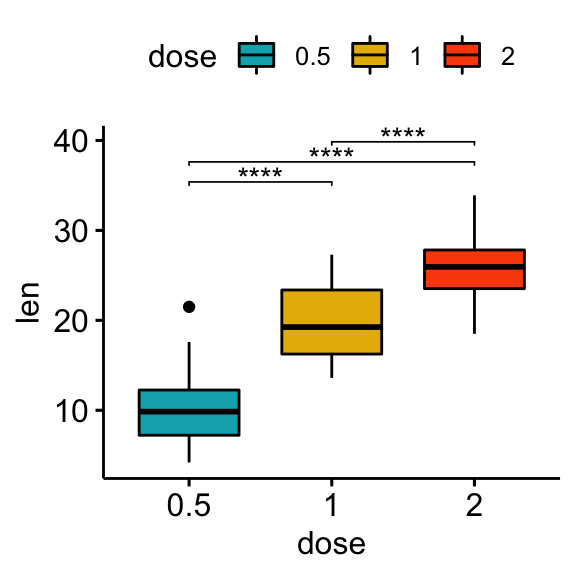
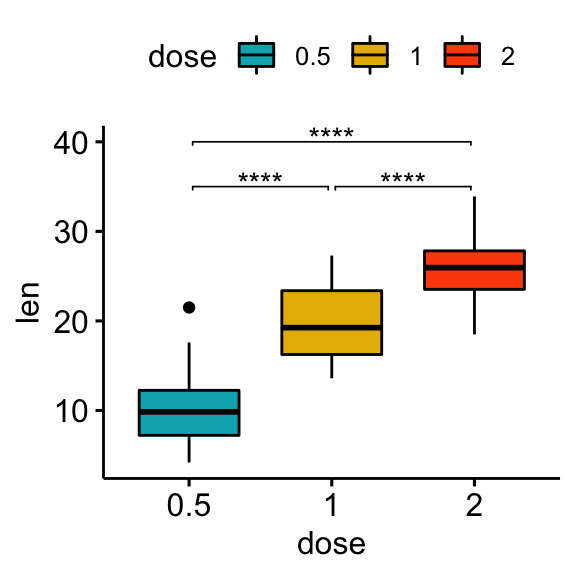
Spécifier manuellement la position y des étiquettes de p-values et raccourcir la largeur des crochets:
bxp +
stat_pvalue_manual(
stat.test, label = "p.adj.signif", tip.length = 0.01,
y.position = c(35, 40, 35), bracket.shorten = 0.05
)
Comparaisons par rapport à un groupe de référence
# Tests statistiques
stat.test <- df %>% t_test(len ~ dose, ref.group = "0.5")
stat.test## # A tibble: 2 x 10
## .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 len 0.5 1 20 20 -6.48 38.0 1.27e- 7 1.27e- 7 ****
## 2 len 0.5 2 20 20 -11.8 36.9 4.40e-14 8.80e-14 ****# Box plot
stat.test <- stat.test %>% add_xy_position(x = "dose")
bxp + stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01)
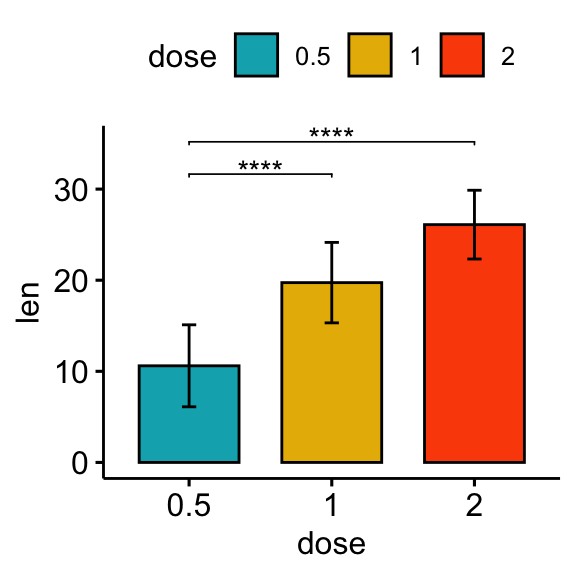
# Bar plot
stat.test <- stat.test %>% add_xy_position(fun = "mean_sd", x = "dose")
bp + stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01)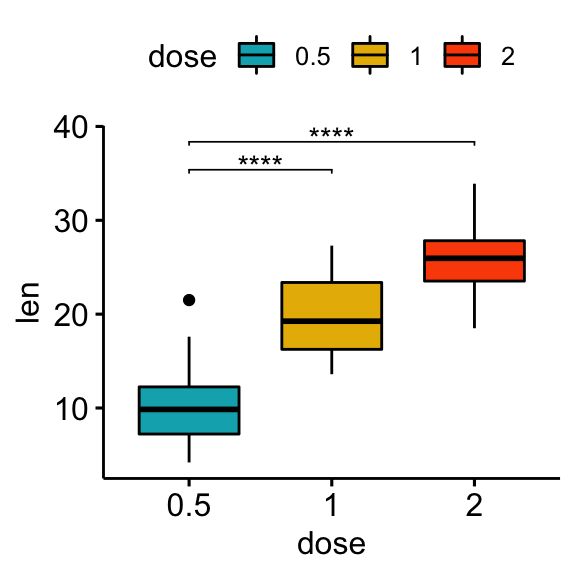
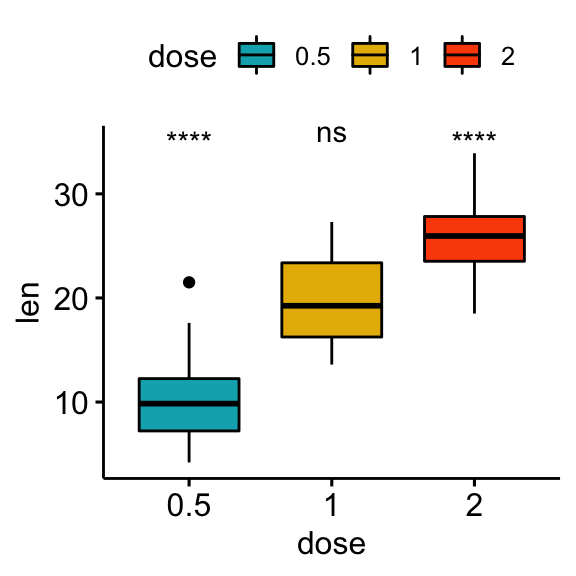
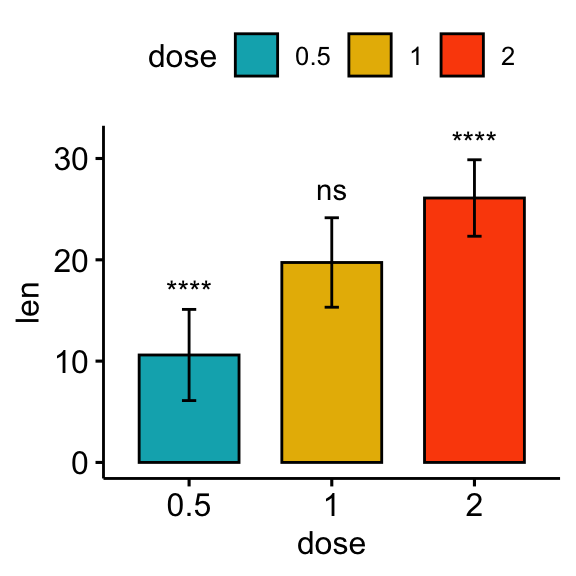
Comparaisons par rapport à l’ensemble (basemean)
Chaque groupe est comparé à tous les groupes combinés.
# Tests statistiques
stat.test <- df %>% t_test(len ~ dose, ref.group = "all")
stat.test## # A tibble: 3 x 10
## .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 len all 0.5 60 20 5.82 56.4 0.000000290 0.00000087 ****
## 2 len all 1 60 20 -0.660 57.5 0.512 0.512 ns
## 3 len all 2 60 20 -5.61 66.5 0.000000425 0.00000087 ****# Box plot
stat.test <- stat.test %>% add_xy_position(x = "dose")
bxp + stat_pvalue_manual(stat.test, label = "p.adj.signif")
# Préciser manuellement la position y
bxp + stat_pvalue_manual(stat.test, label = "p.adj.signif", y.position = 35)
# Bar plot
stat.test <- stat.test %>% add_xy_position(fun = "mean_sd", x = "dose")
bp + stat_pvalue_manual(stat.test, label = "p.adj.signif")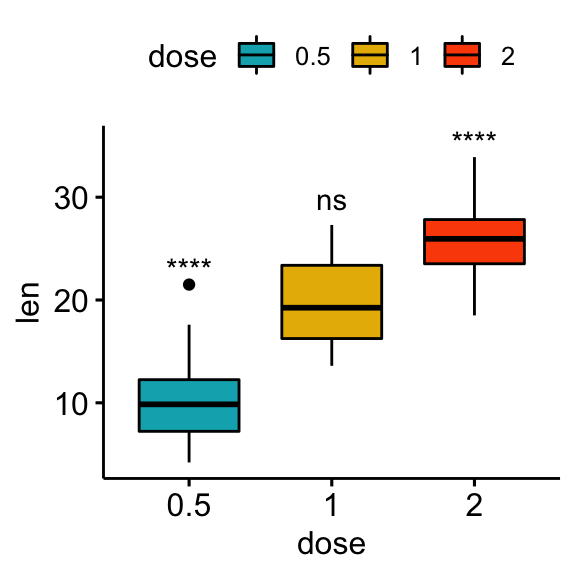
Comparaisons par rapport à une valeur théorique: test à un échantillon
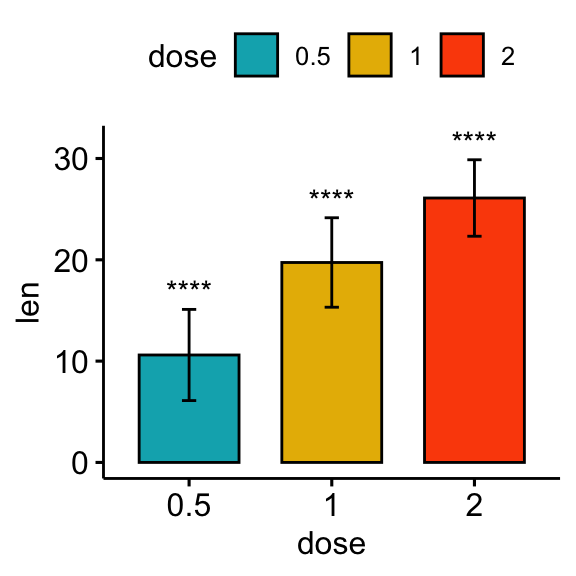
Le test à un échantillon est utilisé pour comparer la moyenne d’un échantillon à une moyenne standard (ou théorique / hypothétique) connue (mu). La valeur par défaut de `mu est zéro.
# Tests statistiques
stat.test <- df %>%
group_by(dose) %>%
t_test(len ~ 1) %>%
adjust_pvalue() %>%
add_significance("p.adj")
stat.test## # A tibble: 3 x 10
## dose .y. group1 group2 n statistic df p p.adj p.adj.signif
## * <fct> <chr> <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 0.5 len 1 null model 20 10.5 19 2.24e- 9 2.24e- 9 ****
## 2 1 len 1 null model 20 20.0 19 3.22e-14 6.44e-14 ****
## 3 2 len 1 null model 20 30.9 19 1.03e-17 3.09e-17 ****# Box plot
stat.test <- stat.test %>% add_xy_position(x = "dose")
bxp + stat_pvalue_manual(stat.test, x = "dose", label = "p.adj.signif")
# bar plot
stat.test <- stat.test %>% add_xy_position(fun = "mean_sd", x = "dose")
bp + stat_pvalue_manual(stat.test, x = "dose", label = "p.adj.signif")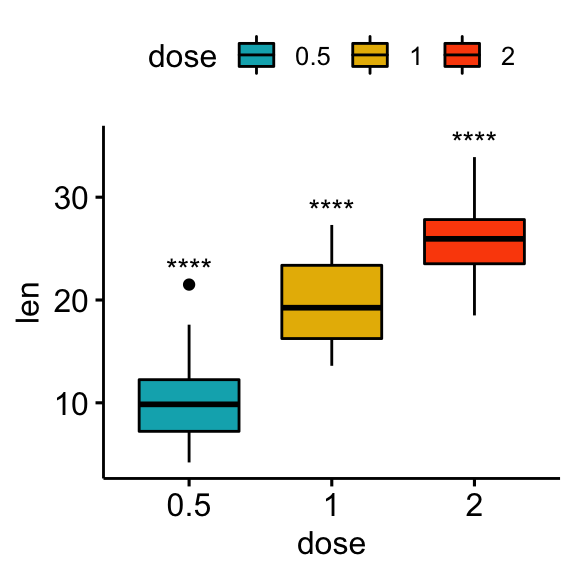
Conclusion
Cet article explique comment calculer et ajouter facilement des p-values sur des graphiques, tels que les box plots et les bar plots. Voir les autres questions fréquemment posées : ggpubr FAQ.
Version:
 English
English






No Comments