Cet article décrit comment calculer et ajouter automatiquement des p-values sur des ggplots groupés en utilisant les packages R ggpubr et rstatix.
Vous apprendrez à:
- Ajouter des p-values sur des graphiques (box plot, bar plot et line plot) groupés. Des exemples, contenant deux et trois groupes par position x, sont présentés.
- Indiquer les p-values combinées avec les niveaux de significativité sur les graphiques groupés
Nous suivrons les étapes suivantes pour ajouter des niveaux de significativité sur un ggplot:
- Calculer facilement des tests statistiques (
t_test()ouwilcox_test()) en utilisant le packagerstatix - Auto-calculez les positions des étiquettes des p-values en utilisant la fonction
add_xy_position()[dans le package rstatix]. - Ajoutez les p-values au graphique en utilisant la fonction
stat_pvalue_manual()[dans le package ggpubr]. Les options clés suivantes sont illustrées dans certains des exemples:- L’option
bracket.nudge.yest utilisée pour monter ou descendre les crochets. - L’option
step.increaseest utilisée pour ajouter de l’espace entre les crochets. - L’option
vjustest utilisée pour ajuster verticalement la position des étiquettes des p-values
- L’option
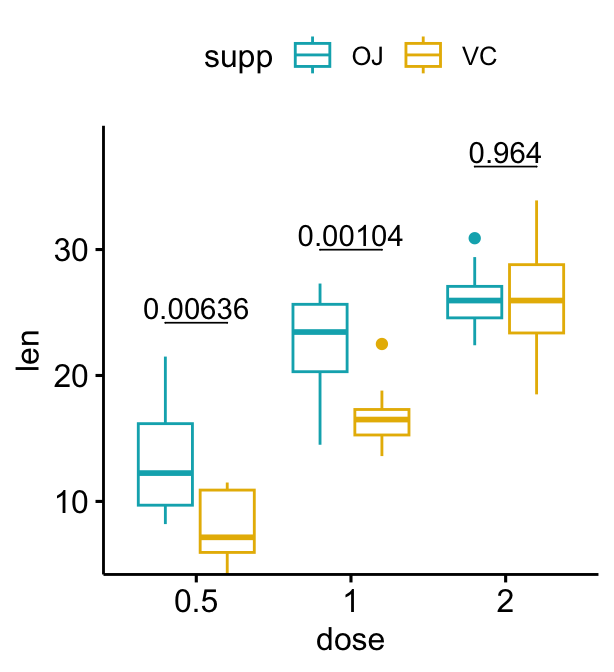
Notez que, dans certains cas, les étiquettes de p-values sont partiellement cachées par la bordure supérieure du graphique. Dans ce cas, la fonction ggplot2 scale_y_continuous(expand = expansion(mult = c(0, 0.1))) peut être utilisée pour ajouter des espaces entre les étiquettes et la bordure supérieure du graphique. L’option mult = c(0, 0.1) indique que des espaces de 0% et 10% sont respectivement ajoutés en bas et en haut du graphique.
Sommaire:
Prérequis
Assurez-vous d’avoir installé les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixcontient des fonctions R facilitant les analyses statistiques.
Commencez par charger les packages requis suivants:
library(ggpubr)
library(rstatix)Fonctions R clés
Fonctions de tests statistiques pour les comparaisons par paires : t_test() et wilcox_test() [package rstatix]
Une interface pipe-compatible pour comparer la moyenne de deux groupes. Si la variable de regroupement contient plus de deux niveaux, alors une comparaison par paire entre les niveaux est automatiquement calculée et la p-value est ajustée en utilisant les méthodes Holm d’ajustement de la p-value (par défaut). Vous pouvez changer cela en utilisant par exemple la méthode p.adjust.method = "bonferroni", qui est plus stringent mais fréquemment utilisée.
Calculez automatiquement les positions x et y de la p-value pour représenter graphiquement les p-values et les niveaux de significativité : add_xy_position() [rstatix package]
l’un des arguments clés est fun, qui indique les fonctions de statistiques descriptives utilisées pour calculer automatiquement les positions y appropriées des étiquettes et des crochets de p-value. La valeur par défaut est fun = "max", ce qui permet de calculer les positions des p-values pour des box plots.
Si vous créez un graphique à barres ou un graphique linéaire avec des barres d’erreur (moyenne +/- SD ou moyenne +/_ se), alors vous devez spécifier les fonctions de statistiques descriptives correspondantes lors du calcul des positions des étiquettes de p-valueq. Par exemple, fun = "mean_sd". Les valeurs possibles sont les suivantes: "max", "mean", "mean_sd", "mean_se", "mean_ci", "median", "median_iqr", "median_mad".
Ajouter manuellement des p-values à un ggplot : stat_pvalue_manual() [dans le package ggpubr]
Cette fonction peut être utilisée pour ajouter manuellement des p-values à un ggplot, comme les box blots, les points, les stripcharts, les lignes et les barres. Les questions fréquemment posées sont disponibles sur la page [Datanovia ggpubr FAQ] (https://www.datanovia.com/en/fr/blog/tag/ggpubr-fr/).
Les principaux arguments sont les suivants:
data: un tableau de données contenant des résultats de tests statistiques. Le format par défaut attendu doit contenir les colonnes suivantes:group1 | group2 | p | y.position | etc.group1etgroup2sont les groupes qui ont été comparés.pest la p-value résultante.y.positionest la coordonnée y des p-values dans le graphique.label: la colonne contenant l’étiquette (par exemple :label = "p"oulabel = "p.adj"), où p est la p-value. Peut également être une expression qui peut être formatée par le packageglue. Par exemple, lorsque l’on spécifielabel = "t-test, p = {p}", l’expression{p}sera remplacée par sa valeur.bracket.nudge.y: Ajustement vertical des crochets par. Utile pour monter ou baisser le crochet. Si la valeur est positive, les crochets seront déplacés vers le haut ; en cas de valeur négative, les crochets seront déplacés vers le bas.remove.bracket: valeur logique, siTRUE, les crochets sont supprimés du graphique.step.increase: fraction d’espace supplémentaire à ajouter entre les crochets pour minimiser le chevauchement.hide.ns: valeur logique. SiTRUE, cache le symbole ns lors de l’affichage des niveaux de significativité.
Préparation des données
# Transformer `dose` en variable factorielle
df <- ToothGrowth
df$dose <- as.factor(df$dose)
head(df, 3)## len supp dose
## 1 4.2 VC 0.5
## 2 11.5 VC 0.5
## 3 7.3 VC 0.5Deux groupes par position x
Tests statistiques
Regroupez les données par la variable dose et comparez ensuite les niveaux de la variable suppl (OJ contre CV):
stat.test <- df %>%
group_by(dose) %>%
t_test(len ~ supp) %>%
adjust_pvalue(method = "bonferroni") %>%
add_significance("p.adj")
stat.test## # A tibble: 3 x 11
## dose .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 0.5 len OJ VC 10 10 3.17 15.0 0.00636 0.0191 *
## 2 1 len OJ VC 10 10 4.03 15.4 0.00104 0.00312 **
## 3 2 len OJ VC 10 10 -0.0461 14.0 0.964 1 nsBox plots groupés
# Créer un box plot
bxp <- ggboxplot(
df, x = "dose", y = "len",
color = "supp", palette = c("#00AFBB", "#E7B800")
)
# Ajoutez des p-values sur les graphiques en box plot
stat.test <- stat.test %>%
add_xy_position(x = "dose", dodge = 0.8)
bxp + stat_pvalue_manual(
stat.test, label = "p", tip.length = 0
)
# Ajoutez 10 % d'espaces entre les étiquettes des p-values et la bordure du graphique
bxp + stat_pvalue_manual(
stat.test, label = "p", tip.length = 0
) +
scale_y_continuous(expand = expansion(mult = c(0, 0.1)))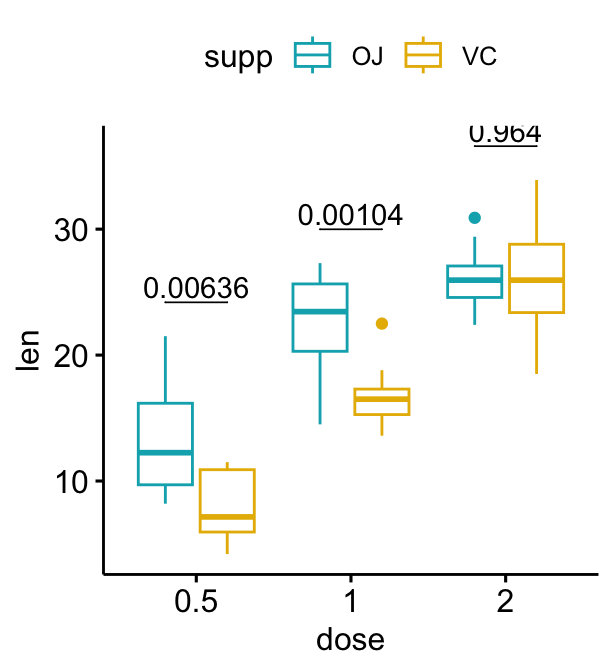
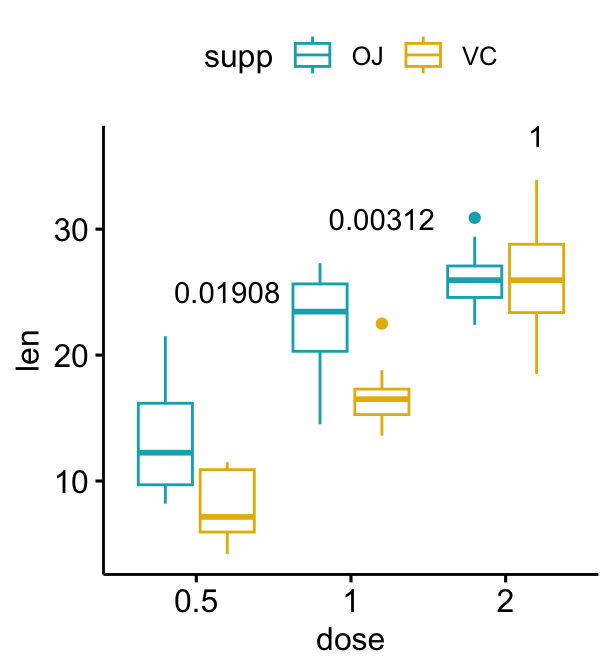
# Utiliser des p-values ajustées comme étiquettes
# Supprimer les crochets
bxp + stat_pvalue_manual(
stat.test, label = "p.adj", tip.length = 0,
remove.bracket = TRUE
)
# Afficher les p-values ajustées et les niveaux de significativité
# Cacher les ns (non significatif)
bxp + stat_pvalue_manual(
stat.test, label = "{p.adj}{p.adj.signif}",
tip.length = 0, hide.ns = TRUE
)
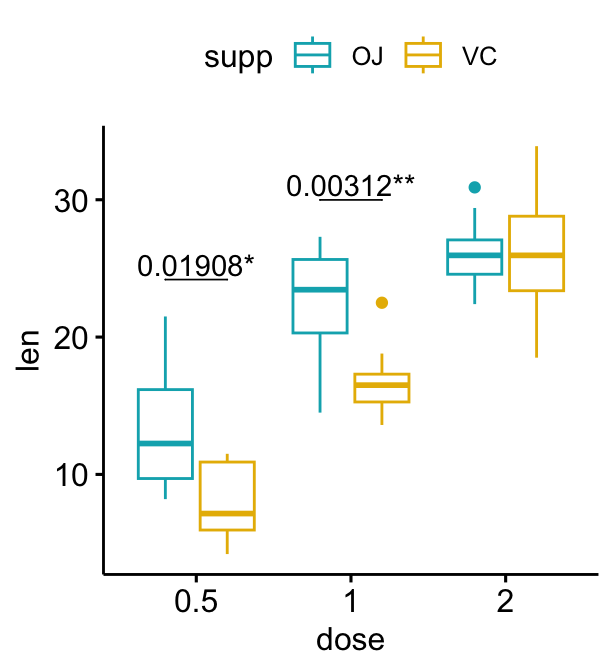
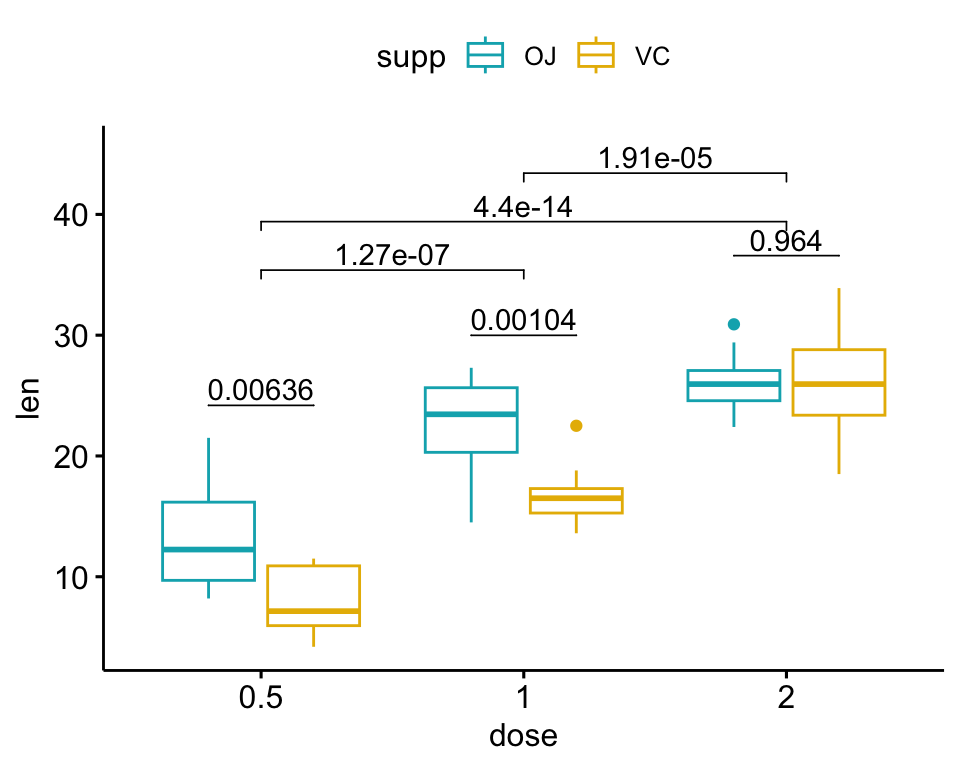
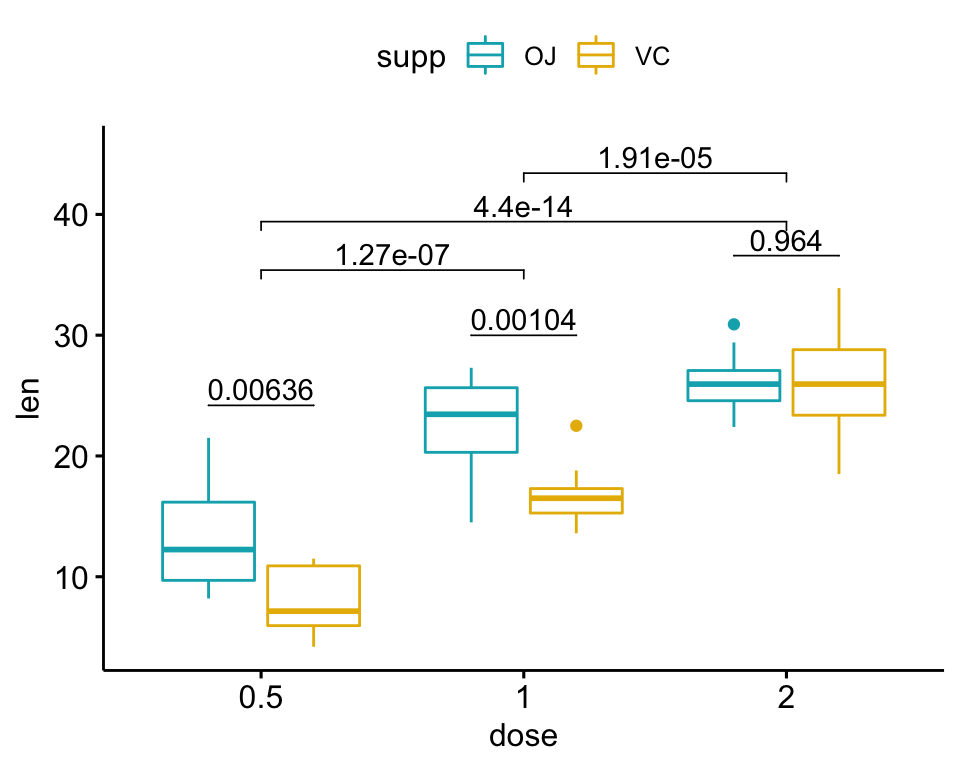
Le script R suivant crée un graphique en box plot représentant deux résultats de tests statistiques:
- le
stat.testci-dessus comparant les niveaux de la variablesupp(OJvsVC) - un test statistique supplémentaire (
stat.test2) permettant de comparer par paires les niveaux dedose. Les p-values sont automatiquement ajustés car la variable “dose” contient 3 niveaux.
# Test statistique supplémentaire
stat.test2 <- df %>%
t_test(len ~ dose, p.adjust.method = "bonferroni")
stat.test2## # A tibble: 3 x 10
## .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 len 0.5 1 20 20 -6.48 38.0 1.27e- 7 3.81e- 7 ****
## 2 len 0.5 2 20 20 -11.8 36.9 4.40e-14 1.32e-13 ****
## 3 len 1 2 20 20 -4.90 37.1 1.91e- 5 5.73e- 5 ****# Ajouter les p-values de `stat.test` and `stat.test2`
# 1. Ajouter stat.test
stat.test <- stat.test %>%
add_xy_position(x = "dose", dodge = 0.8)
bxp.complex <- bxp + stat_pvalue_manual(
stat.test, label = "p", tip.length = 0
)
# 2. Ajouter stat.test2
# Ajoutez de l'espace entre crochets en utilisant `step.increase`
stat.test2 <- stat.test2 %>% add_xy_position(x = "dose")
bxp.complex <- bxp.complex +
stat_pvalue_manual(
stat.test2, label = "p", tip.length = 0.02,
step.increase = 0.05
) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.1)))
# 3. Afficher le graphique
bxp.complex 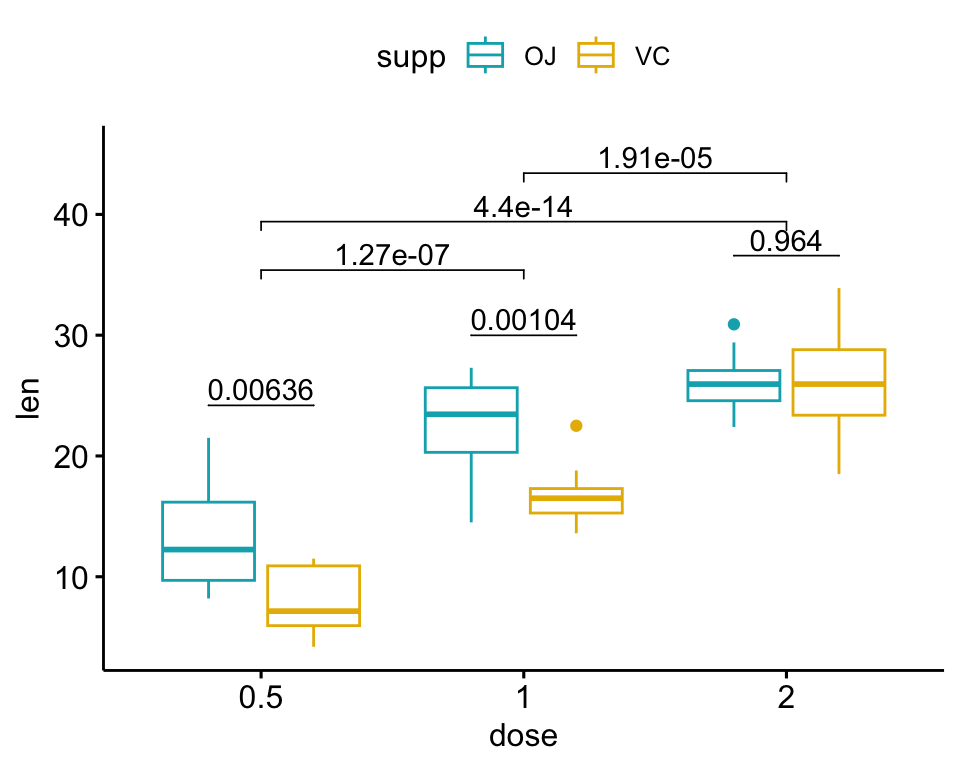
Graphiques groupés
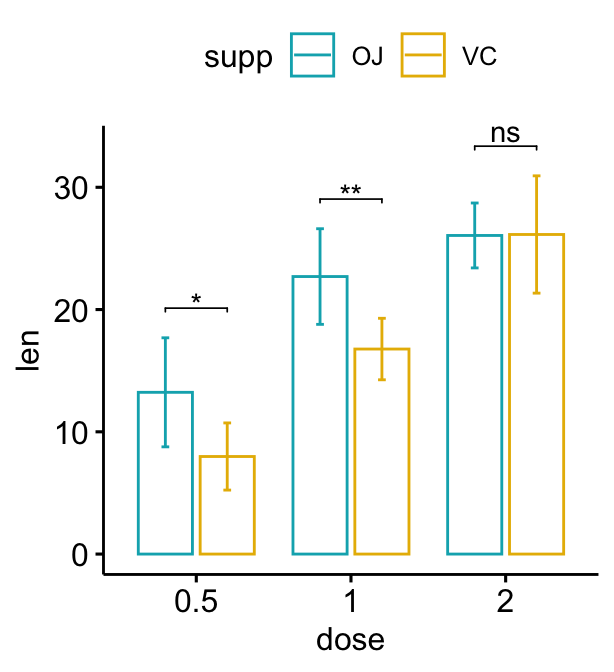
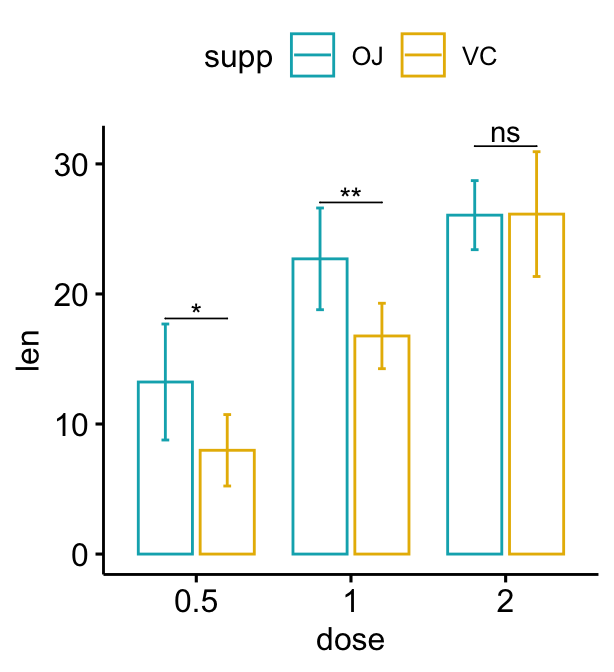
L’option add = "mean_sd" est spécifiée dans la fonction de graphique à barres pour créer un graphique à barres avec des barres d’erreur (mean +/- SD). Vous devez spécifier la même fonction de statistiques sommaires pour calculer automatiquement les positions des étiquettes de p-values dans add_xy_position() en utilisant l’option fun.
Bar plot dodged
# Créer un graphique avec des barres d'erreur (moyenne +/- sd)
bp <- ggbarplot(
df, x = "dose", y = "len", add = "mean_sd",
color= "supp", palette = c("#00AFBB", "#E7B800"),
position = position_dodge(0.8)
)
# Ajoutez des p-values sur les graphiques en barres
stat.test <- stat.test %>%
add_xy_position(fun = "mean_sd", x = "dose", dodge = 0.8)
bp + stat_pvalue_manual(
stat.test, label = "p.adj.signif", tip.length = 0.01
)
# Faites baisser les crochets en utilisant `bracket.nudge.y`
bp + stat_pvalue_manual(
stat.test, label = "p.adj.signif", tip.length = 0,
bracket.nudge.y = -2
)
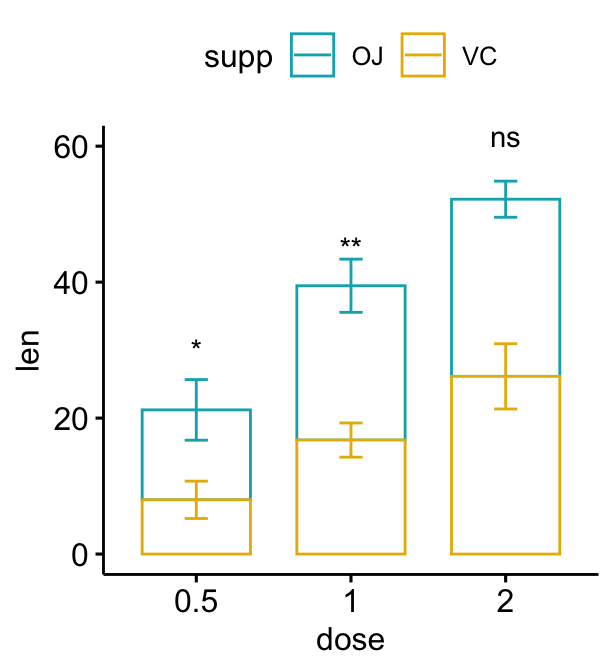
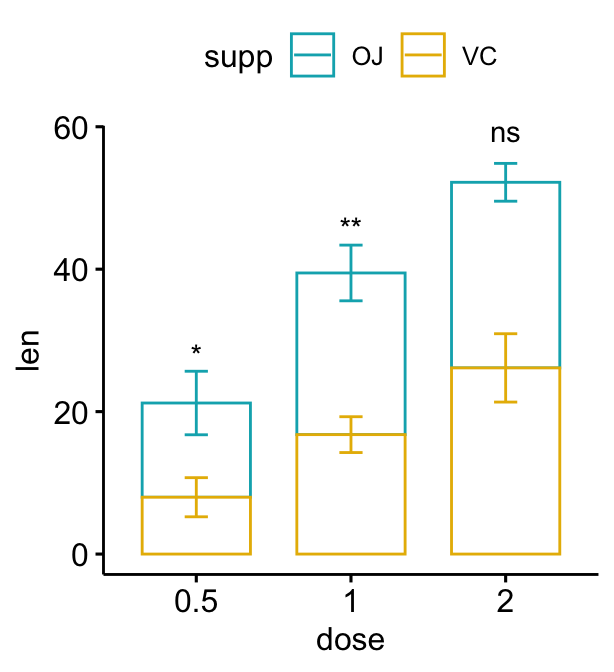
Bar plot empilés
# Créer un graphique avec des barres d'erreur (moyenne +/- sd)
bp2 <- ggbarplot(
df, x = "dose", y = "len", add = "mean_sd",
color = "supp", palette = c("#00AFBB", "#E7B800"),
position = position_stack()
)
# Ajoutez des p-values sur les graphiques en barres
# Préciser manuellement la position y des p-values
bp2 + stat_pvalue_manual(
stat.test, label = "p.adj.signif", tip.length = 0.01,
x = "dose", y.position = c(30, 45, 60)
)
# Calcul automatique de la position y des p-values
# Ajustez les positions verticales des étiquettes en utilisant vjust
stat.test <- stat.test %>%
add_xy_position(fun = "mean_sd", x = "dose", stack = TRUE)
bp2 + stat_pvalue_manual(
stat.test, label = "p.adj.signif",
remove.bracket = TRUE, vjust = -0.2
)
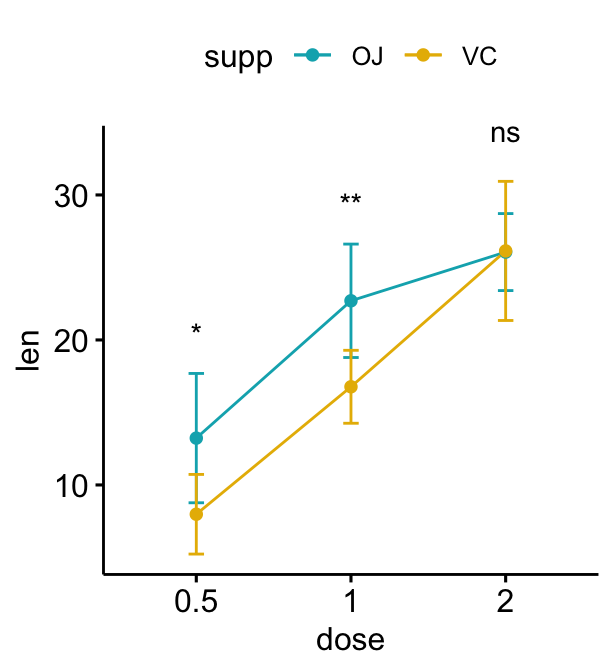
Line plots groupés
# Créer un graphique linéaire avec des barres d'erreur (moyenne +/- sd)
lp <- ggline(
df, x = "dose", y = "len", add = "mean_sd",
color = "supp", palette = c("#00AFBB", "#E7B800")
)
# Ajoutez des p-values sur les graphiques linéaires
# Supprimer les crochets en utilisant linetype = "blanc"
stat.test <- stat.test %>%
add_xy_position(fun = "mean_sd", x = "dose")
lp + stat_pvalue_manual(
stat.test, label = "p.adj.signif",
tip.length = 0, linetype = "blank"
)
# Baissez les niveaux de significativité en utilisant vjust
lp + stat_pvalue_manual(
stat.test, label = "p.adj.signif",
linetype = "blank", vjust = 2
)
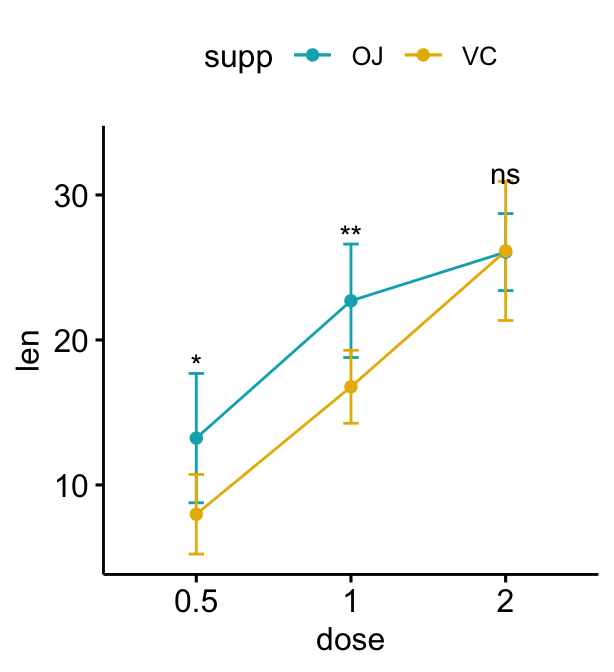
Comparaisons par paires
Tests statistiques
Regroupez les données par la variable supp et effectuez ensuite de multiples comparaisons par paires entre les niveaux de la variable dose (0,5, 1 et 2). Les p-values sont ajustées indépendamment pour chaque niveau de groupe.
pwc <- df %>%
group_by(supp) %>%
t_test(len ~ dose, p.adjust.method = "bonferroni")
pwc## # A tibble: 6 x 11
## supp .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 OJ len 0.5 1 10 10 -5.05 17.7 0.0000878 0.000263 ***
## 2 OJ len 0.5 2 10 10 -7.82 14.7 0.00000132 0.00000396 ****
## 3 OJ len 1 2 10 10 -2.25 15.8 0.039 0.118 ns
## 4 VC len 0.5 1 10 10 -7.46 17.9 0.000000681 0.00000204 ****
## 5 VC len 0.5 2 10 10 -10.4 14.3 0.0000000468 0.00000014 ****
## 6 VC len 1 2 10 10 -5.47 13.6 0.0000916 0.000275 ***Créer des graphiques représentant les p-values de la comparaison par paires
L’argument step.group.by est utilisé pour regrouper les crochets par une variable.
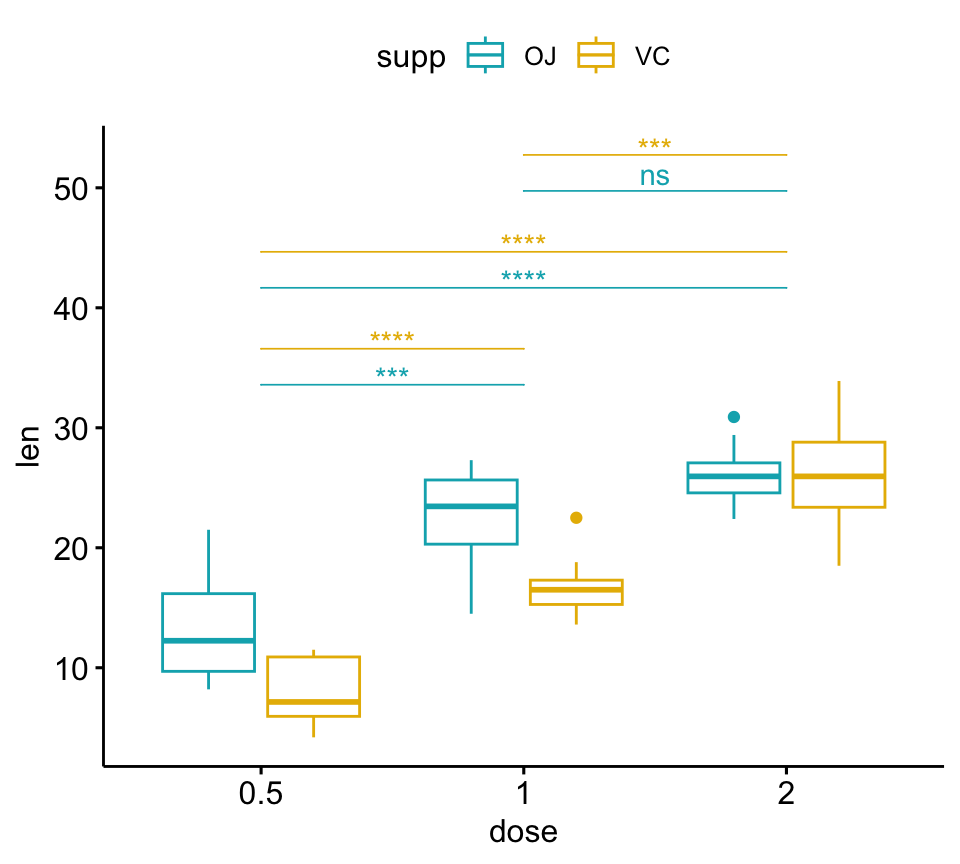
# Box plot
pwc <- pwc %>% add_xy_position(x = "dose")
bxp +
stat_pvalue_manual(
pwc, color = "supp", step.group.by = "supp",
tip.length = 0, step.increase = 0.1
)
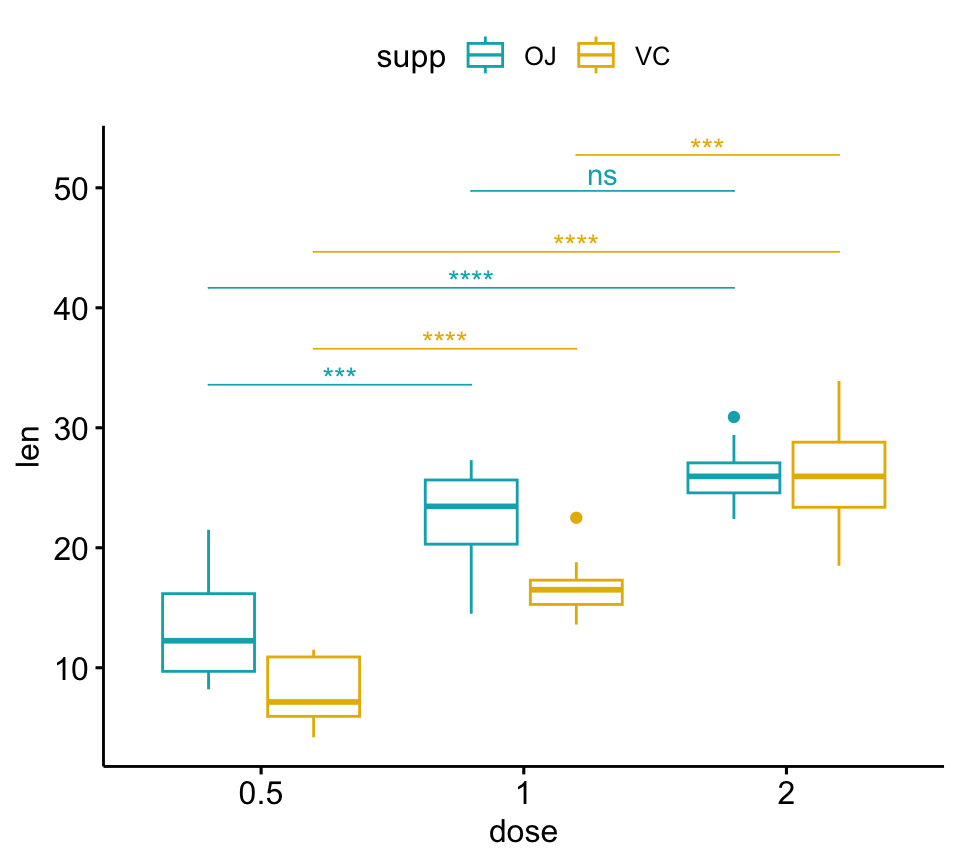
# Bar plots
pwc <- pwc %>% add_xy_position(x = "dose", fun = "mean_sd", dodge = 0.8)
bp + stat_pvalue_manual(
pwc, color = "supp", step.group.by = "supp",
tip.length = 0, step.increase = 0.1
)
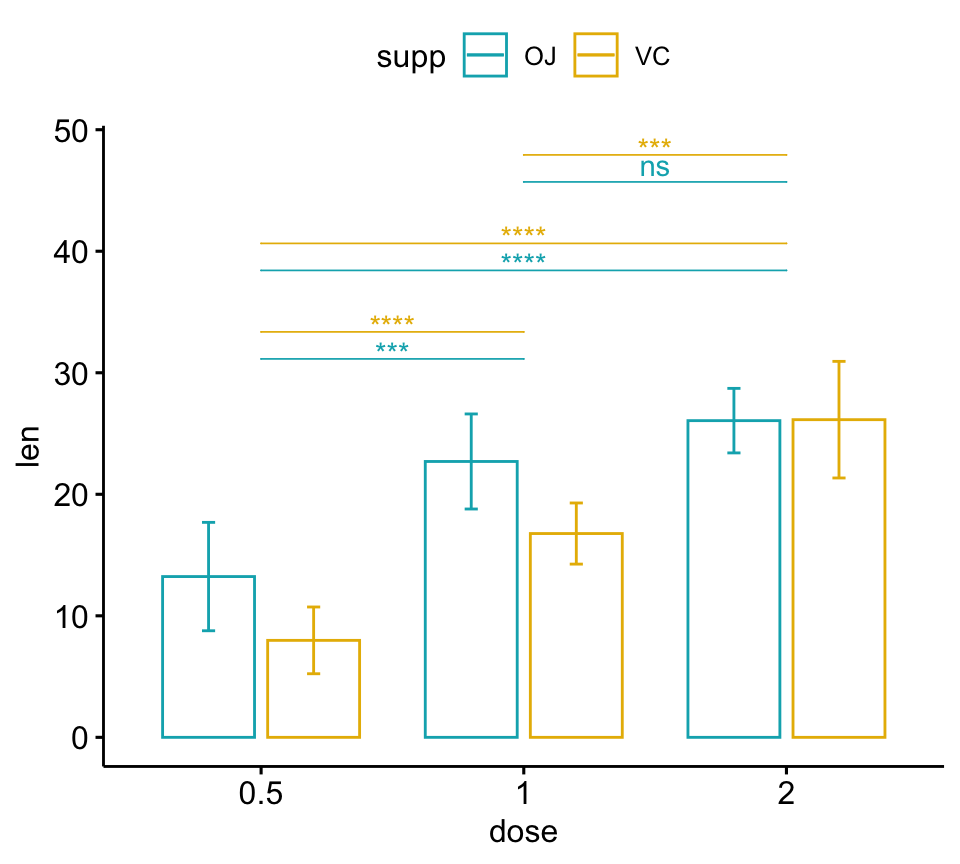
# Line plots
pwc <- pwc %>% add_xy_position(x = "dose", fun = "mean_sd")
lp + stat_pvalue_manual(
pwc, color = "supp", step.group.by = "supp",
tip.length = 0, step.increase = 0.1
)
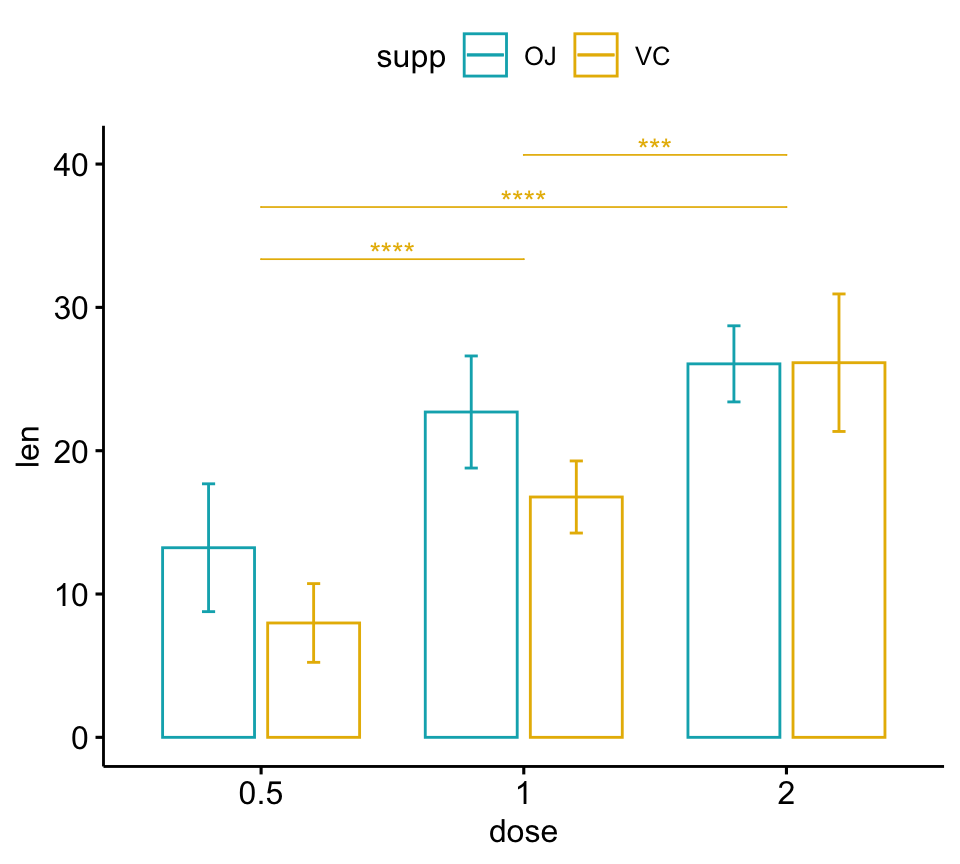
Afficher les p-values pour un sous-ensemble de comparaisons
Dans l’exemple ci-dessous, les p-values sont indiquées pour les comparaisons par paires dans le groupe VC:
# Bar plot (dodge)
# Prenez un sous-ensemble des comparaisons par paires
pwc.filtered <- pwc %>%
add_xy_position(x = "dose", fun = "mean_sd", dodge = 0.8) %>%
filter(supp == "VC")
bp +
stat_pvalue_manual(
pwc.filtered, color = "supp", step.group.by = "supp",
tip.length = 0, step.increase = 0
)
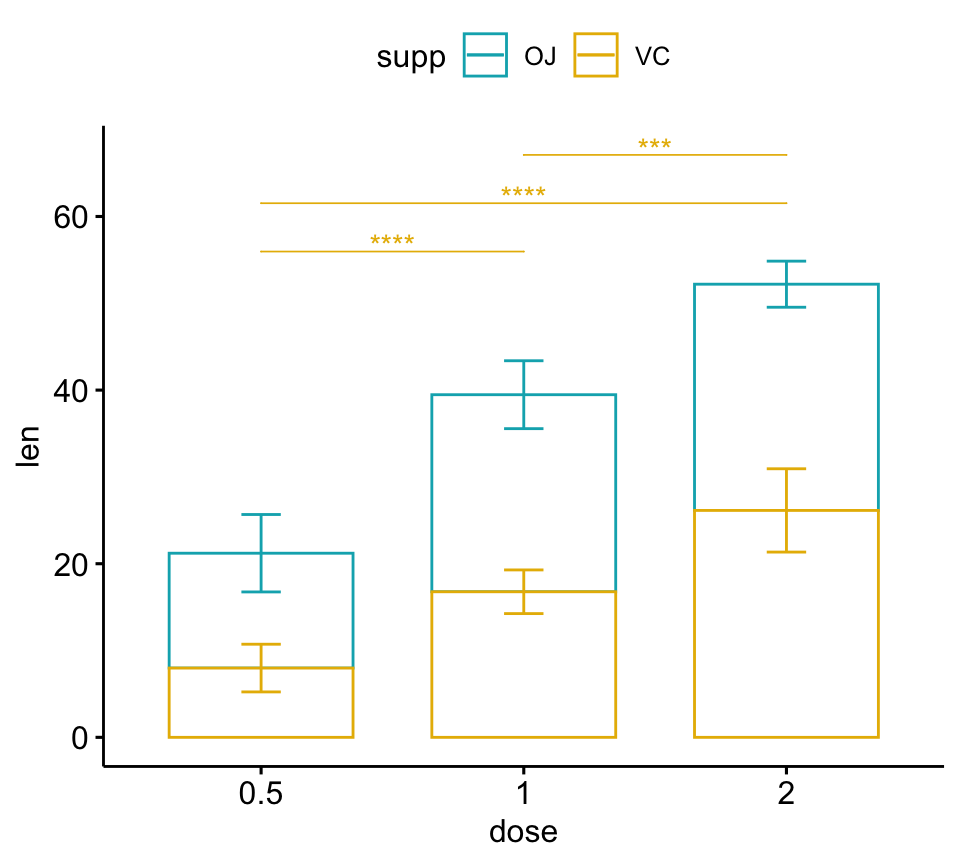
# Bar plot (empilés)
pwc.filtered <- pwc %>%
add_xy_position(x = "dose", fun = "mean_sd", stack = TRUE) %>%
filter(supp == "VC")
bp2 +
stat_pvalue_manual(
pwc.filtered, color = "supp", step.group.by = "supp",
tip.length = 0, step.increase = 0.1
)
Trois groupes par position x
Graphiques simples
# Box plots
bxp <- ggboxplot(
df, x = "supp", y = "len", fill = "dose",
palette = "npg"
)
bxp
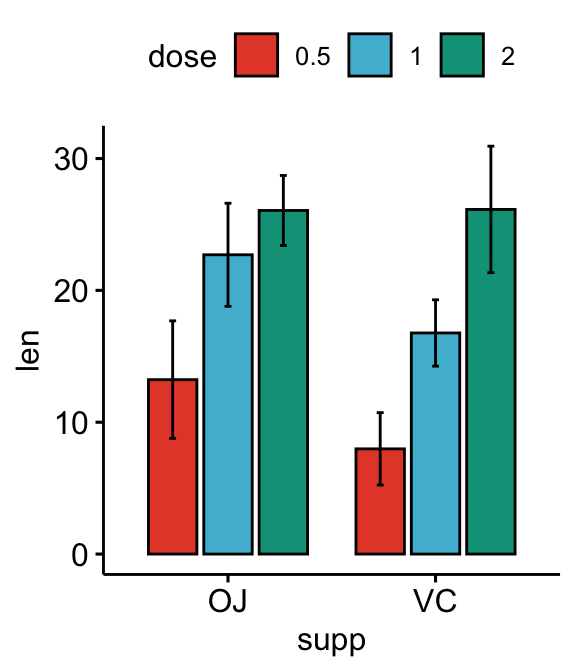
# Bar plots
bp <- ggbarplot(
df, x = "supp", y = "len", fill = "dose",
palette = "npg", add = "mean_sd",
position = position_dodge(0.8)
)
bp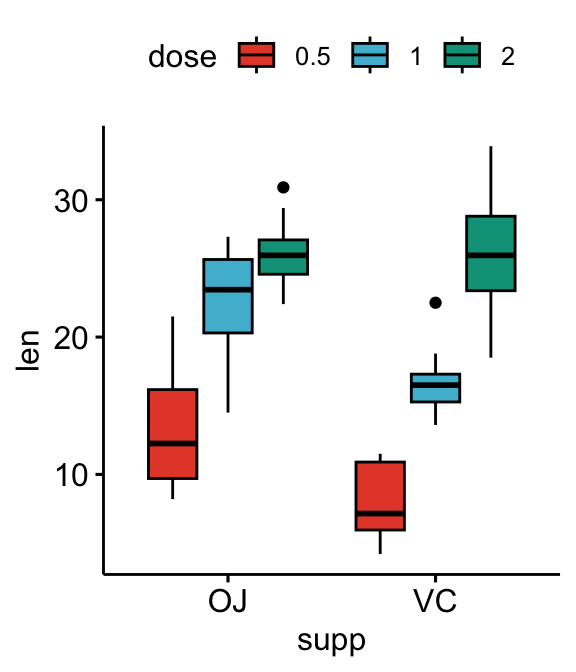
Effectuer toutes les comparaisons par paires
Regrouper par la variable supp et ensuite effectuer des comparaisons par paires entre les niveaux de la variable dose.
Test statistique:
stat.test <- df %>%
group_by(supp) %>%
t_test(len ~ dose)
stat.test ## # A tibble: 6 x 11
## supp .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 OJ len 0.5 1 10 10 -5.05 17.7 0.0000878 0.000176 ***
## 2 OJ len 0.5 2 10 10 -7.82 14.7 0.00000132 0.00000396 ****
## 3 OJ len 1 2 10 10 -2.25 15.8 0.039 0.039 *
## 4 VC len 0.5 1 10 10 -7.46 17.9 0.000000681 0.00000136 ****
## 5 VC len 0.5 2 10 10 -10.4 14.3 0.0000000468 0.00000014 ****
## 6 VC len 1 2 10 10 -5.47 13.6 0.0000916 0.0000916 ****Ajouter les p-values sur les graphiques:
- L’argument
bracket.nudge.yest utilisé pour baisser les crochets. - La fonction ggplot2
scale_y_continuous(expand = expansion(mult = c(0, 0.1)))est utilisée pour ajouter des espaces supplémentaires entre les étiquettes et la bordure supérieure du graphique
# Box plot avec p-values
stat.test <- stat.test %>%
add_xy_position(x = "supp", dodge = 0.8)
bxp +
stat_pvalue_manual(
stat.test, label = "p.adj", tip.length = 0.01,
bracket.nudge.y = -2
) +
scale_y_continuous(expand = expansion(mult = c(0, 0.1)))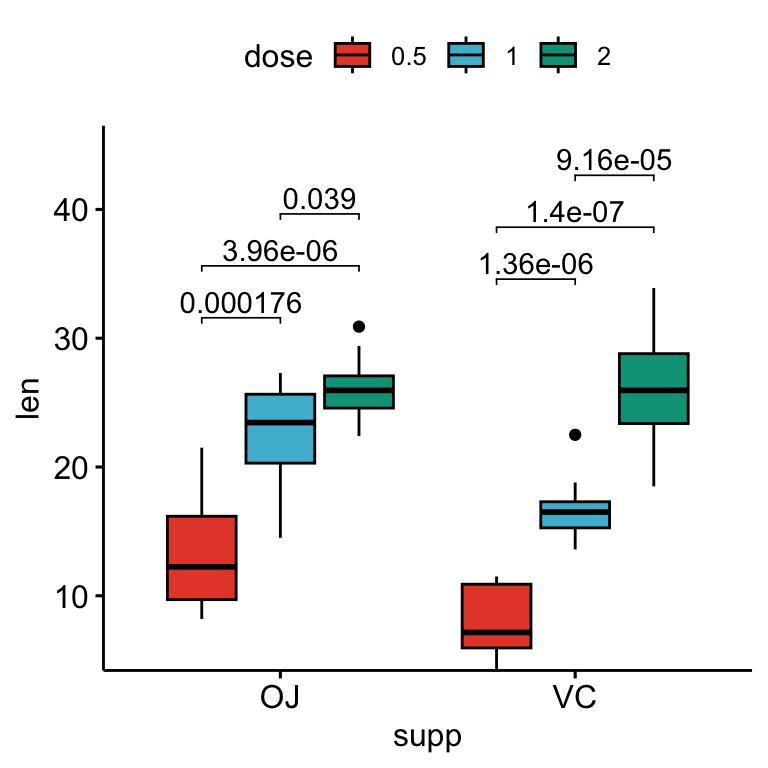
# Bar plot avec p-values
stat.test <- stat.test %>%
add_xy_position(x = "supp", fun = "mean_sd", dodge = 0.8)
bp +
stat_pvalue_manual(
stat.test, label = "p.adj", tip.length = 0.01,
bracket.nudge.y = -2
) +
scale_y_continuous(expand = expansion(mult = c(0, 0.1)))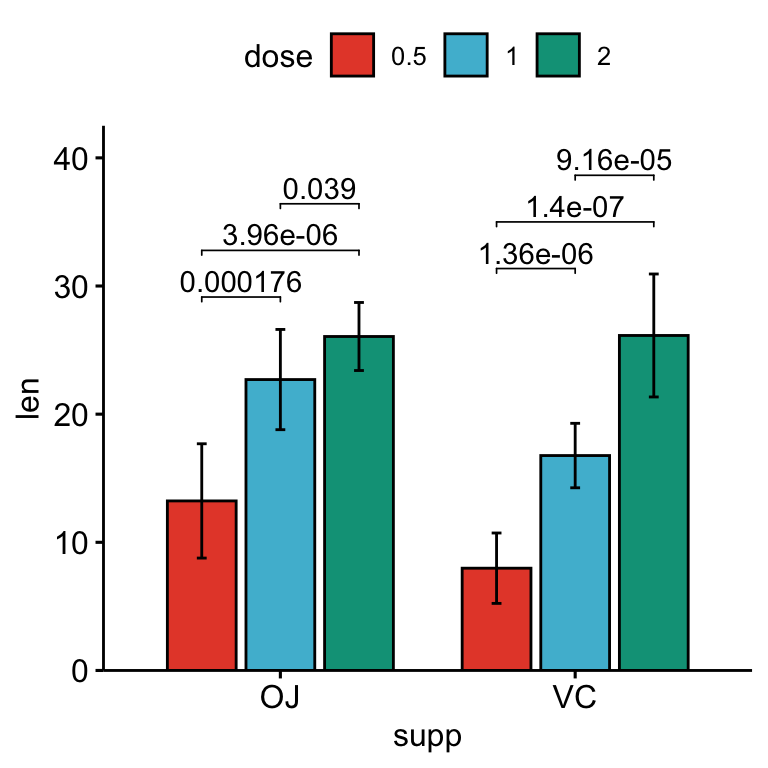
Comparaisons par paires par rapport à un groupe de référence
Tests statistiques:
stat.test <- df %>%
group_by(supp) %>%
t_test(len ~ dose, ref.group = "0.5")
stat.test## # A tibble: 4 x 11
## supp .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 OJ len 0.5 1 10 10 -5.05 17.7 0.0000878 0.0000878 ****
## 2 OJ len 0.5 2 10 10 -7.82 14.7 0.00000132 0.00000264 ****
## 3 VC len 0.5 1 10 10 -7.46 17.9 0.000000681 0.000000681 ****
## 4 VC len 0.5 2 10 10 -10.4 14.3 0.0000000468 0.0000000936 ****# Box plot avec p-values
stat.test <- stat.test %>%
add_xy_position(x = "supp", dodge = 0.8)
bxp +
stat_pvalue_manual(
stat.test, label = "p.adj", tip.length = 0.01,
bracket.nudge.y = -2
) +
scale_y_continuous(expand = expansion(mult = c(0, 0.1)))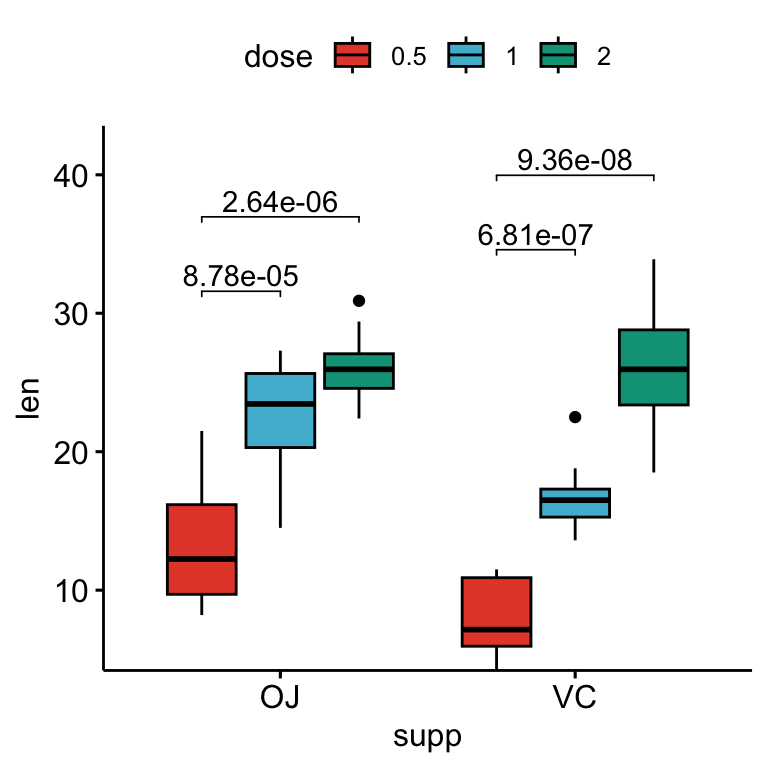
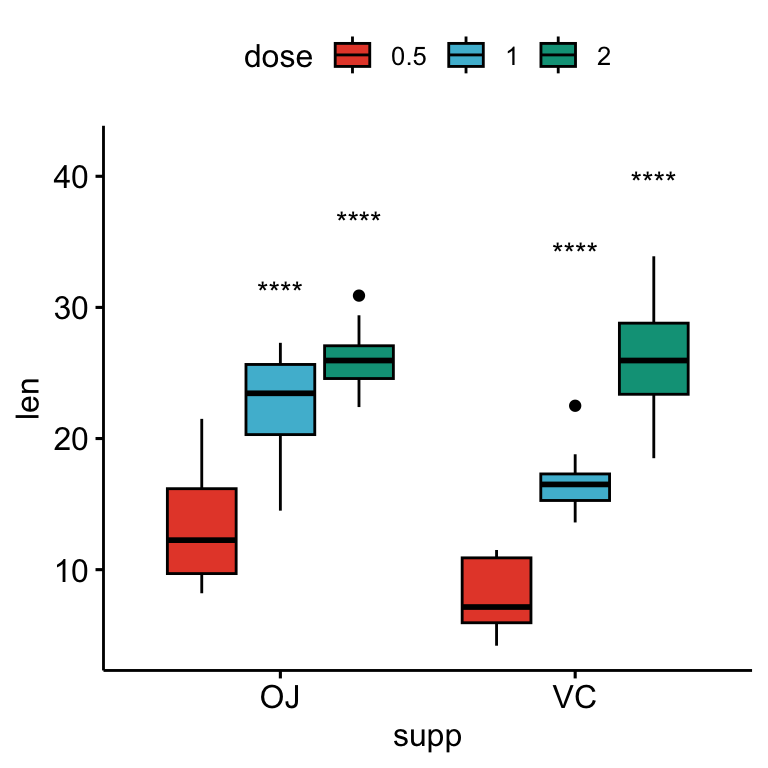
# Afficher uniquement les niveaux de significativité
# Faites baisser les symboles de significativité en utilisant vjust
bxp + stat_pvalue_manual(
stat.test, x = "supp", label = "p.adj.signif",
tip.length = 0.01, vjust = 2
)
# Bar plot avec p-values
stat.test <- stat.test %>%
add_xy_position(x = "supp", fun = "mean_sd", dodge = 0.8)
bp +
stat_pvalue_manual(
stat.test, label = "p.adj", tip.length = 0.01,
bracket.nudge.y = -2
) +
scale_y_continuous(expand = expansion(mult = c(0, 0.1)))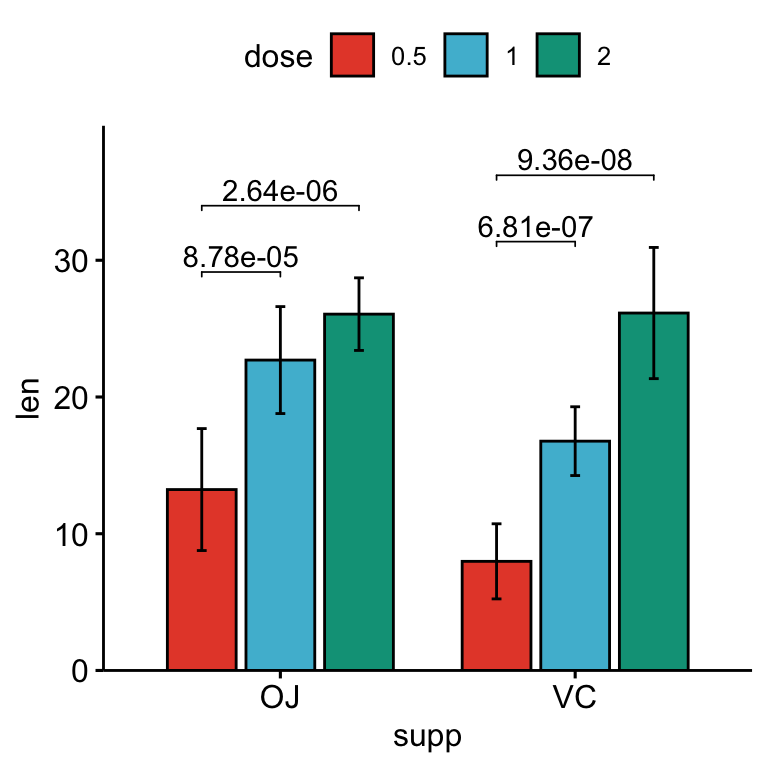
Conclusion
Cet article décrit comment ajouter des p-values sur des ggplots groupés, tels que les box plots, les bar plots et les line plots. Voir les autres questions fréquemment posées : ggpubr FAQ.
Version:
 English
English






No Comments