Cet article décrit comment ajouter des p-values générées ailleurs à un ggplot en utilisant le package ggpubr. Les fonctions clés suivantes de ggpubr seront utilisées:
stat_pvalue_manual(): Ajouter manuellement des p-values à un ggplot, tels que des box blots, des points et des stripcharts.geom_bracket(): Ajouter des crochets avec annotations à un ggplot. Aides pour ajouter une p-value ou des niveaux de significativité à un graphique.
Vous apprendrez à:
- Ajouter des p-values personnalisées créées ailleurs
- Ajouter les p-values obtenues à partir du package R rstatix
- Ajouter des crochets avec des étiquettes de p-value personnalisées à un ggplot
Sommaire:
Prérequis
Assurez-vous d’avoir les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixcontient des fonctions R facilitant les analyses statistiques
Charger les packages R requis:
library(tidyverse)
library(ggpubr)
library(rstatix)Préparation des données
# Convertir la variable `dose` en facteur
df <- ToothGrowth
df$dose <- factor(df$dose)
# Inspecter les données
# Afficher deux lignes aléatoires par groupes
set.seed(123)
df %>% sample_n_by(dose, size = 2)## # A tibble: 6 x 3
## len supp dose
## <dbl> <fct> <fct>
## 1 10 VC 0.5
## 2 14.5 OJ 0.5
## 3 18.8 VC 1
## 4 25.8 OJ 1
## 5 29.4 OJ 2
## 6 23.6 VC 2Ajouter les p-values calculées ailleurs
Fonction R clé : stat_pvalue_manual() [dans le package ggpubr]
stat_pvalue_manual(data, label = NULL)data: un tableau de données contenant des résultats de tests statistiques. Le format par défaut attendu doit contenir les colonnes suivantes:group1 | group2 | p | y.position | etc.group1etgroup2sont les groupes qui ont été comparés.pest la p-value résultante.y.positionest la coordonnée y des p-values dans le graphique.label: la colonne contenant l’étiquette (par exemple :label = "p"oulabel = "p.adj"), où p est la p-value. Peut également être une expression qui peut être formatée par le packageglue. Par exemple, lorsque l’on spécifielabel = "t-test, p = {p}", l’expression{p}sera remplacée par sa valeur.
# p-values
stat.test <- tibble::tribble(
~group1, ~group2, ~p.adj,
"0.5", "1", 2.54e-07,
"0.5", "2", 1.32e-13,
"1", "2", 1.91e-05
)
stat.test## # A tibble: 3 x 3
## group1 group2 p.adj
## <chr> <chr> <dbl>
## 1 0.5 1 2.54e- 7
## 2 0.5 2 1.32e-13
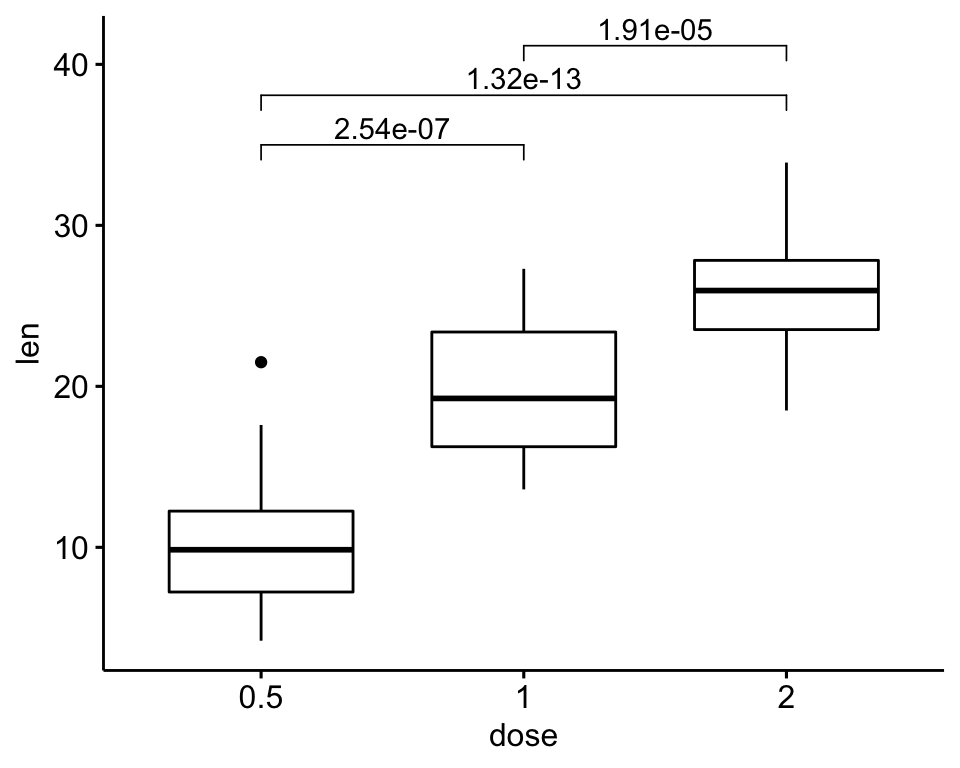
## 3 1 2 1.91e- 5# Box plots + p-values
ggboxplot(df, x = "dose", y = "len") +
stat_pvalue_manual(
stat.test,
y.position = 35, step.increase = 0.1,
label = "p.adj"
)
Ajouter les p-values obtenues à partir du package rstatix
Créer un box plot simple
bxp <- ggboxplot(df, x = "dose", y = "len")
bxp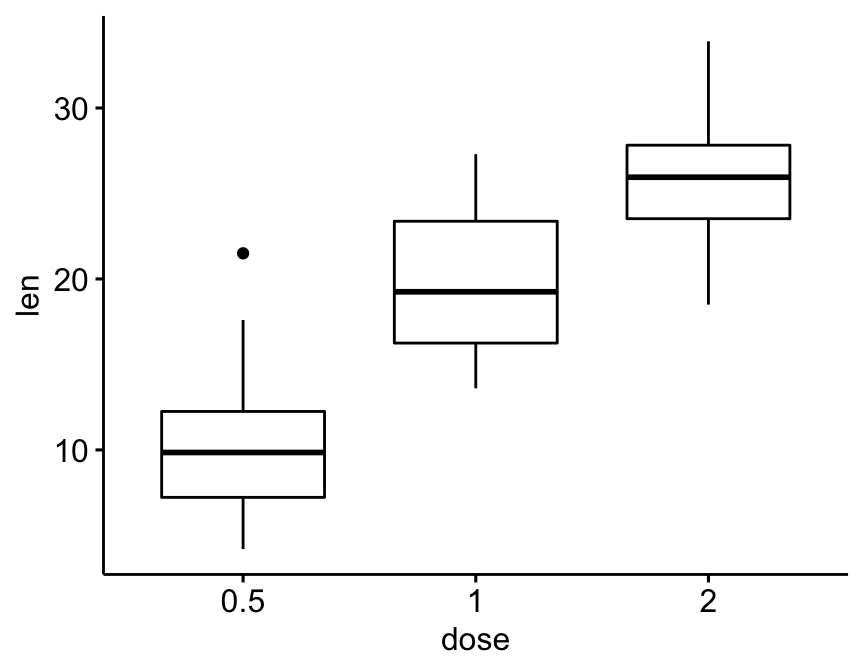
Comparaisons par paires
# Test statistique
stat.test <- df %>% t_test(len ~ dose)
stat.test## # A tibble: 3 x 10
## .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 len 0.5 1 20 20 -6.48 38.0 1.27e- 7 2.54e- 7 ****
## 2 len 0.5 2 20 20 -11.8 36.9 4.40e-14 1.32e-13 ****
## 3 len 1 2 20 20 -4.90 37.1 1.91e- 5 1.91e- 5 ****# Box plot
stat.test <- stat.test %>% add_xy_position(x = "dose")
bxp + stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01)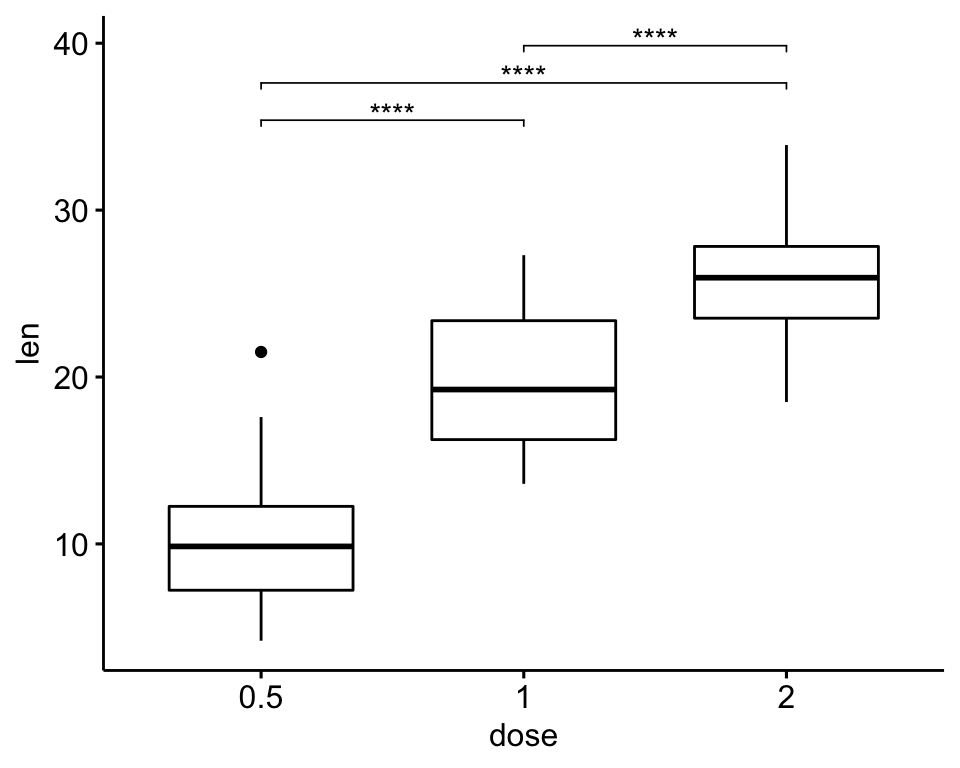
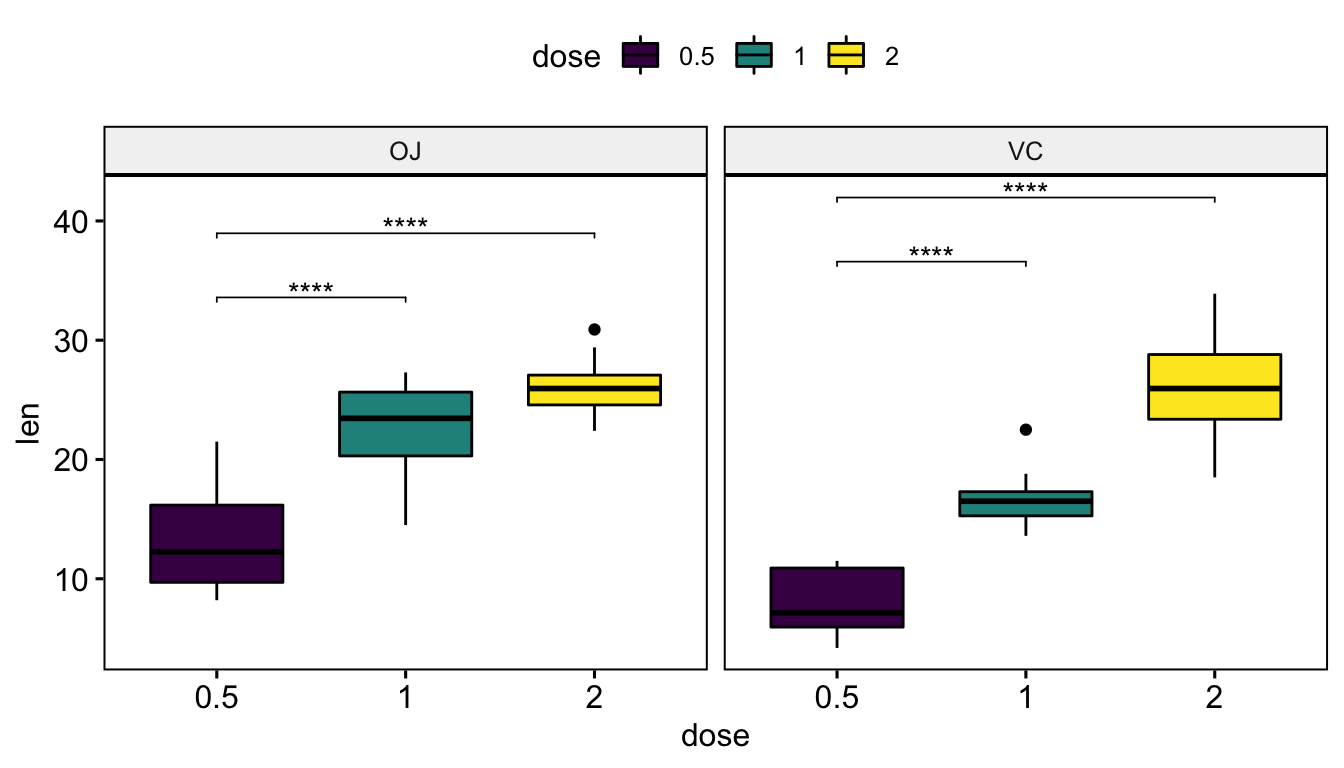
Graphiques à facettes
# Tests statistiques
stat.test <- df %>%
group_by(supp) %>%
t_test(len ~ dose, ref.group = "0.5")
stat.test## # A tibble: 4 x 11
## supp .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 OJ len 0.5 1 10 10 -5.05 17.7 0.0000878 0.0000878 ****
## 2 OJ len 0.5 2 10 10 -7.82 14.7 0.00000132 0.00000264 ****
## 3 VC len 0.5 1 10 10 -7.46 17.9 0.000000681 0.000000681 ****
## 4 VC len 0.5 2 10 10 -10.4 14.3 0.0000000468 0.0000000936 ****# Box plots
stat.test <- stat.test %>% add_xy_position(x = "dose")
ggboxplot(df, x = "dose", y = "len", facet.by = "supp") +
stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01)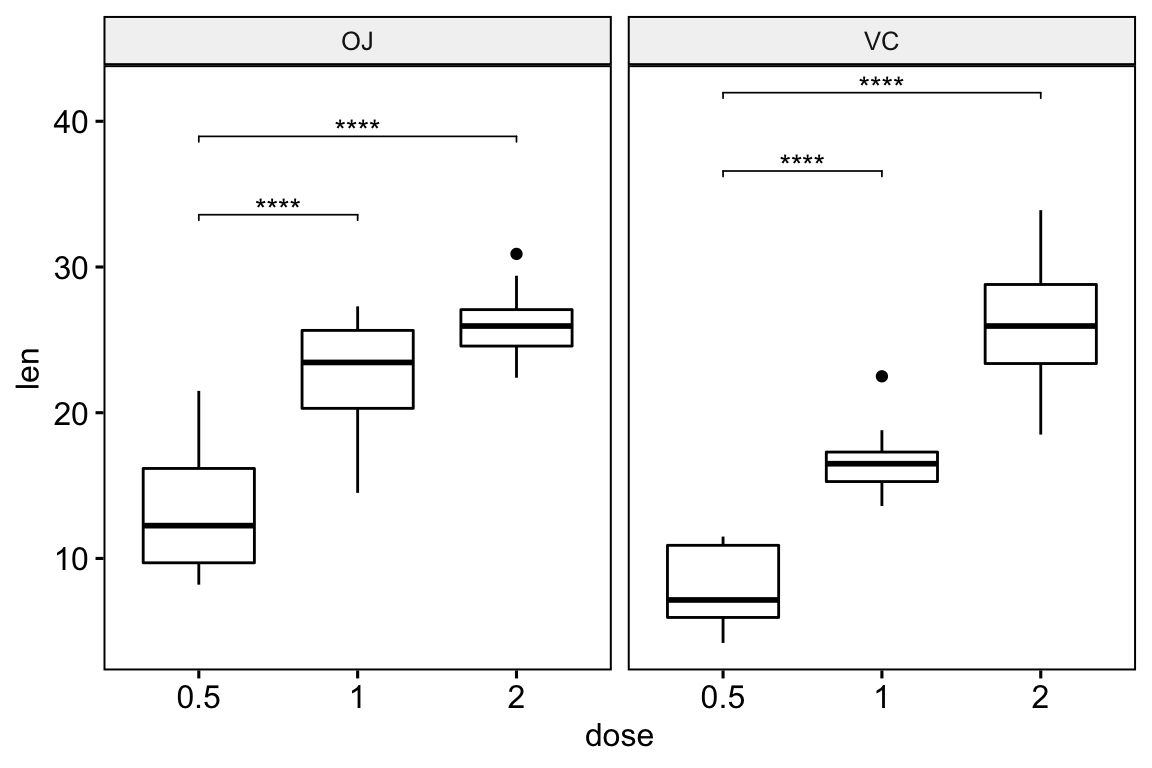
Plots groupés
# Box plot : comparaison par rapport à la référence
stat.test <- df %>%
group_by(supp) %>%
t_test(len ~ dose, ref.group = "0.5")
# Box plots
stat.test <- stat.test %>%
add_xy_position(x = "supp", dodge = 0.8)
bxp <- ggboxplot(df, x = "supp", y = "len", color = "dose")
bxp + stat_pvalue_manual(stat.test, label = "p.adj", tip.length = 0.01)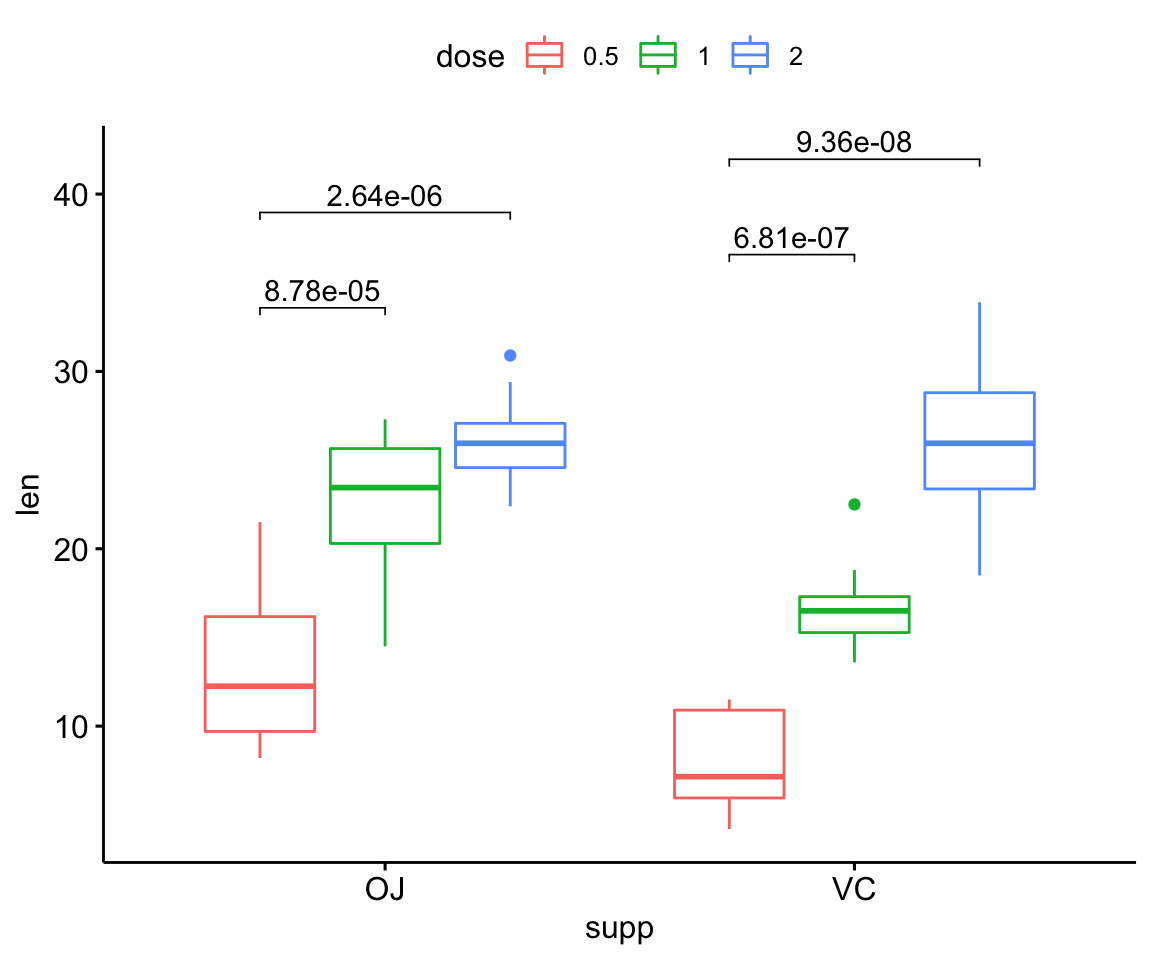
Préciser manuellement la position y des p-values
Créer un box plot simple:
# Test t par paire entre les groupes
stat.test <- ToothGrowth %>%
t_test(len ~ dose) %>%
mutate(y.position = c(29, 35, 39))
stat.test## # A tibble: 3 x 11
## .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif y.position
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 len 0.5 1 20 20 -6.48 38.0 1.27e- 7 2.54e- 7 **** 29
## 2 len 0.5 2 20 20 -11.8 36.9 4.40e-14 1.32e-13 **** 35
## 3 len 1 2 20 20 -4.90 37.1 1.91e- 5 1.91e- 5 **** 39# Créer un box plot et ajouter la p-value
ggboxplot(ToothGrowth, x = "dose", y = "len") +
stat_pvalue_manual(stat.test, label = "p.adj")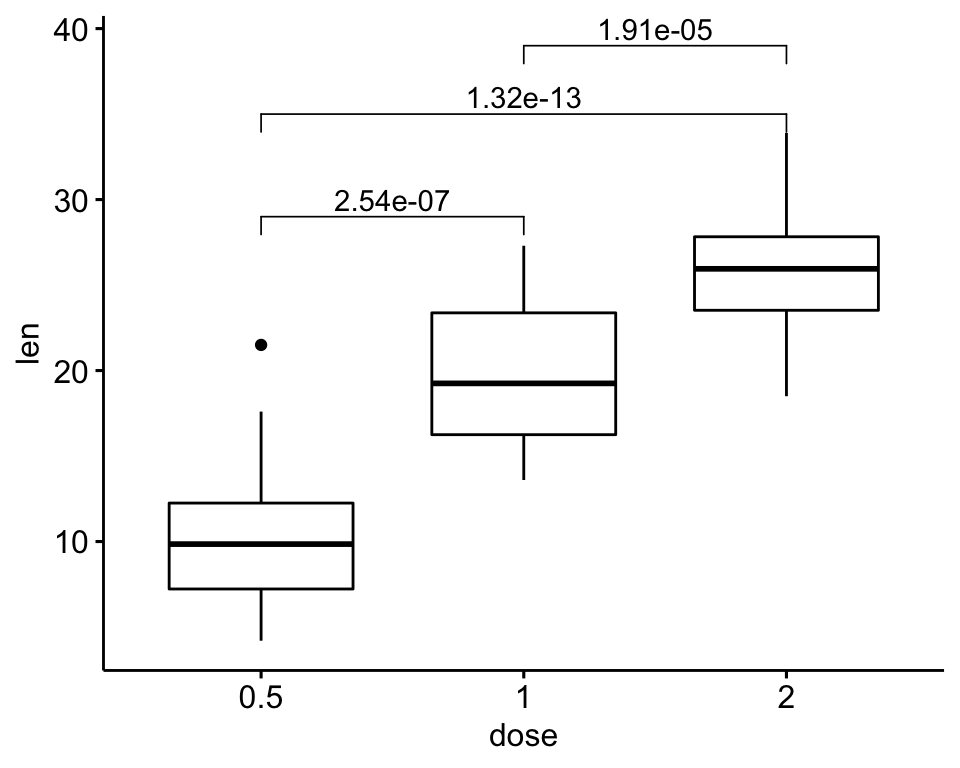
Graphiques à facettes : Comparaisons entre deux groupes
# Test t par paire entre les groupes
stat.test <- df %>%
group_by(dose) %>%
t_test(len ~ supp) %>%
adjust_pvalue() %>%
mutate(y.position = 35)
stat.test## # A tibble: 3 x 11
## dose .y. group1 group2 n1 n2 statistic df p p.adj y.position
## * <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 0.5 len OJ VC 10 10 3.17 15.0 0.00636 0.0127 35
## 2 1 len OJ VC 10 10 4.03 15.4 0.00104 0.00312 35
## 3 2 len OJ VC 10 10 -0.0461 14.0 0.964 0.964 35# Créer un box plot et ajouter la p-value
p <- ggboxplot(
df, x = "supp", y = "len",
color = "supp", palette = "jco",
facet.by = "dose", ylim = c(0, 40)
)
p + stat_pvalue_manual(stat.test, label = "p.adj")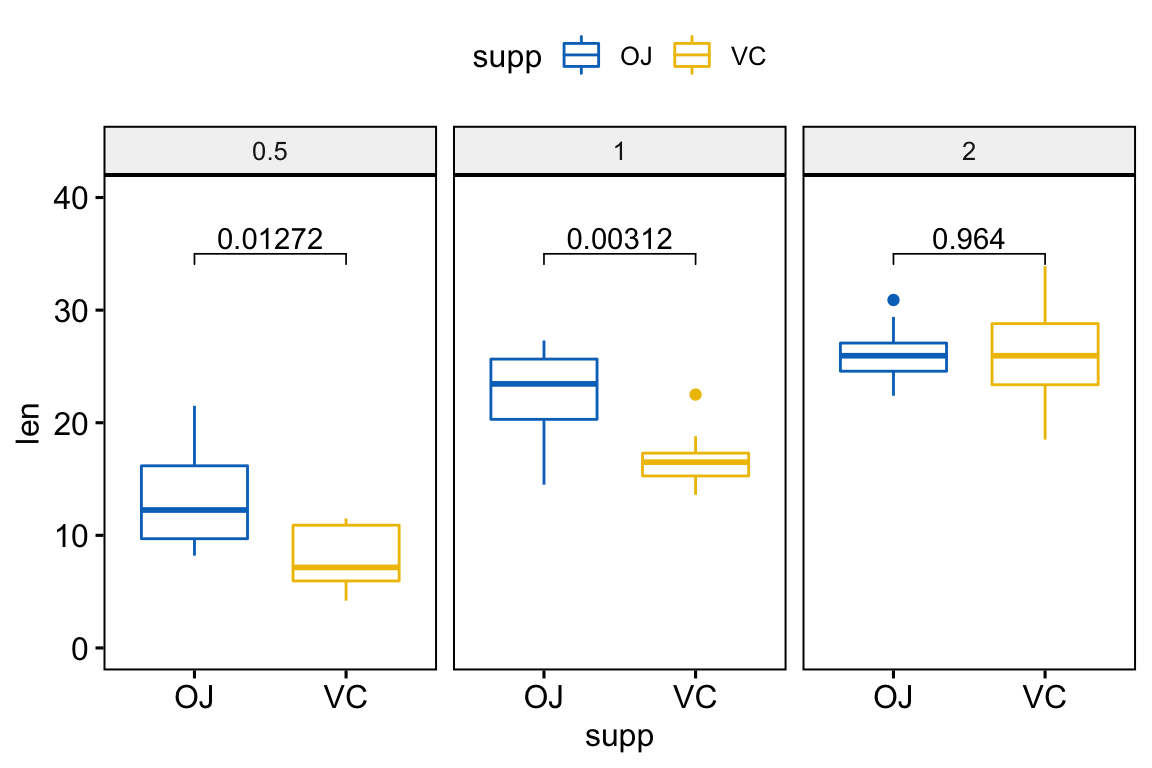
Graphiques à facettes : Comparaisons par paires entre plusieurs groupes
# Test t par paire entre les groupes
stat.test <- df %>%
group_by(supp) %>%
t_test(len ~ dose) %>%
adjust_pvalue() %>%
mutate(y.position = rep(c(29, 35, 39), 2))
stat.test## # A tibble: 6 x 12
## supp .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif y.position
## * <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 OJ len 0.5 1 10 10 -5.05 17.7 0.0000878 0.000263 *** 29
## 2 OJ len 0.5 2 10 10 -7.82 14.7 0.00000132 0.00000528 **** 35
## 3 OJ len 1 2 10 10 -2.25 15.8 0.039 0.039 * 39
## 4 VC len 0.5 1 10 10 -7.46 17.9 0.000000681 0.00000340 **** 29
## 5 VC len 0.5 2 10 10 -10.4 14.3 0.0000000468 0.000000281 **** 35
## 6 VC len 1 2 10 10 -5.47 13.6 0.0000916 0.000263 **** 39# Créer un box plot et ajouter la p-value
p <- ggboxplot(
df, x = "dose", y = "len",
color = "supp", palette = "jco",
facet.by = "supp", ylim = c(0, 40)
)
p + stat_pvalue_manual(stat.test)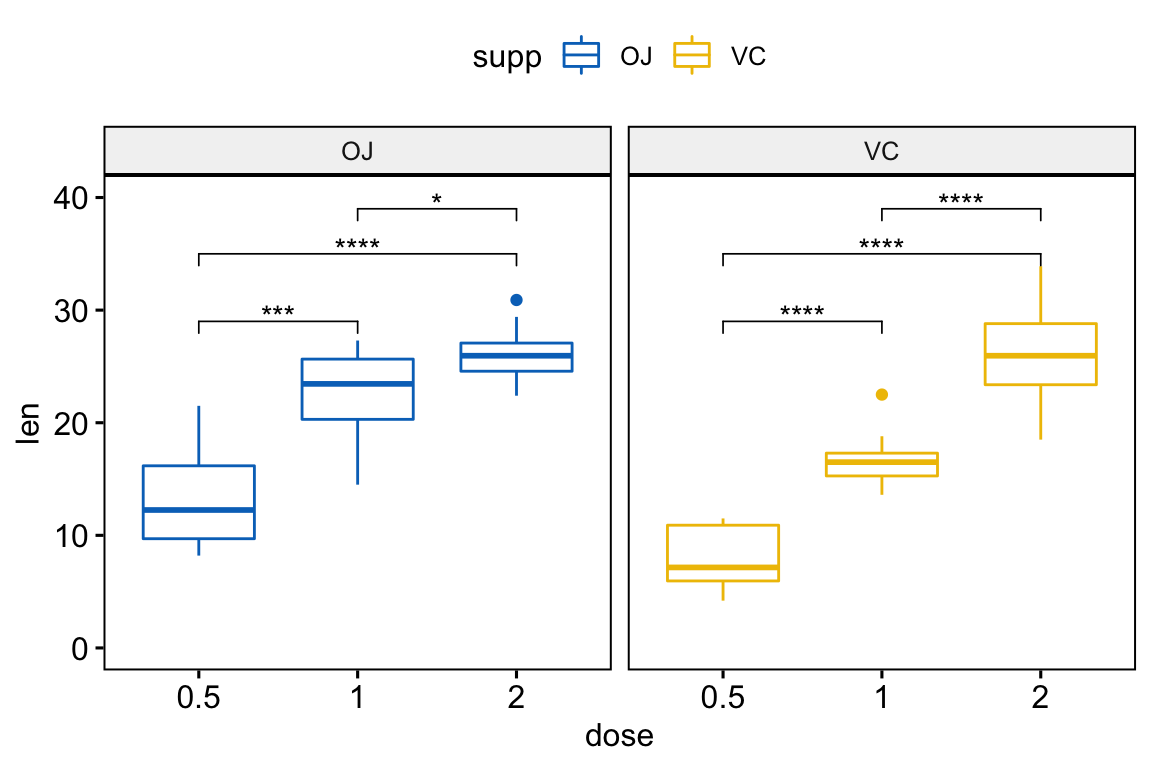
Plots groupés
# Test t par paire entre les groupes
stat.test <- df %>%
group_by(dose) %>%
t_test(len ~ supp) %>%
adjust_pvalue() %>%
mutate(y.position = 35)
stat.test## # A tibble: 3 x 11
## dose .y. group1 group2 n1 n2 statistic df p p.adj y.position
## * <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 0.5 len OJ VC 10 10 3.17 15.0 0.00636 0.0127 35
## 2 1 len OJ VC 10 10 4.03 15.4 0.00104 0.00312 35
## 3 2 len OJ VC 10 10 -0.0461 14.0 0.964 0.964 35# Créer un box plot et ajouter la p-value
p <- ggboxplot(
ToothGrowth, x = "dose", y = "len",
color = "supp", palette = "jco",
ylim = c(0, 40)
)
p + stat_pvalue_manual(stat.test, xmin = "dose", xmax = NULL)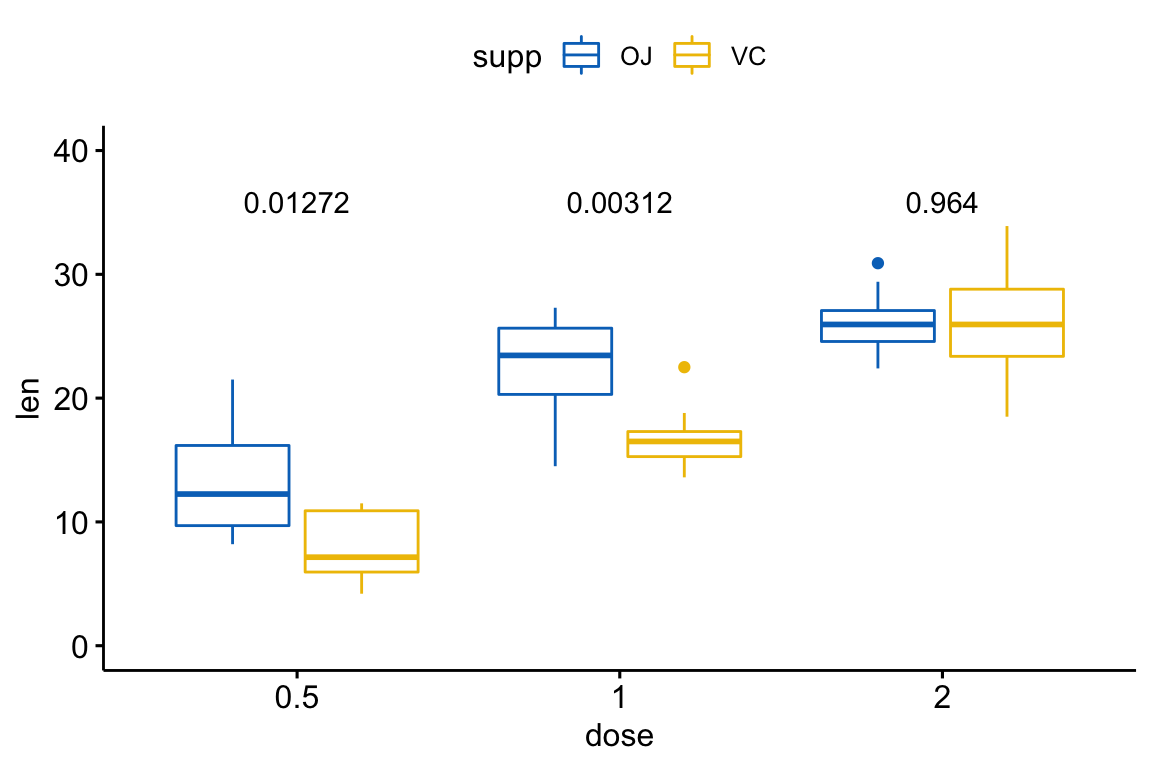
Ajouter des crochets avec des étiquettes de p-value personnalisées à un ggplot
Cette section décrit la fonction geom_bracket() [dans le package ggpubr] pour ajouter des crochets avec annotations dans un graphique ggplot. Il permet d’ajouter facilement à un graphique des p-values ou des niveaux de significativités statistiques créés ailleurs.
Crochets de base avec étiquettes
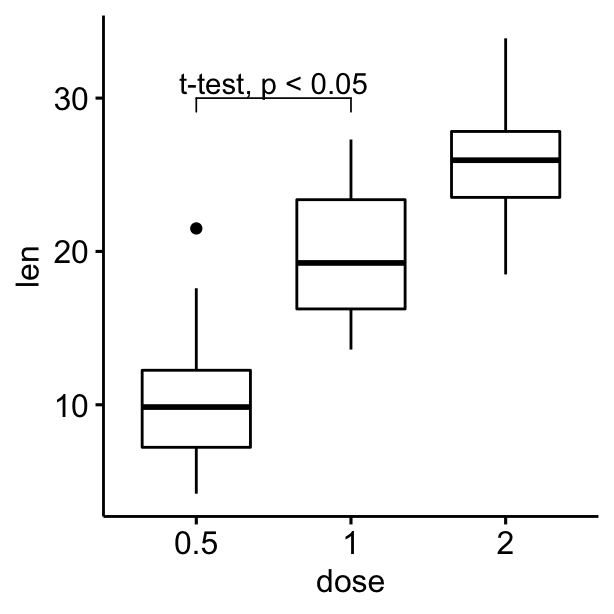
# Ajouter un crochet avec des étiquettes
ggboxplot(df, x = "dose", y = "len") +
geom_bracket(
xmin = "0.5", xmax = "1", y.position = 30,
label = "t-test, p < 0.05"
)
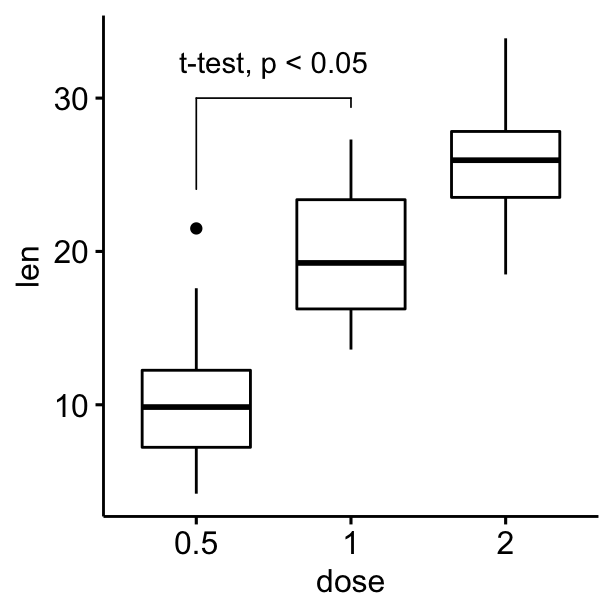
# Personnalisation de la longueur du crochet avec l'option tip.length
# Remonter l'étiquette en utilisant vjust
ggboxplot(df, x = "dose", y = "len") +
geom_bracket(
xmin = "0.5", xmax = "1", y.position = 30,
label = "t-test, p < 0.05",
tip.length = c(0.2, 0.02), vjust = -1
)
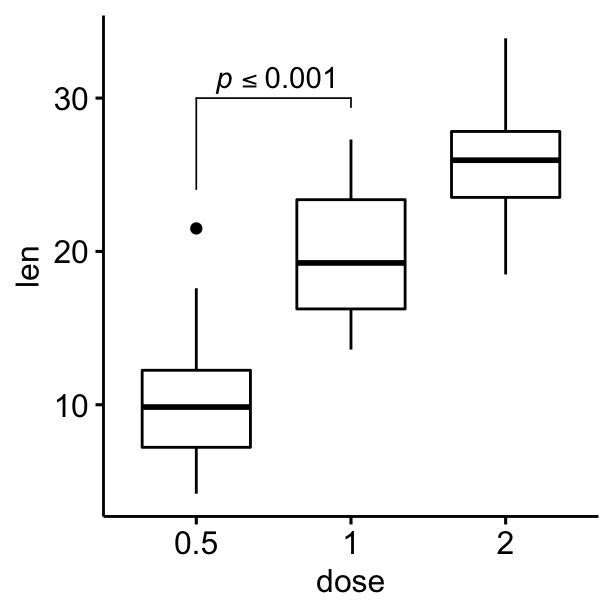
Utilisation de l’expression “plotmath” et spécification manuelle de plusieurs crochets
#Utilisation de l'expression plotmath
ggboxplot(df, x = "dose", y = "len") +
geom_bracket(
xmin = "0.5", xmax = "1", y.position = 30,
label = "list(~italic(p)<=0.001)", type = "expression",
tip.length = c(0.2, 0.02)
)
# Spécifiez manuellement les crochets multiples
ggboxplot(df, x = "dose", y = "len") +
geom_bracket(
xmin = c("0.5", "1"), xmax = c("1", "2"),
y.position = c(30, 35), label = c("***", "**"),
tip.length = 0.01
)
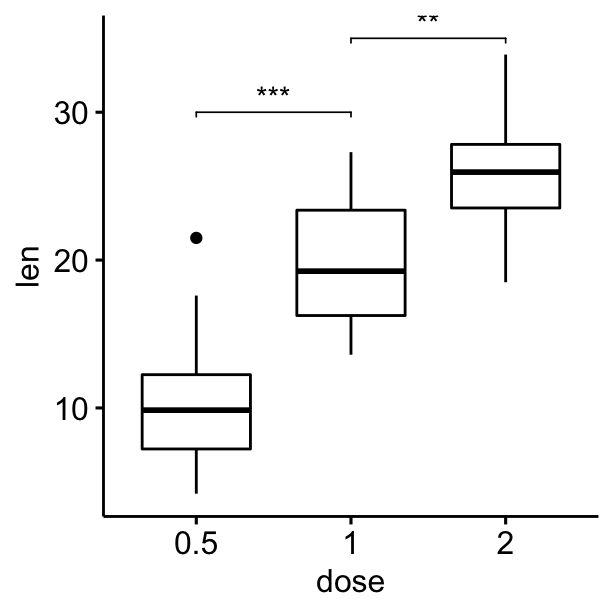
Calculer des tests statistiques et ajouter des p-values
# Calculer des tests statistiques et ajouter des p-values
stat.test <- compare_means(len ~ dose, ToothGrowth, method = "t.test")
ggboxplot(df, x = "dose", y = "len") +
geom_bracket(
aes(xmin = group1, xmax = group2, label = signif(p, 2)),
data = stat.test, y.position = 35
)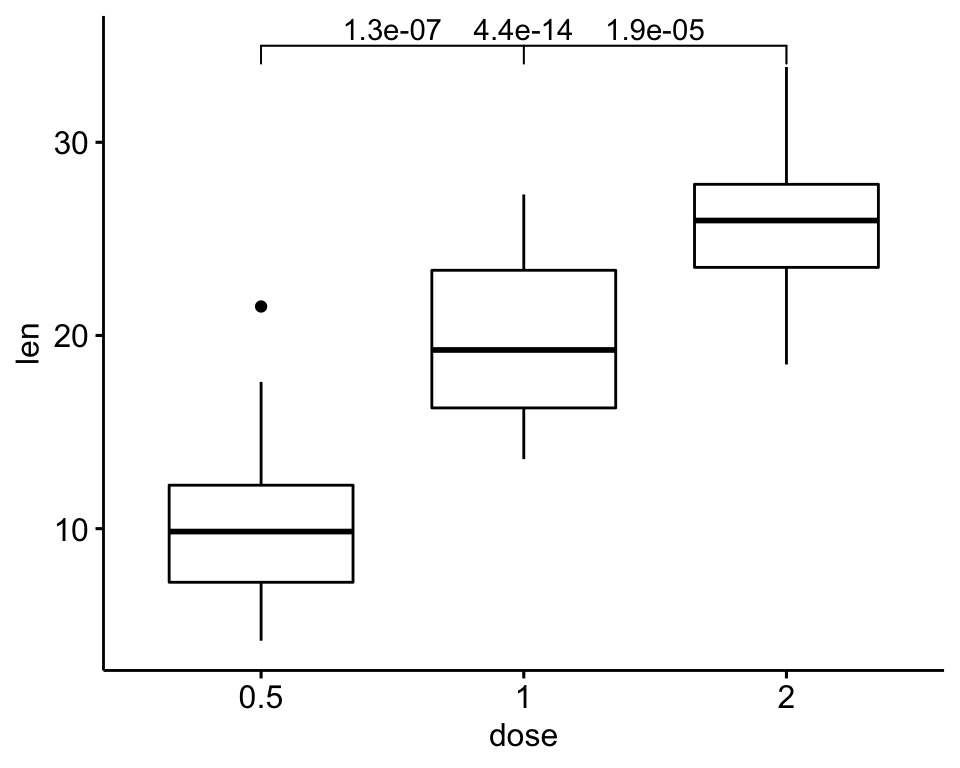
# Augmenter la longueur des pas entre les crochets
ggboxplot(df, x = "dose", y = "len") +
geom_bracket(
aes(xmin = group1, xmax = group2, label = signif(p, 2)),
data = stat.test, y.position = 35, step.increase = 0.1
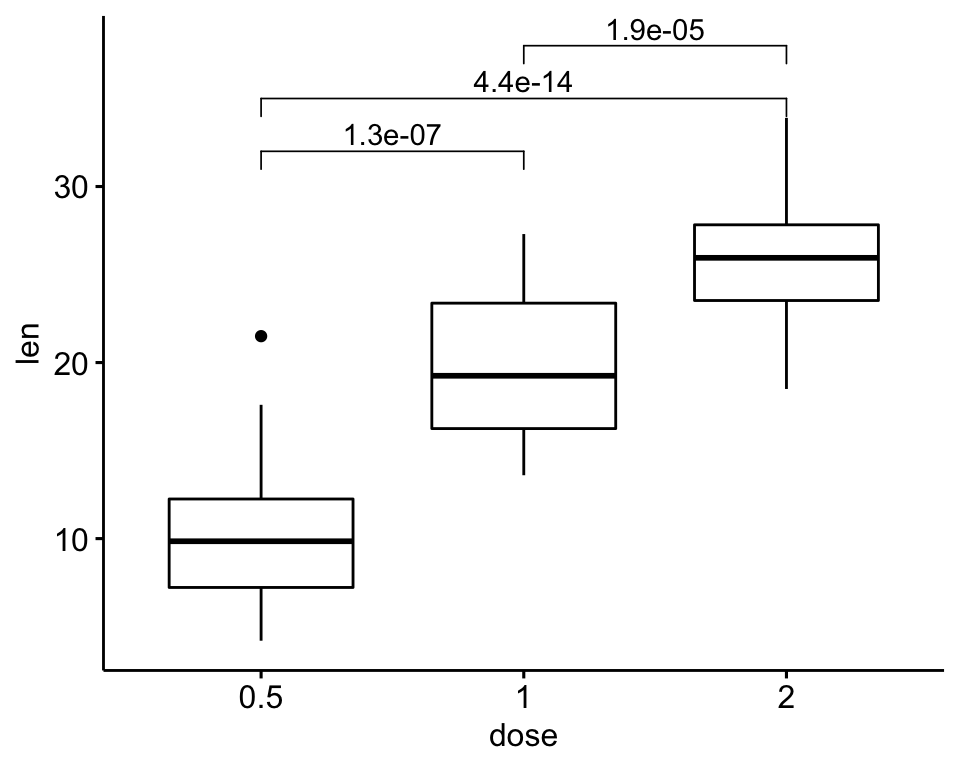
)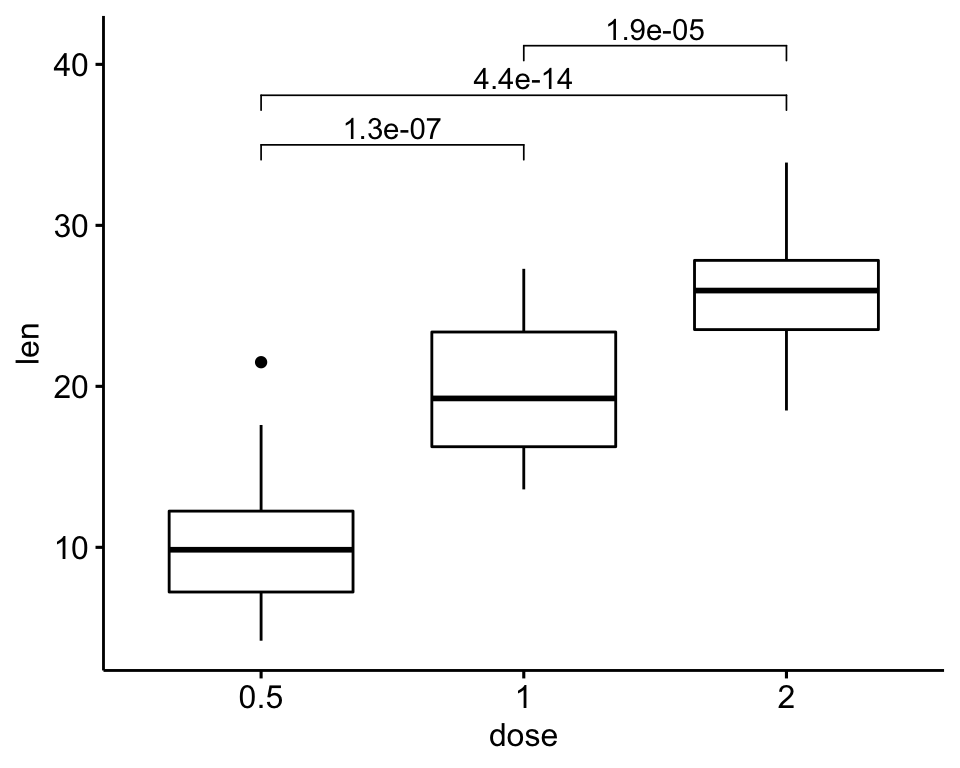
# Ou préciser les positions de chaque comparaison
ggboxplot(df, x = "dose", y = "len") +
geom_bracket(
aes(xmin = group1, xmax = group2, label = signif(p, 2)),
data = stat.test, y.position = c(32, 35, 38)
)
Conclusions
Cet article décrit comment ajouter des p-values générées ailleurs à un ggplot en utilisant le package ggpubr.
Version:
 English
English






No Comments