Cet article décrit les méthodes de sériation, qui consistent à trouver un ordre linéaire approprié pour un ensemble d’objets dans les données en utilisant des fonctions de perte (loss) ou de mérite.
Il existe différents algorithmes de sériation. Les données d’entrée peuvent être soit une matrice de dissimilarité, soit une matrice de données standard.
Vous apprendrez à effectuer la sériation dans R et à visualiser les données réorganisées à l’aide du package R seriation.
Sommaire:
Prérequis
Charger les packages R requis:
library(seriation)Préparation des données
Données de démonstration: iris
# Charger les données
data("iris")
df <- iris
head(df, 2)## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa# Supprimer la colonne `species` (colonne 5)
df <- df[, -5]
# Réorganiser les objets au hasard
set.seed(123)
df <- df[sample(seq_len(nrow(df))),]
head(df, 2)## Sepal.Length Sepal.Width Petal.Length Petal.Width
## 44 5.0 3.5 1.6 0.6
## 118 7.7 3.8 6.7 2.2Exemple basic de sériation
Réorganiser les objets et inspecter l’effet de la sériation sur la matrice de dissimilarité:
# Calculer la matrice de dissimilarité
dist_result <- dist(df)
# Sérier les objets, réorganiser les lignes en fonction de leur similarité
object_order <- seriate(dist_result)
# Extraire l'ordre des objets
head(get_order(object_order), 15)## [1] 78 2 52 83 76 139 148 32 59 20 129 103 143 4 85# Visualiser l'effet de la sériation sur la matrice de dissimilarité
pimage(dist_result, main = "Random order")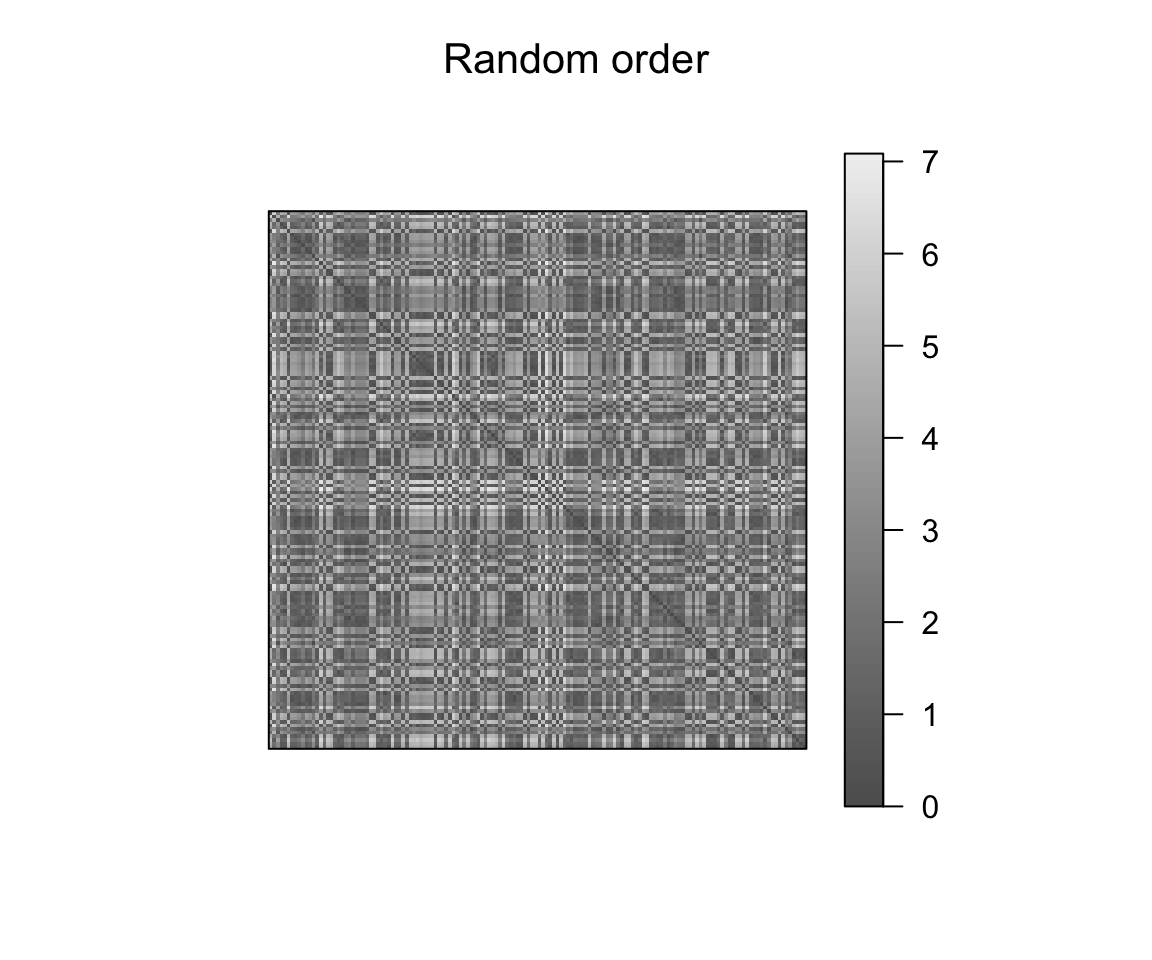
pimage(dist_result, order = object_order, main = "Reordered")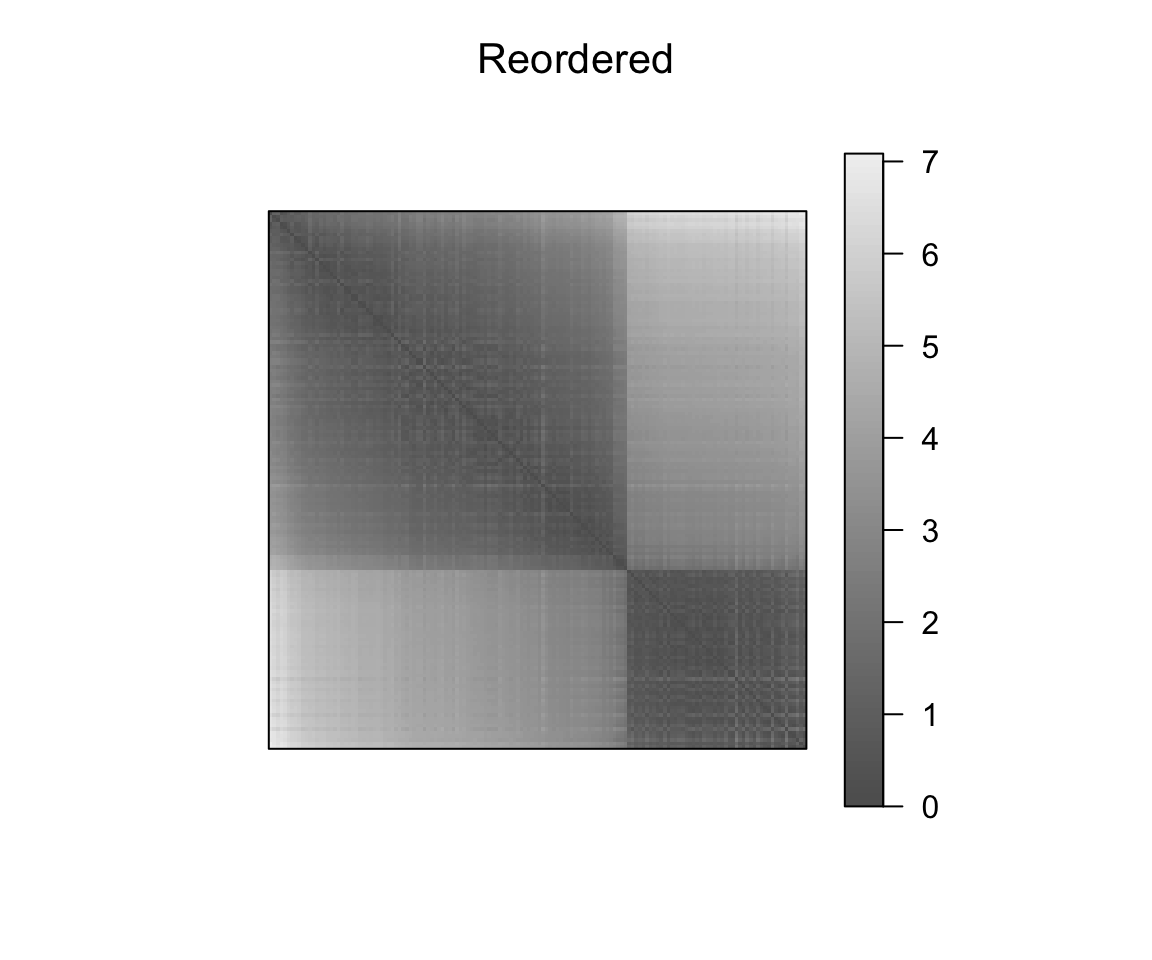
Effet de la sériation sur l’échelle des données d’origine. Nous normalisons les données en utilisant la fonction scale() telle que la valeur visualisée est le nombre de d’écart type qu’un objet diffère de la moyenne variable. Pour les matrices contenant des valeurs négatives, pimage() utilise automatiquement une palette divergente.
Étant donné que la sériation ci-dessus n’a produit qu’un ordre pour les lignes des données, nous utilisons NA pour les ordres des colonnes dans le code R ci-dessous.
# Heatmap des données brutes
pimage(scale(df), main = "Random")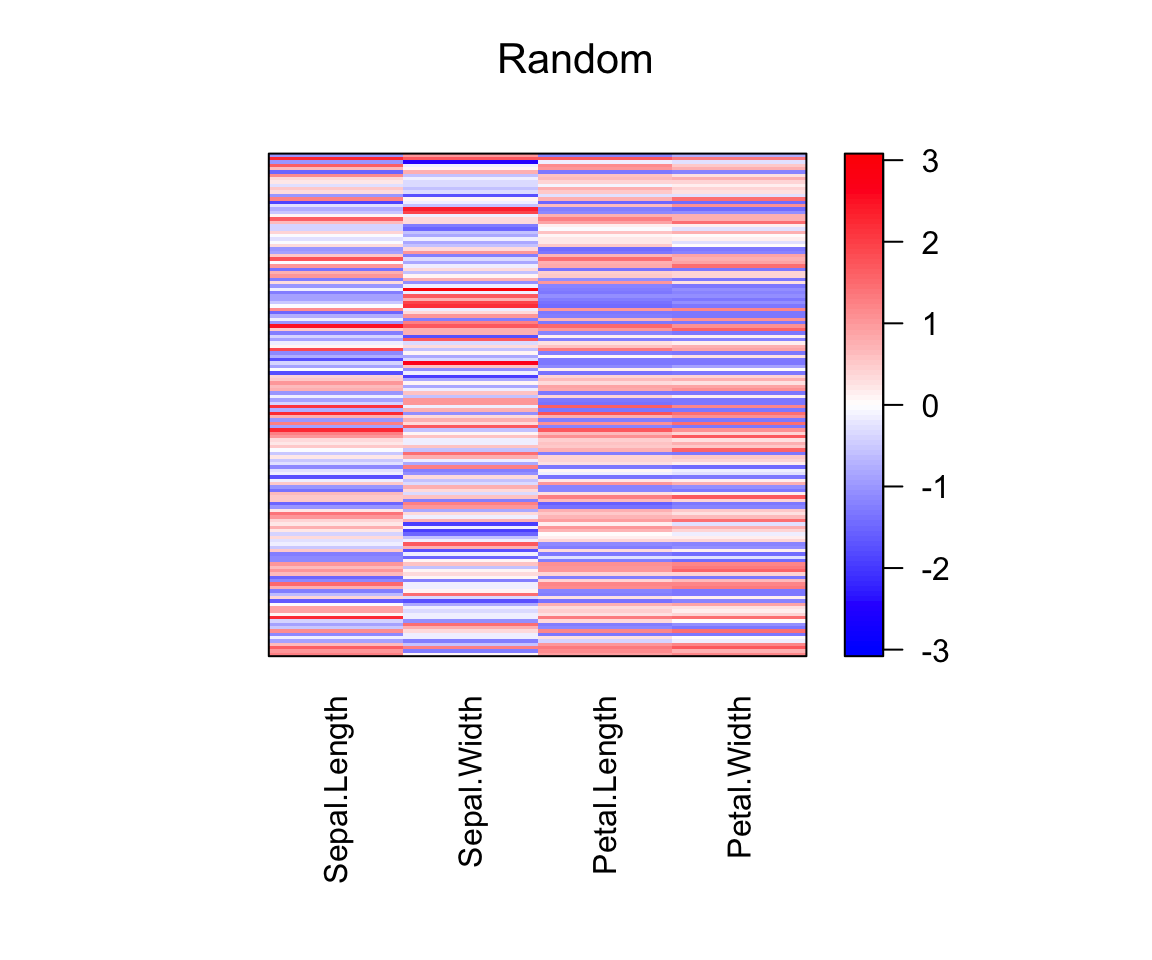
# Heatmap des données réorganisées
pimage(scale(df), order = c(object_order, NA), main = "Reordered")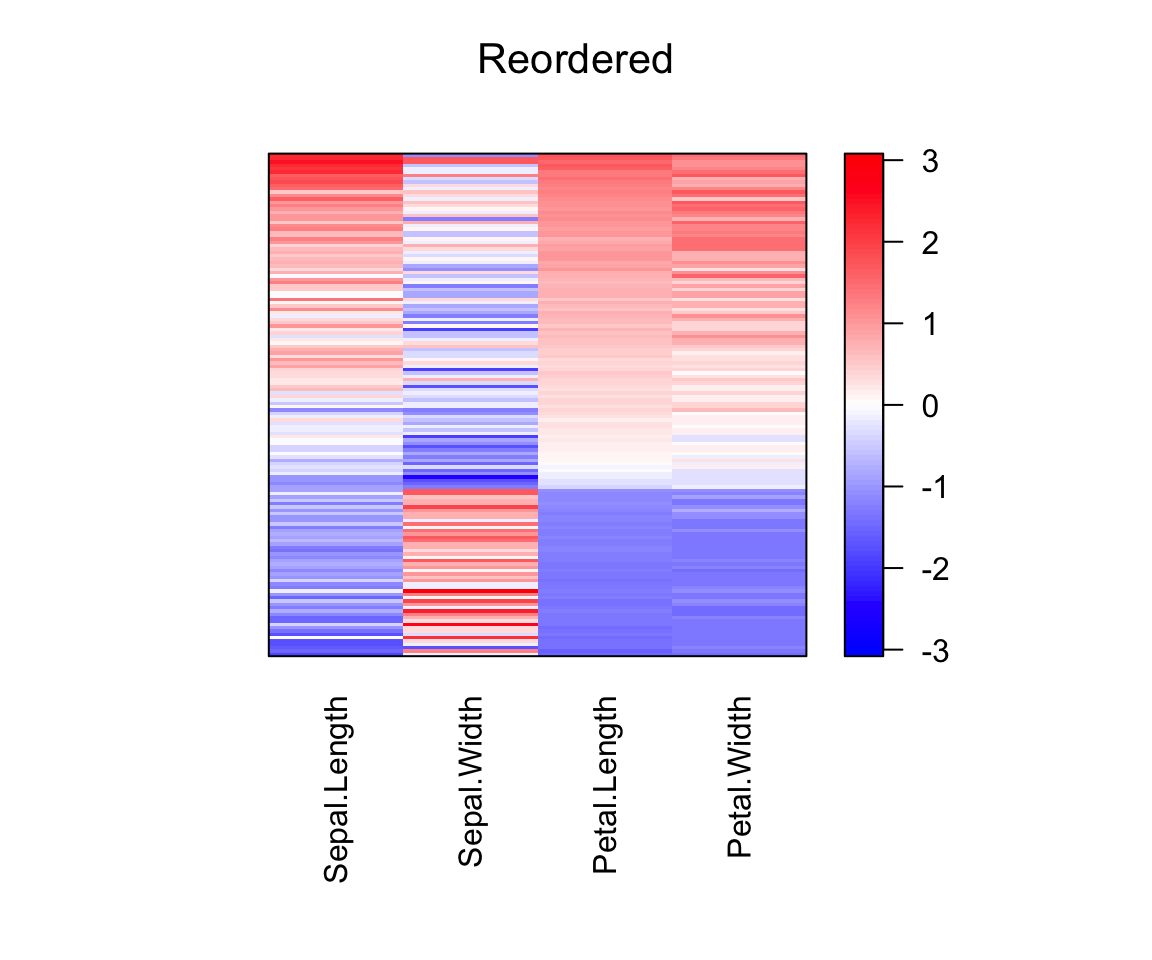
Heat maps
Une Heat Map est une matrice de données codées en couleur, avec un dendrogramme ajouté sur un côté et en haut pour indiquer l’ordre des lignes et des colonnes.
En règle générale, le réarrangement se fait en fonction des moyennes des lignes ou des colonnes dans les limites imposées par le dendrogramme.
Il est possible de trouver l’ordre optimal des nœuds des feuilles d’un dendrogramme qui minimise les distances entre les objets adjacents. Un tel ordre pourrait apporter une amélioration visuelle par rapport à l’utilisation d’un simple réarrangement tel que les moyennes de lignes ou de colonnes.
La fonction R hmap() [package seriation] utilise un ordre optimal et peut également utiliser la sériation directement sur des matrices de distance sans utiliser de clustering hiérarchique pour produire d’abord des dendrogrammes. Il utilise la fonction gplots::heatmap.2() pour créer la heatmap.
Pour l’exemple suivant, nous utilisons à nouveau le jeu de données iris réorganisé de façon aléatoire à partir des exemples précédents. Pour rendre les variables (colonnes) comparables, nous utilisons une normalisation standard.
# Standardiser les données
df_scaled <- scale(df, center = FALSE)
# Produire une heatmap avec des dendrogrammes réordonnés de manière optimale
# Le clustering hiérarchique est utilisé pour produire des dendrogrammes
hmap(df_scaled, margin = c(7, 4), cexCol = 1, labRow = FALSE)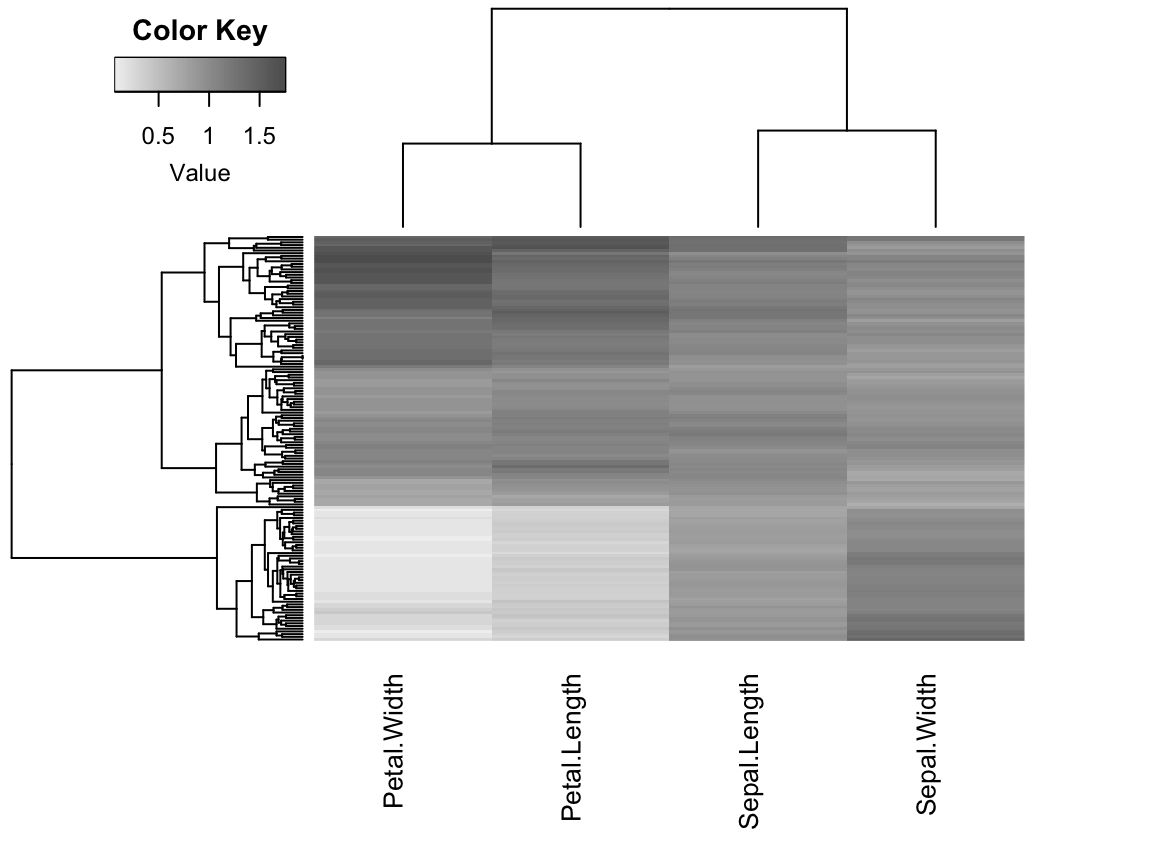
# Préciser la méthode de sériation
# la sériation sur les matrices de dissimilarité pour les lignes et les colonnes est effectuée
hmap(df_scaled, method = "MDS")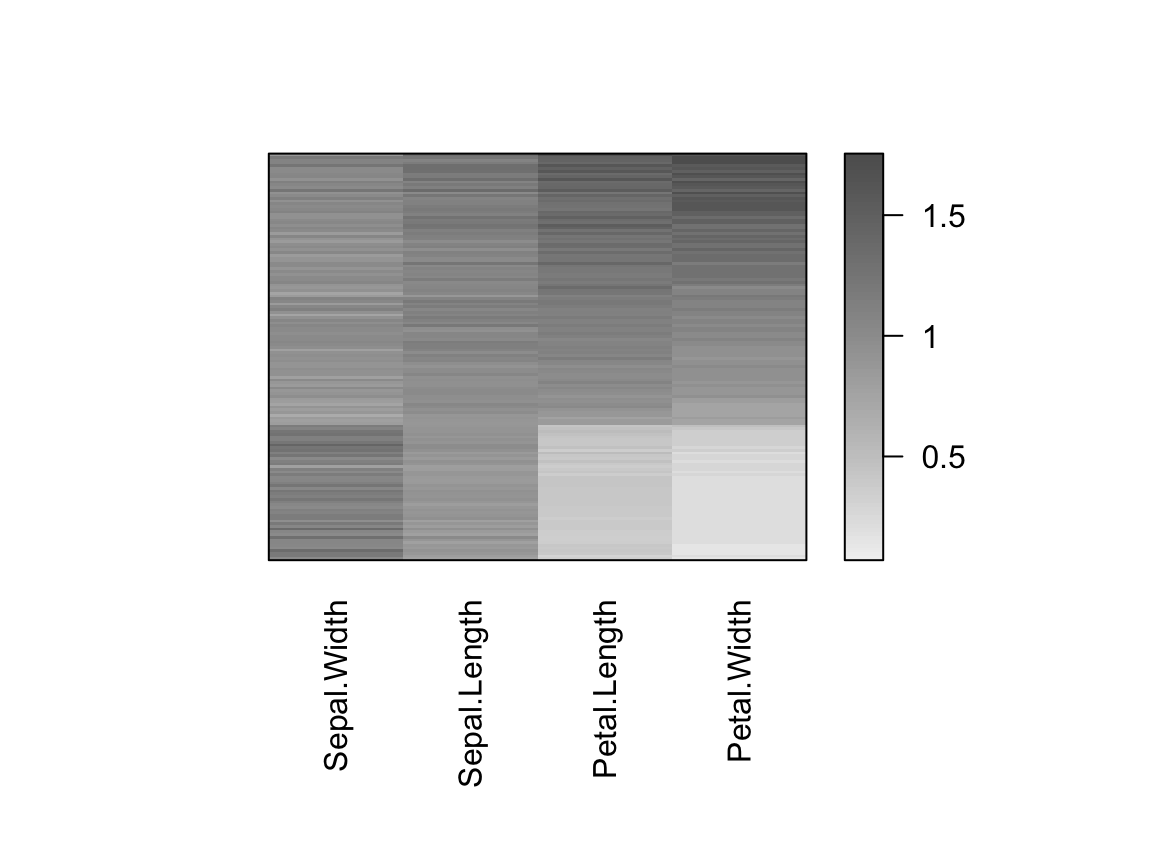
Matrice de permutation de Bertin
L’idée est de révéler une structure plus homogène dans une matrice de données en réorganisant simultanément les lignes et les colonnes. La matrice réorganisée est affichée et, les cas et les variables peuvent être regroupés manuellement pour mieux comprendre les données.
À titre d’exemple, nous utilisons le jeu de données USArrests, qui contient les taux de criminalité des États américains en 1973. Pour rendre les valeurs comparables entre les colonnes (variables), les rangs des valeurs de chaque variable sont utilisés à la place des valeurs initiales.
Pour la sériation, nous calculons les distances entre les lignes et entre les colonnes en utilisant la somme des différences de rang absolues (ceci est égal à la distance de Minkowski avec la puissance 1).
Dans l’exemple ci-dessous, le résultat est illustré par une matrice de barres. Les valeurs élevées sont mises en évidence (blocs remplis). Les cas sont affichés sous forme de colonnes et les variables sous forme de lignes.
# Préparation des données
# Charger le jeu de données
data("USArrests")
# Remplacer les valeurs initiales par leurs rangs
df <- head(apply(USArrests, 2, rank), 30)
# Effectuer la sériation sur les lignes et les colonnes
row_order <- seriate(dist(df, "minkowski", p = 1), method ="TSP")
col_order <- seriate(dist(t(df), "minkowski", p = 1), method ="TSP")
orders <- c(row_order, col_order)
# Visualisation : matrice de barres
# Matrice originale
bertinplot(df)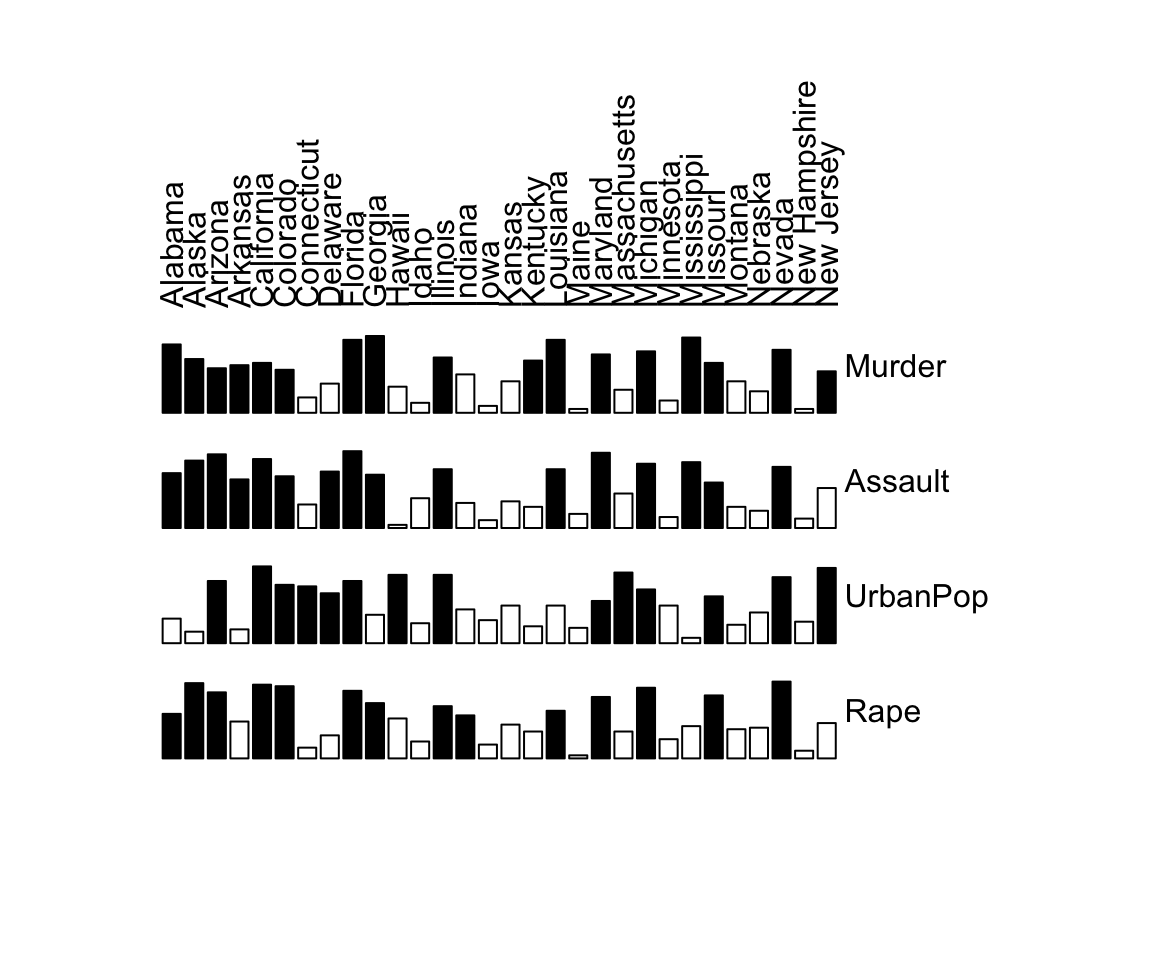
# Matrice réorganisée
bertinplot(df, orders)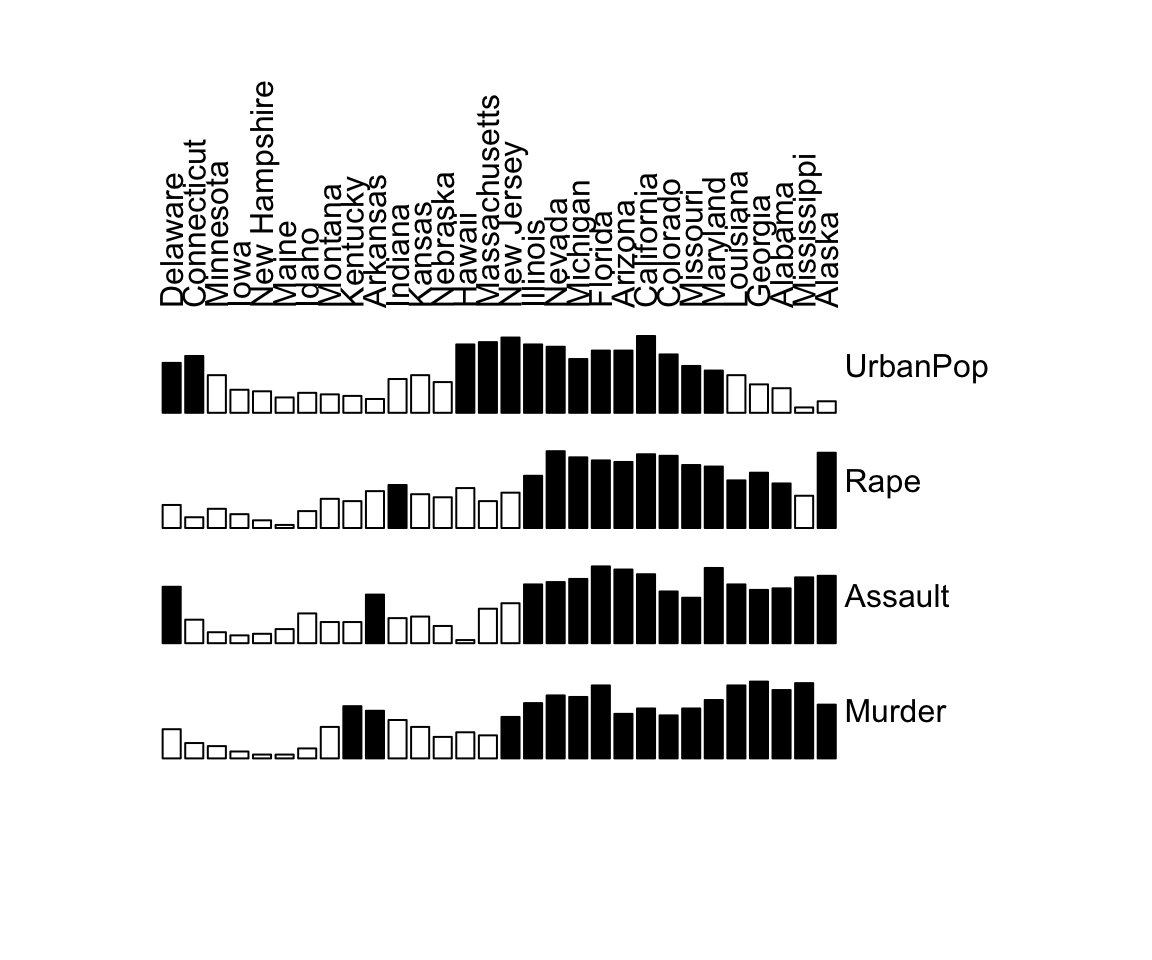
Matrices de données binaires
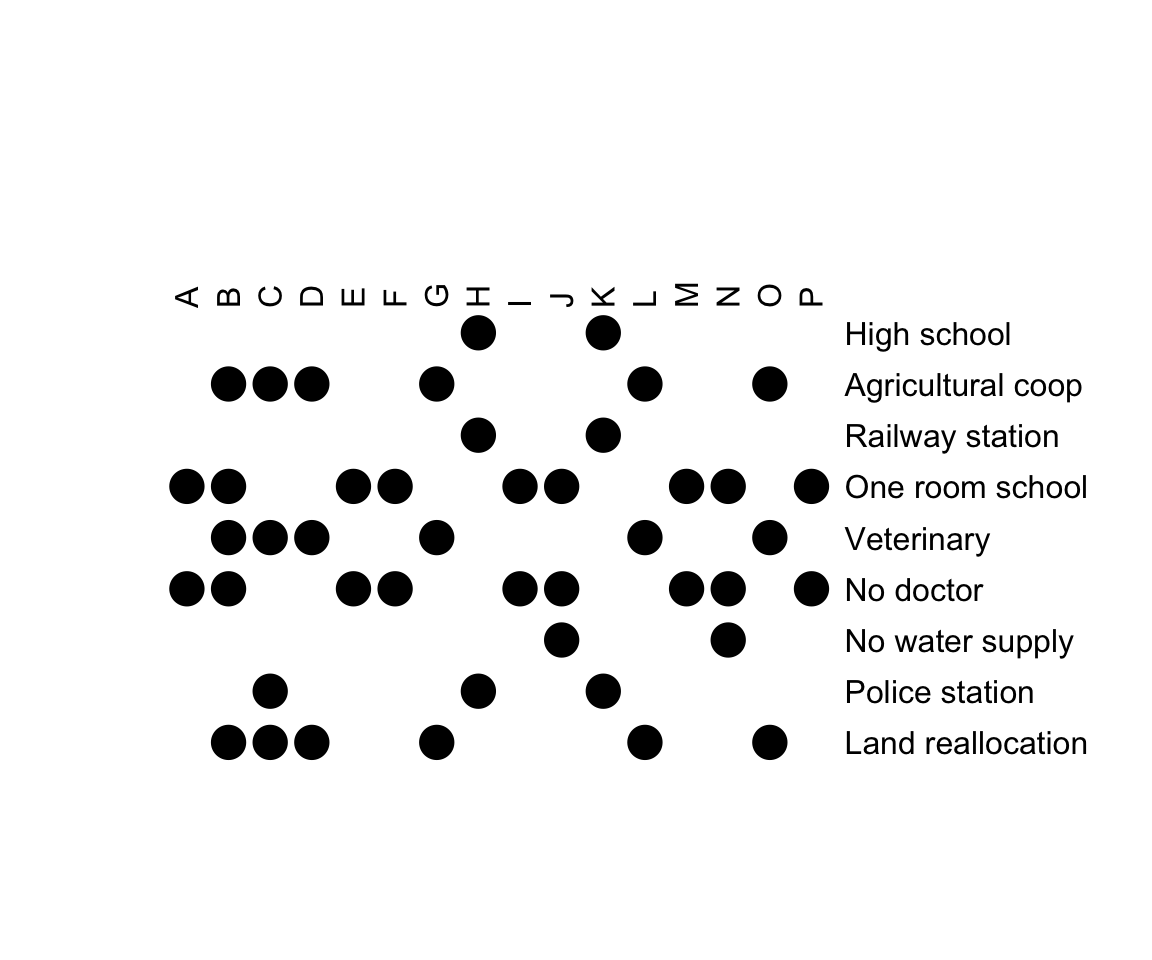
Les données binaires sont des matrices de données 0-1. La visualisation standard de bertinplot(), n’a pas beaucoup de sens pour les données binaires. Nous pouvons utiliser les fonctions de panel panel.squares() ou panel.circles().
# Charger les données de démonstration
data("Townships")
# Visualiser les données originales
bertinplot(
Townships,
options = list(panel = panel.circles)
)
# Sérier les lignes et les colonnes en utilisant l'algorithme de l'énergie de liaison (BEA: bond energy algorithm)
set.seed(1234)
orders <- seriate(Townships, method = "BEA", control = list(rep = 10))
bertinplot(
Townships, order = orders,
options = list(panel = panel.circles)
)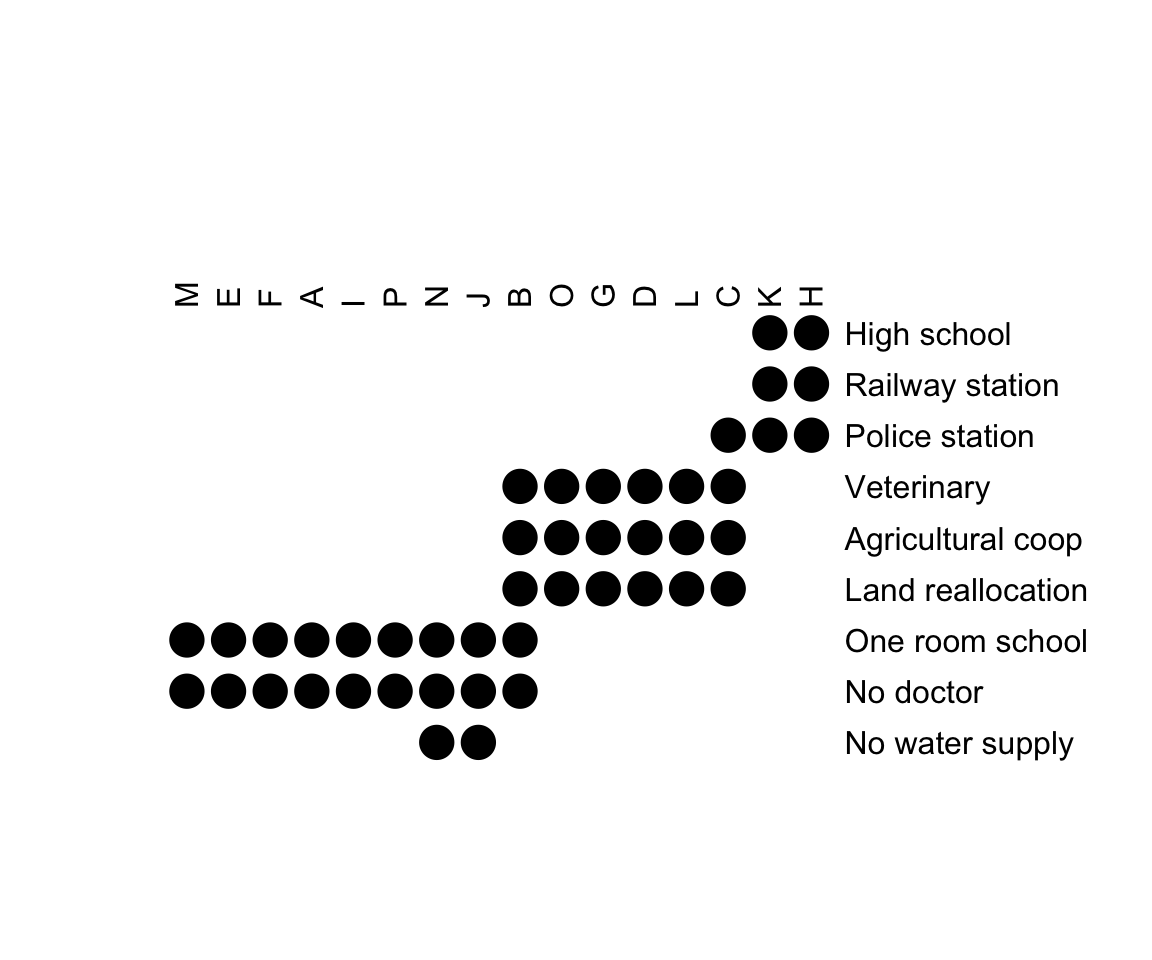
Une structure claire est visible dans la matrice réorganisée est affichée. Les variables peuvent être réparties en trois catégories décrivant différents états d’évolution des communes:
- Rurale : Pas de médecin, d’école à classe unique et peut-être aussi pas d’approvisionnement en eau
- Intermédiaire : Réallocation des terres, coopérative vétérinaire et agricole
- Urbain : Gare, lycée et poste de police
Les townships font aussi clairement partie de ces trois groupes qui peuvent être provisoirement appelés villages (7 premiers), villes (5 suivants) et cités (2 derniers). Les townships (“cantons”) B et C sont en transition vers le groupe supérieur suivant.
Résumé
Cet article décrit comment réorganiser les objets dans un jeu de données en utilisant des méthodes de sériation.
Références
- Michael Hahsler, Kurt Hornik et Christian Buchta, Getting Things in Order : An Introduction to the R Package seriation, Journal of Statistical Software, 25(3), 2008.
Version:
 English
English







No Comments