Cet article fournit des exemples de codes pour la visualisation des clusters K-means dans R en utilisant les packages R factoextra et ggpubr. Vous pouvez en apprendre davantage sur l’algorithme k-means en lisant le billet de blog suivant : K-means clustering in R : Step by Step Practical Guide.
Sommaire:
Packages R requis
- ggpubr : crée des graphiques prêts à être publiés.
- factoextra : Extraire et visualiser les résultats d’analyses de données multivariées.
library(ggpubr)
library(factoextra)Préparation des données
data("iris")
df <- iris
head(df, 3)## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosaExemple de calcul de clustering K-means
- Suppression de la 5ème colonne (
Species) et normalisation des données pour rendre les variables comparables - Calculer le clustering k-means en utilisant k = 3. Comme le résultat final de la classification k-means est sensible aux assignations initiales aléatoires, nous spécifions nstart = 25. Cela signifie que R va essayer 25 affectations différentes de départ aléatoire et ensuite sélectionner les meilleurs résultats.
# Calculer k-means avec k = 3
set.seed(123)
res.km <- kmeans(scale(df[, -5]), 3, nstart = 25)
# Clustering K-means montrant le groupe de chaque individu
res.km$cluster## [1] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 3 3 3 2 2 2 3 2 2 2
## [61] 2 2 2 2 2 3 2 2 2 2 3 2 2 2 2 3 3 3 2 2 2 2 2 2 2 3 3 2 2 2 2 2 2 2 2 2 2 2 2 2 3 2 3 3 3 3 2 3 3 3 3 3 3 2 2 3 3 3 3 2
## [121] 3 2 3 2 3 3 2 3 3 3 3 3 3 2 2 3 3 3 2 3 3 3 2 3 3 3 2 3 3 2Visualiser le k-means
Utilisation du package R factoextra
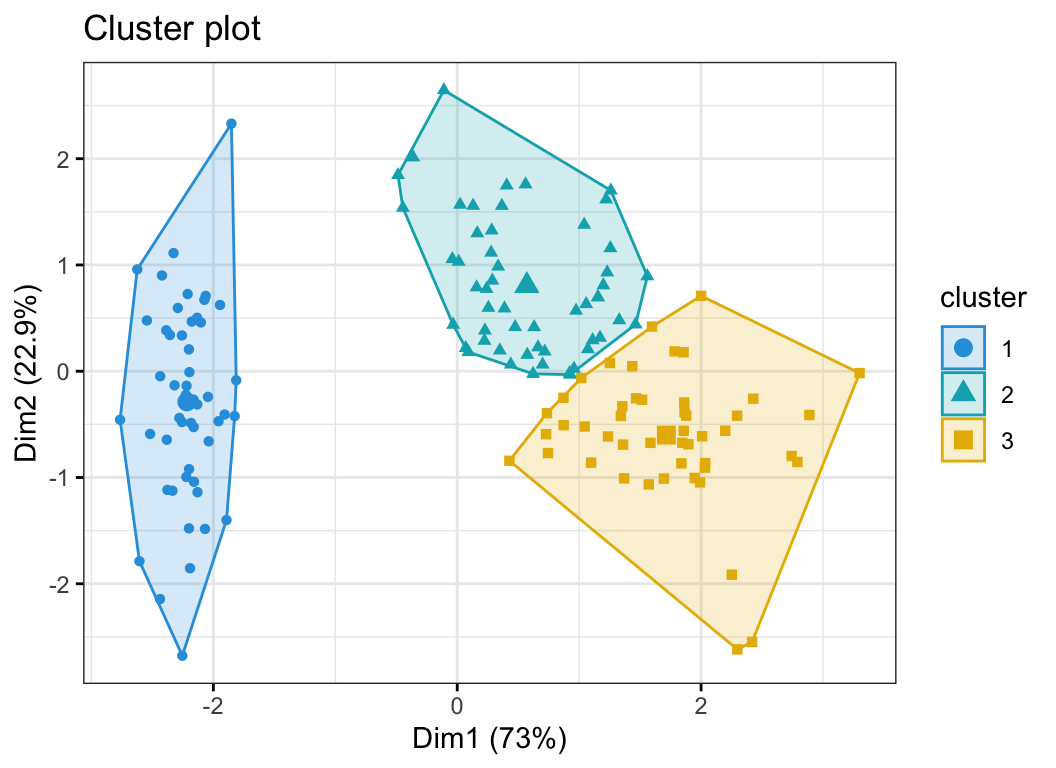
La fonction fviz_cluster() [package factoextra] peut être utilisée pour visualiser facilement les clusters k-means. Il prend comme arguments les résultats de k-means et les données originales. Dans le graphique qui en résulte, les observations sont représentées par des points, en utilisant les composantes principales si le nombre de variables est supérieur à 2. Il est également possible de dessiner une ellipse de concentration autour de chaque cluster.
fviz_cluster(res.km, data = df[, -5],
palette = c("#2E9FDF", "#00AFBB", "#E7B800"),
geom = "point",
ellipse.type = "convex",
ggtheme = theme_bw()
)
Utilisation du package R ggpubr
Si vous souhaitez adapter le graphique de clustering k-means, vous pouvez suivre les étapes suivantes:
- Calculer l’analyse en composantes principales (ACP) pour réduire les données en petites dimensions pour la visualisation
- Utilisez la fonction R
ggscatter()[dans ggpubr] ou les fonctions ggplot2 pour visualiser les clusters
Calculer la PCA et extraire les coordonnées des individus
# Réduction de dimension en utilisant l'ACP
res.pca <- prcomp(df[, -5], scale = TRUE)
# Coordonnées des individus
ind.coord <- as.data.frame(get_pca_ind(res.pca)$coord)
# Ajouter les clusters obtenus à l'aide de l'algorithme k-means
ind.coord$cluster <- factor(res.km$cluster)
# Ajouter les groupes d'espèces issues du jeu de données initial
ind.coord$Species <- df$Species
# Inspection des données
head(ind.coord)## Dim.1 Dim.2 Dim.3 Dim.4 cluster Species
## 1 -2.26 -0.478 0.1273 0.02409 1 setosa
## 2 -2.07 0.672 0.2338 0.10266 1 setosa
## 3 -2.36 0.341 -0.0441 0.02828 1 setosa
## 4 -2.29 0.595 -0.0910 -0.06574 1 setosa
## 5 -2.38 -0.645 -0.0157 -0.03580 1 setosa
## 6 -2.07 -1.484 -0.0269 0.00659 1 setosa# Pourcentage de la variance expliquée par les dimensions
eigenvalue <- round(get_eigenvalue(res.pca), 1)
variance.percent <- eigenvalue$variance.percent
head(eigenvalue)## eigenvalue variance.percent cumulative.variance.percent
## Dim.1 2.9 73.0 73.0
## Dim.2 0.9 22.9 95.8
## Dim.3 0.1 3.7 99.5
## Dim.4 0.0 0.5 100.0Visualiser les clusters k-means
- Colorier les individus selon les clusters
- Changer la forme des points en fonction des groupes d’espèces (grouping de référence)
- Ajouter des ellipses de concentration
- Ajoutez le centroid des clusters en utilisant la fonction R
stat_mean()[ggpubr]
ggscatter(
ind.coord, x = "Dim.1", y = "Dim.2",
color = "cluster", palette = "npg", ellipse = TRUE, ellipse.type = "convex",
shape = "Species", size = 1.5, legend = "right", ggtheme = theme_bw(),
xlab = paste0("Dim 1 (", variance.percent[1], "% )" ),
ylab = paste0("Dim 2 (", variance.percent[2], "% )" )
) +
stat_mean(aes(color = cluster), size = 4)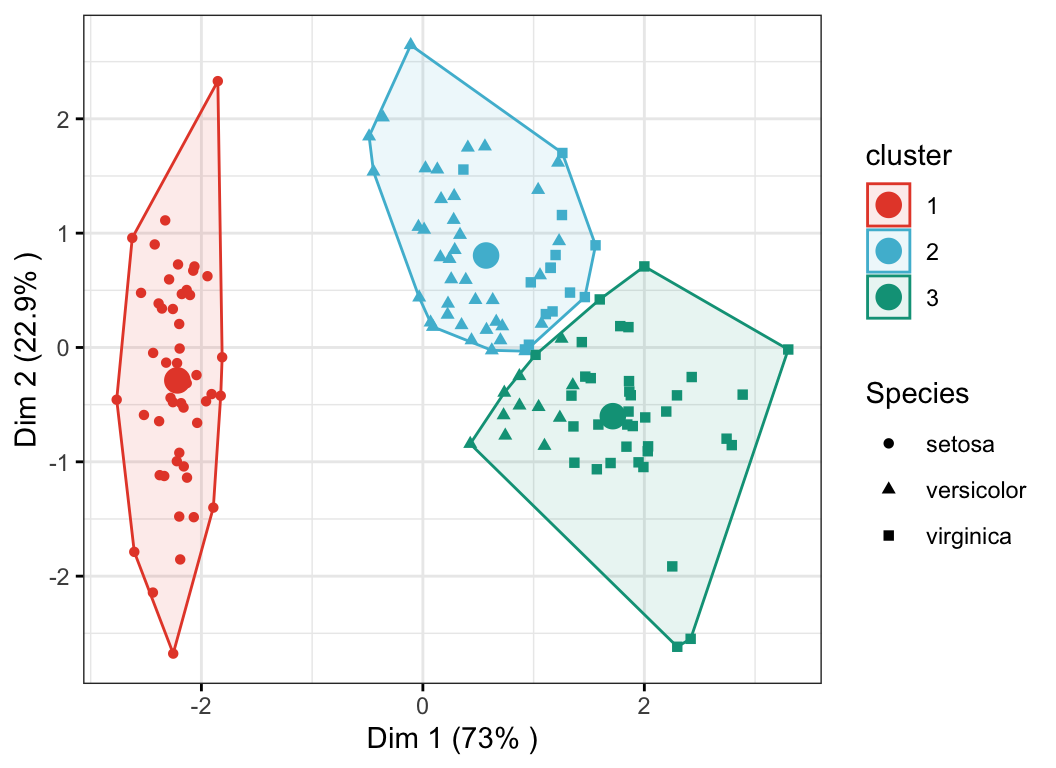
Conclusion
Cet article fournit des exemples de codes pour la visualisation des clusters K-means dans R en utilisant les packages R factoextra et ggpubr. Pour en savoir plus sur l’algorithme de clustering k-means, consultez le site K-means clustering in R : Step by Step Practical Guide.
Version:
 English
English






No Comments